背景与历史人类基因组计划(Human Genome Project):- 启动时间:1990年,目标是绘制完整的人类基因组序列。
- 完成情况:2003年完成了85%的基因组序列,2022年完成了所有缺失片段的填补。
- 技术对比:最初使用的是Sanger测序技术,一次只能测序一条DNA链。而NGS技术可以同时测序数十亿条DNA链,大大提高了速度。
- 通过NGS技术,测序一个人的全基因组只需一天,显著缩短了测序时间。

NGS的原理与步骤
样品处理文库构建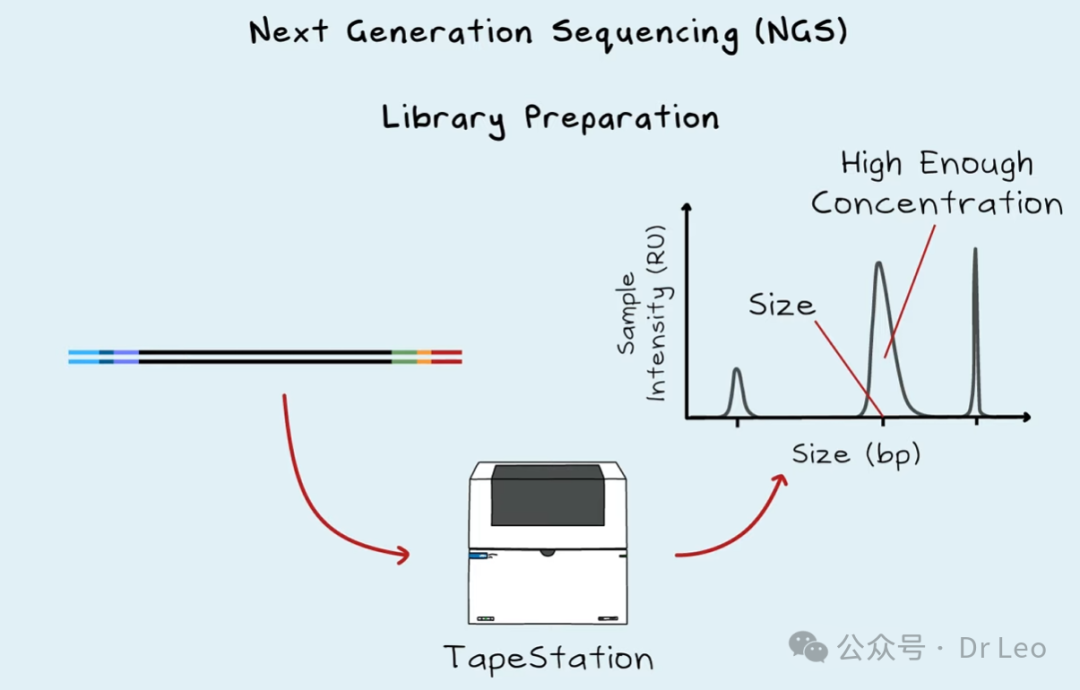
- 这些适配子包含了测序仪器所需的序列信息,还包含样品标识(Index),用于区分不同的样本。
- 通过磁珠等方法去除未结合的适配子,保证文库的质量。
- 根据具体应用,可能加入PCR扩增步骤来增加文库的量。
- 一个成功的文库需达到指定的片段长度和足够的浓度,才能进入下一步测序。
测序准备与合成- 测序发生在玻璃表面的流动池(Flow Cell)中,流动池表面附有与文库适配子序列匹配的寡核苷酸(Oligonucleotides)。
- 将文库片段通过变性(Denaturation)处理,转变为单链DNA。
- 单链DNA与流动池表面的寡核苷酸结合,形成DNA链。
- 克隆扩增(Clonal Amplification):
- 使用PCR技术对文库片段进行扩增,以便形成簇(Cluster),增加荧光信号的强度。
- 过程:DNA链通过桥式PCR扩增,形成多个拷贝(双链DNA),然后将双链分离,重复此过程形成大量局部簇。最终切割掉反向链,留下前向链用于测序。
测序过程


- 测序合成(Sequencing by Synthesis, SBS):
- 原理:测序时通过DNA聚合酶将荧光标记的核苷酸(A、T、G、C)依次加入待测DNA链中,每次加入一个碱基,荧光信号被相机捕获和记录。
- 每种碱基(A、T、G、C)带有不同颜色的荧光标签和终止子,确保每个循环只能加入一个碱基。
- 去除终止子,继续加入下一个核苷酸,重复此过程,直到完成预设的测序长度。
- 指数(Index)测序:通过标识序列区分不同的样本。
- 多重样本测序:利用独特的双重指数标签,最高可同时在一个流动池中处理384个样本。
- 双端测序(Paired-End Sequencing):
- 双端测序生成两个来自同一片段的读取(一个来自前向链,一个来自反向链),增加了测序的准确性,尤其是长片段分析。
- 过程:前向链测序完成后,创建桥接结构使反向链成为模板,接着对反向链进行测序。

数据处理- 在测序完成后,过滤掉质量较差的读取,如重叠的簇、强度过低的簇,以及多重簇的情况。
- 将过滤后的读取比对到参考基因组上,重组DNA片段,并通过算法识别和定位这些片段。
- 读数深度(Read Depth):指某个核苷酸被测序的次数。常规全基因组测序需要30倍的平均读数深度,癌症研究中检测稀有突变时需要更高的读数深度(如1500倍)。
- 覆盖度(Coverage):目标是确保测序过程中目标区域没有遗漏,覆盖率越高,数据越完整。
参考文献 Goodwin, S., McPherson, J.D., & McCombie, W.R. (2016). Coming of age: ten years of next-generation sequencing technologies. Nature Reviews Genetics, 17(6), 333-351. [DOI: 10.1038/nrg.2016.49]van Dijk, E.L., Jaszczyszyn, Y., Naquin, D., & Thermes, C. (2018). The third revolution in sequencing technology. Trends in Genetics, 34(9), 666-681. [DOI: 10.1016/j.tig.2018.05.008]Mardis, E.R. (2017). DNA sequencing technologies: 2006–2016. Nature Protocols, 12(2), 213-218. [DOI: 10.1038/nprot.2016.182]Shendure, J., & Ji, H. (2008). Next-generation DNA sequencing. Nature Biotechnology, 26(10), 1135-1145. [DOI: 10.1038/nbt1486]Head, S.R., Komori, H.K., LaMere, S.A., Whisenant, T., Van Nieuwerburgh, F., Salomon, D.R., & Ordoukhanian, P. (2014). Library construction for next-generation sequencing: Overviews and challenges. BioTechniques, 56(2), 61-77. [DOI: 10.2144/000114133]Metzker, M.L. (2010). Sequencing technologies - the next generation. Nature Reviews Genetics, 11(1), 31-46. [DOI: 10.1038/nrg2626]Levy, S.E., & Myers, R.M. (2016). Advancements in next-generation sequencing. Annual Review of Genomics and Human Genetics, 17(1), 95-115. [DOI: 10.1146/annurev-genom-083115-022413]Reuter, J.A., Spacek, D.V., & Snyder, M.P. (2015). High-throughput sequencing technologies. Molecular Cell, 58(4), 586-597. [DOI: 10.1016/j.molcel.2015.05.004]Behjati, S., & Tarpey, P.S. (2013). What is next generation sequencing?. Archives of Disease in Childhood Education and Practice Edition, 98(6), 236-238. [DOI: 10.1136/archdischild-2013-304340]Goodwin, S., McPherson, J.D., & McCombie, W.R. (2016). Coming of age: ten years of next-generation sequencing technologies. Nature Reviews Genetics, 17(6), 333-351. [DOI: 10.1038/nrg.2016.49]
|
 /3
/3 