Nat Biotechnol发表基于纳米孔的tRNA直接测序新方法
2023-5-19 08:59|
编辑: mango524|
查看: 2579|
评论: 0|来源: 测序中国
摘要: Nano-tRNAseq方法能够使用纳米孔DRS准确且直接地以单转录物分辨率量化tRNA丰度和修饰谱,是一种简单、稳健、有效的tRNA测序方法。
RNA是存在于生物细胞以及部分病毒、类病毒中的遗传信息载体,不同类型RNA具有不同的作用。转移RNA(tRNA)在蛋白质翻译中起着重要作用。如果tRNA中发生错误修饰或修饰缺失,会产生错误或不完整的蛋白质。已有研究发现,多种tRNA修饰酶的突变与各种人类疾病有关,包括神经退行性疾病、代谢疾病和癌症。但是,研究tRNA一直面临着诸多挑战,部分原因是缺乏一种同时量化其丰度和化学修饰的简单方法。因为目前的纳米孔测序设置会丢弃绝大多数tRNA reads,测序产量低,并且基于转录物长度对tRNA丰度的表示有偏差。 近日,来自西班牙巴塞罗那科学技术研究所的研究团队及合作者在Nature Biotechnology发表了题为“Quantitative analysis of tRNA abundance and modifications by nanopore RNA sequencing”的研究论文。研究团队开发了一种名为Nano-tRNAseq的tRNA纳米孔测序新方法,可直接对天然tRNA进行精确测序,准确定量tRNA丰度并同时捕获tRNA修饰变化。研究团队利用Nano-tRNAseq成功检测了酿酒酵母tRNA群同一分子内不同tRNA修饰类型之间的串扰和相互依赖性,以及tRNA群体对氧化应激的反应变化。
文章发表在Nature Biotechnology牛津纳米孔技术公司(ONT)开发的直接RNA测序(DRS)平台是基于NGS技术来描述tRNA组的一个很有潜力的替代方案。该技术允许对天然RNA分子进行直接测序,因此,原则上可以检测和定量tRNA修饰和tRNA丰度,不需要逆转录或PCR。先前的研究已经证明,纳米孔可以使用固态或生物(ONT)纳米孔捕获tRNA。通过连接接头来延长tRNA分子,tRNA可以通过生物纳米孔进行测序、基础标记和定位。但上述方法tRNA分子的测序产量较低,并且没有报告是否使用该方法再现了现存的体内tRNA丰度和/或tRNA修饰。研究团队发现,对原始纳米孔信号强度再处理,用RNA接头填充5′和3′tRNA末端,可以准确地进行碱基判定和映射,并捕获整个tRNA序列,使tRNA reads的数量增加12倍,并获得准确的tRNA丰度。这种基于纳米孔的方法被命名为Nano-tRNAseq(图1),可用于对天然tRNA进行测序,并在单个实验中获取到tRNA丰度和修饰动力学的定量估计。研究团队表示,在体外和天然tRNA分子测序、碱基判定和映射方面,Nano-tRNAseq方法是使用纳米孔进行DRS最成功的方法。图1.Nano-tRNAseq可以有效地对天然tRNA进行测序。天然tRNA具有短而高度修饰的特性,这使它们的比对具有挑战性。对DRS数据集中修改碱基的不准确判定也使原tRNA检测含有很大比例的不匹配碱基。由于这些错误的碱基,使用推荐设置的常用长读长映射工具minimup2(-ax-map ont-k15)仅可以比对一小部分reads(2.56%)。为了提高Nano-tRNAseq reads可映射性,研究团队测试了短reads映射算法BWA,发现使用推荐参数的BWA-MEM比对在映射readas的比例方面优于minimup2。当使用参数为-W13 -k6 -xont2d -T20的BWA-MEM时,发现增加的映射reads和错误比对之间存在最佳平衡,映射了54.63%的reads,错误比对很少(0.19%)。当在天然tRNA分子中比较两个映射算法的性能时,对比更加明显。随后,研究人员评估了Nano-tRNAseq reads的可映射性是否受5'和3' RNA接头长度的影响。研究发现,即使参考序列中没有RNA接头,短的和未修饰的序列也可以有效地比对,而短的和修饰的reads则从带有接头的延伸序列中受益匪浅,表明带有RNA接头的延伸序列分子对于指导富含“错配”短reads的正确比对至关重要,例如来自天然tRNA的短reads。(图2)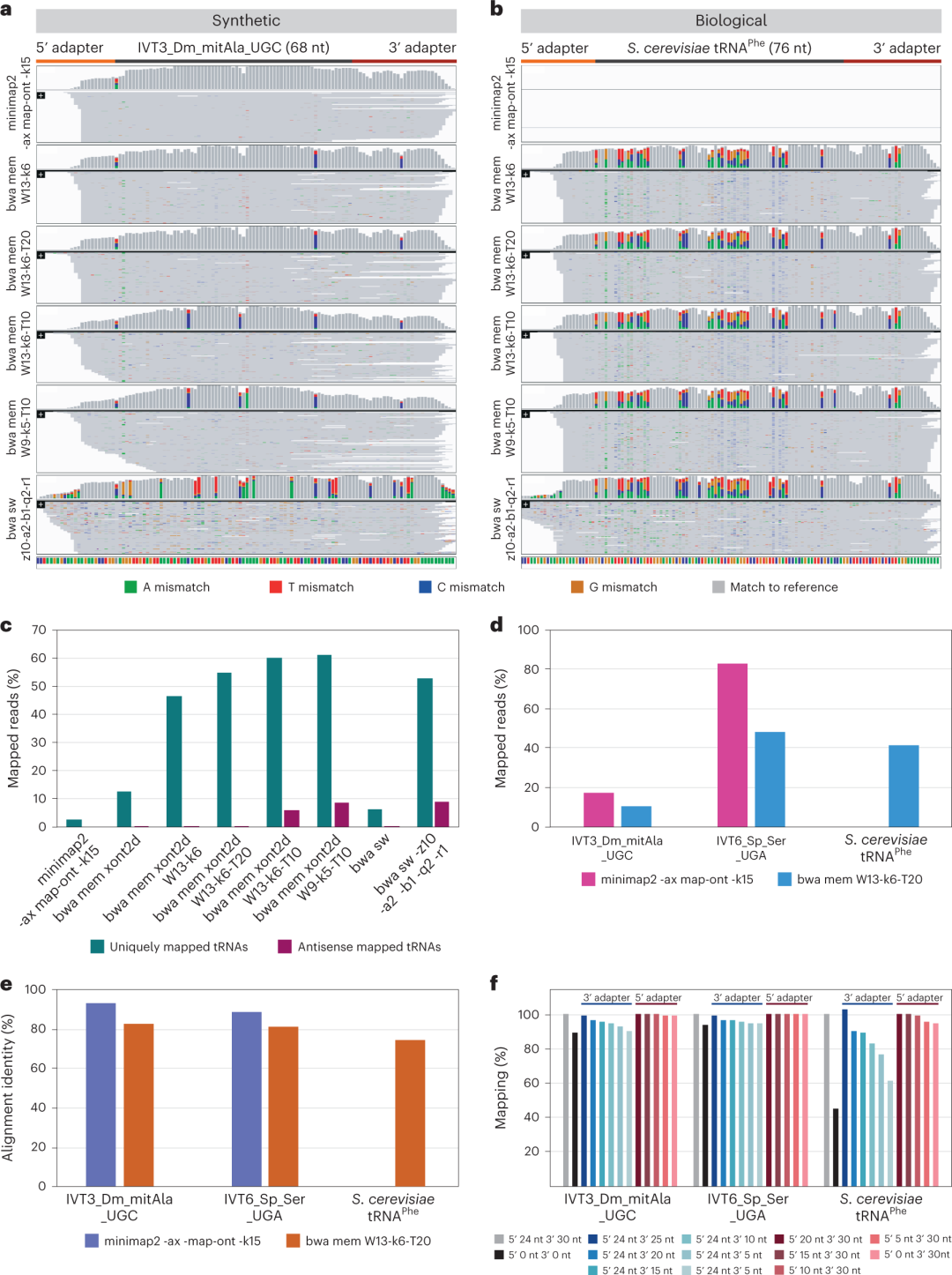
图2。映射软件和参数的选择显著影响映射tRNA reads数量。MinKNOW软件用于分析每个孔处检测的连续电流信号,将对应于reads的信号转化为FASTQ文件(图3a)。研究人员通过调整其配置,与默认MinKNOW配置相比,捕获了约12倍的碱基调用(basecalled)和约4.5倍的唯一映射tRNA reads(图3b、c)。此外,使用自定义配置可以更好地概括tRNA reads的相对比例(图3f),报告的tRNA丰度与预期值相关良好(图3g)。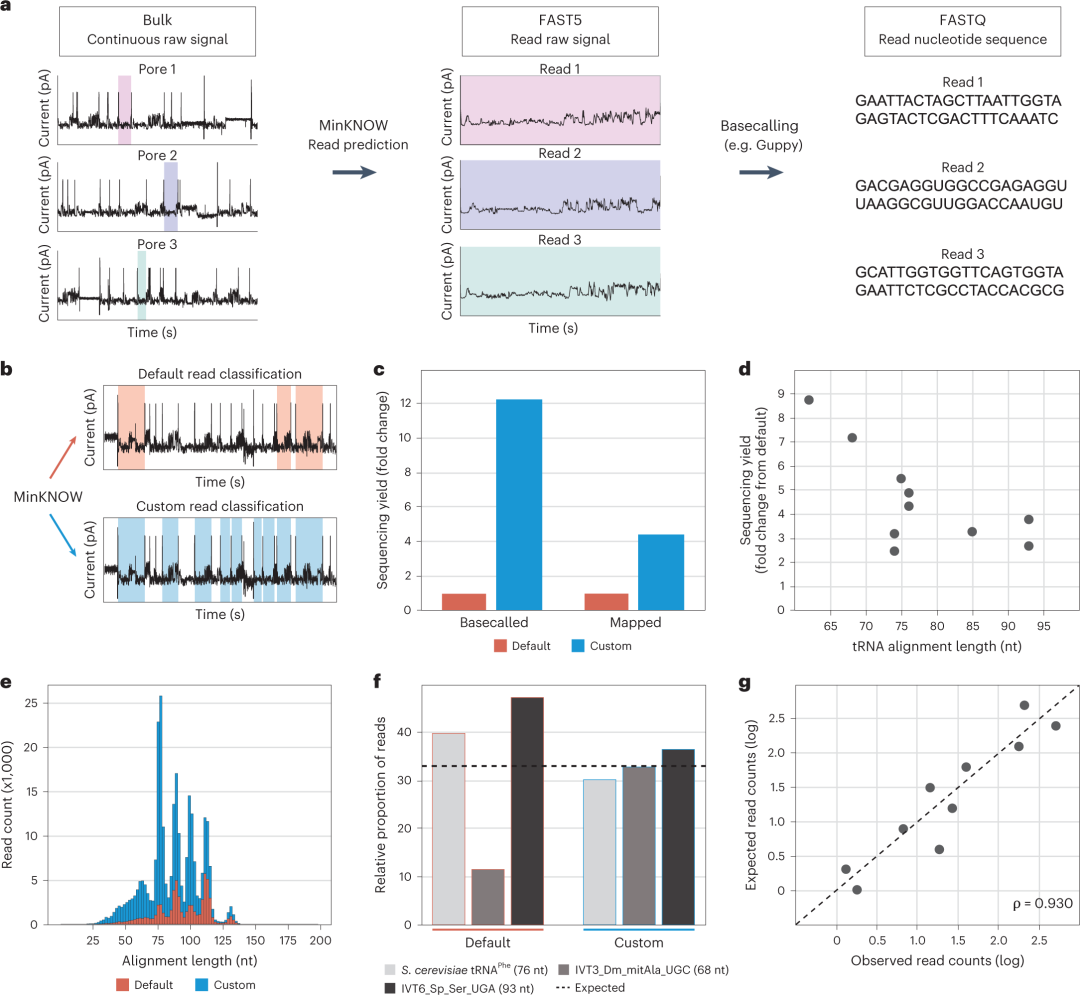
图3.MinKNOW参数的调整增加了测序和映射tRNA reads的数量。DRS的情况下,天然RNA分子可以被测序,而cDNA链则不可以。接下来,研究人员分析了tRNAs的线性化是否会进一步提高测序产量。使用Nano-tRNAseq对酿酒酵母的总tRNA进行测序,分为有和没有逆转录步骤,发现在MinKNOW默认配置中,无逆转录步骤的reads数量更多。原因在于非线性化的tRNA比线性化的tRNA更结构化,导至解旋酶处理这些分子的速度更慢,可能增加了其被MinKNOW分类为reads的可能性。总之,tRNA分子的线性化提高了测序产量,因此逆转录步骤包括在所有随后的Nano-tRNAseq文库制备中。(图4)通过进一步试验验证,研究人员证实Nano-tRNAseq可以量化tRNA丰度、tRNA修饰差异,识别tRNA修饰的相互依赖性。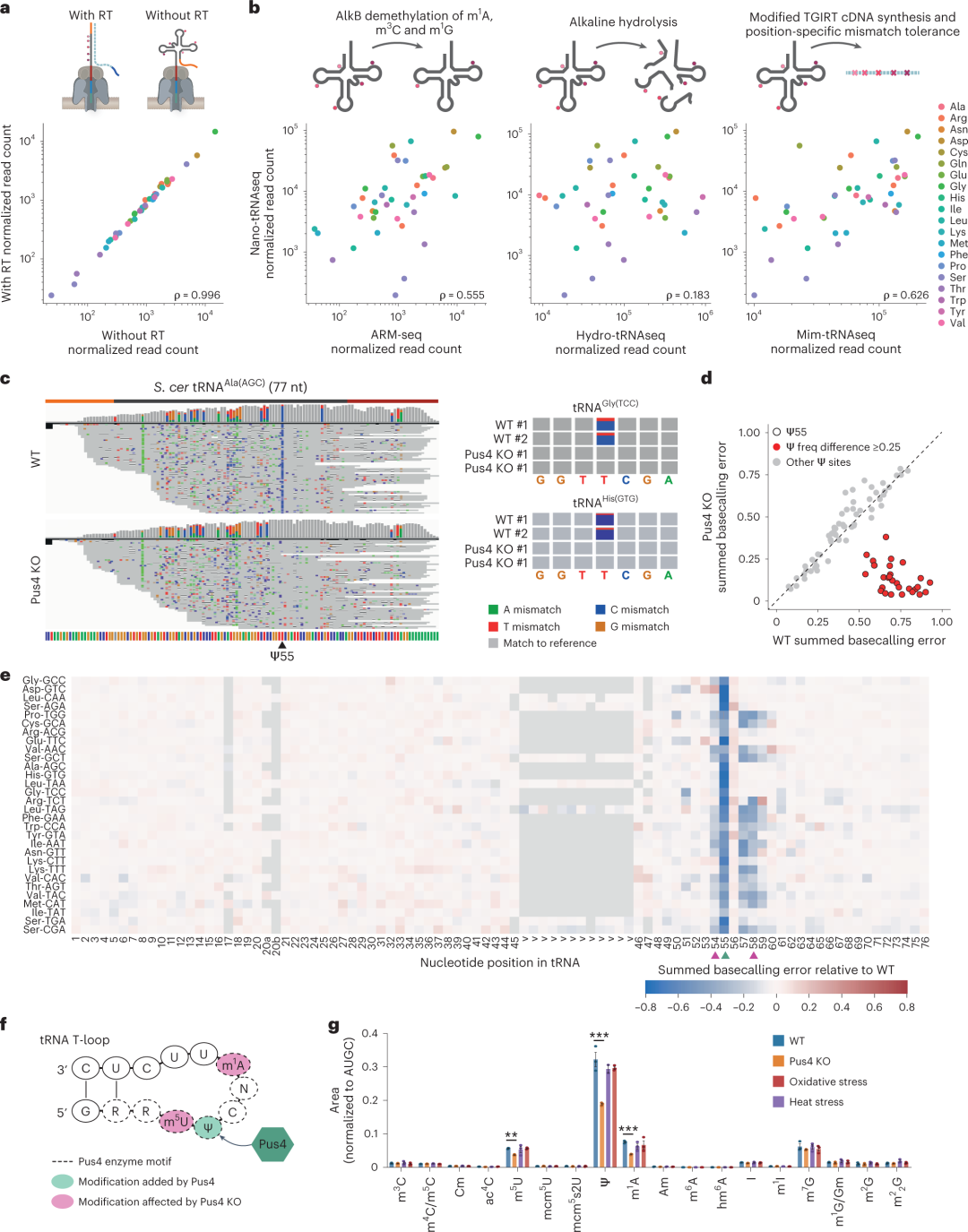
图4:Nano-tRNAseq可以量化tRNA丰度和RNA修饰,以及捕获修饰的相互依赖性。综上所述,Nano-tRNAseq方法能够使用纳米孔DRS准确且直接地以单转录物分辨率量化tRNA丰度和修饰谱,是一种简单、稳健、有效的tRNA测序方法。在文库制备方案中,成熟tRNA的5’和3’末端用RNA接头延伸,提高了tRNA分子的碱基调用和可映射性。研究人员通过自定义的MinKNOW配置更有效地捕获tRNA reads,且与tRNA长度无关,消除了长度依赖性偏差。该方法将有助于揭示tRNA生物学的动力学,并可能在不久的将来用于人类疾病的诊断和预测。该文章通讯作者、基因组调控中心研究员Eva Maria Novoa博士表示:“tRNA分子可以被切割成小而稳定的RNA片段并在血浆中循环。这些分子在癌症患者中通常会发生改变,并且携带丰富的诊断和预后信息。Nano-tRNAseq是一种概念验证技术,可以以非侵入性方式量化这些分子,为开发一种简单、经济高效、高精密的方法铺平了道路。我们的目标是进一步开发这项技术,并将其与人工智能工具相结合,在不到3小时的时间内确定生物样本的恶性程度,且每个样本的成本不超过50欧元。”Lucas MC, Pryszcz LP, Medina R, et al. Quantitative analysis of tRNA abundance and modifications by nanopore RNA sequencing [published online ahead of print, 2023 Apr 6]. Nat Biotechnol. 2023;10.1038/s41587-023-01743-6. doi:10.1038/s41587-023-01743-6https://www.nature.com/articles/s41587-023-01743-6
|
声明:
1、凡本网注明“来源:小桔灯网”的所有作品,均为本网合法拥有版权或有权使用的作品,转载需联系授权。
2、凡本网注明“来源:XXX(非小桔灯网)”的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。其版权归原作者所有,如有侵权请联系删除。
3、所有再转载者需自行获得原作者授权并注明来源。
 /3
/3 