金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
文章预览
欢迎大家关注我的微信公众号生信指北。
文章脉络
文章脉络
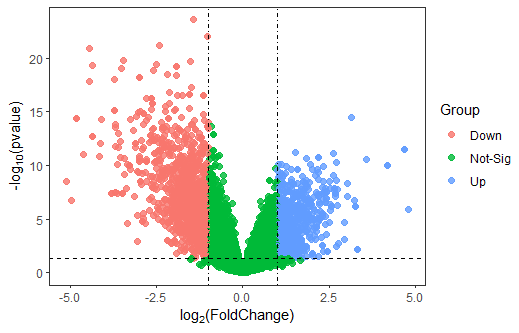
包含图形
包含图形
理解火山图
- 火山图是一种散点图,它展示了统计学显著性(P值)与变化倍数之间的关系,因为形似火山而被称为火山图,生信中用来可视化两组样本间基因表达水平差异的分布状况。
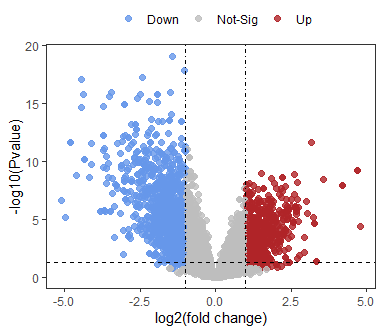
- 火山图的横轴为log2(Fold Change),一般简称为logFC,体现的是基因在样本间的差异表达倍数。从单个基因来理解,Fold Change就是两组间的表达量差异,通常是case(实验)组的平均表达量除以control(对照)组的平均表达量,比如这个基因在case组的平均表达量是8,而在control组的平均表达量是2,那么Fold Change就是4,logFC为2。而如果输入的表达矩阵不是原始的表达值而是log2后的表达矩阵,那么logFC就变成了case组的平均表达值减去control组的平均表达值,二者由公式log(x/y)=log(x)-log(y)统一。(参考生信菜鸟团(关于limma包差异分析结果的logFC解释)),这样差异越大的基因越分布在x轴的两端,左边为下调,右边为上调。
- 纵坐标通常为log10转化后的显著性p值然后取负,
-log10(pvalue=0.05)≈1.3,
-log10(pvalue=0.02)=2,
那么纵轴越往上p值越小,统计差异越显著。
了解了上述基本信息之后,我们就可以开始作图了。
ggplot2
ggplot2是R中最常用的作图库,对于ggplot2的作图我们将逐步详细进行介绍,其他方法提供代码参考。 首先我们需要加载之前差异分析的结果并加载ggplot2包:
rm(list=ls())
load('deg.RData')
library(ggplot2)
取出logFC,p值,校正后p值,差异分组四列,然后统一命名,这里是:
deg<-deg[,c(1,4,5,7)]
colnames(deg)<-c(&#39;log2FoldChange&#39;,&#39;pvalue&#39;,&#39;p.adjust&#39;,&#39;Group&#39;)
colnames(deg)
注意这里差异的分组Group是需要按自己设定的差异基因筛选标准来确定的,具体请看上篇。图中的虚线会展示筛选标准,所以这里我们也需要进行设置,我们之前采用的阈值是|logFC|>1,pvalue<0.05:
logfc.cut=1
p.cut=0.05
通过将log2FoldChange映射到x,-log10(pvalue)映射到y,差异分组映射到点的颜色,然后用geom_vline和geom_hline分别加上竖线和横线展示筛选阈值即可绘制完成简单的图形。
p<-ggplot(data = deg, aes(x = log2FoldChange,y = -log10(pvalue)))+
geom_point(alpha=0.8, size=2, aes(color=Group)) +
xlab(expression(&#39;log&#39;[2]*&#39;(FoldChange)&#39;))+ #修改坐标轴名称
ylab(expression(&#39;-log&#39;[10]*&#39;(pvalue)&#39;))+
geom_vline(xintercept=c(-logfc.cut,logfc.cut),lty=4,col=&#34;black&#34;,lwd=0.5) +
geom_hline(yintercept = -log10(p.cut),lty=2,col=&#34;black&#34;,lwd=0.5) +
theme_test() #这个主题可以去掉背景网格线
p
出图如下:
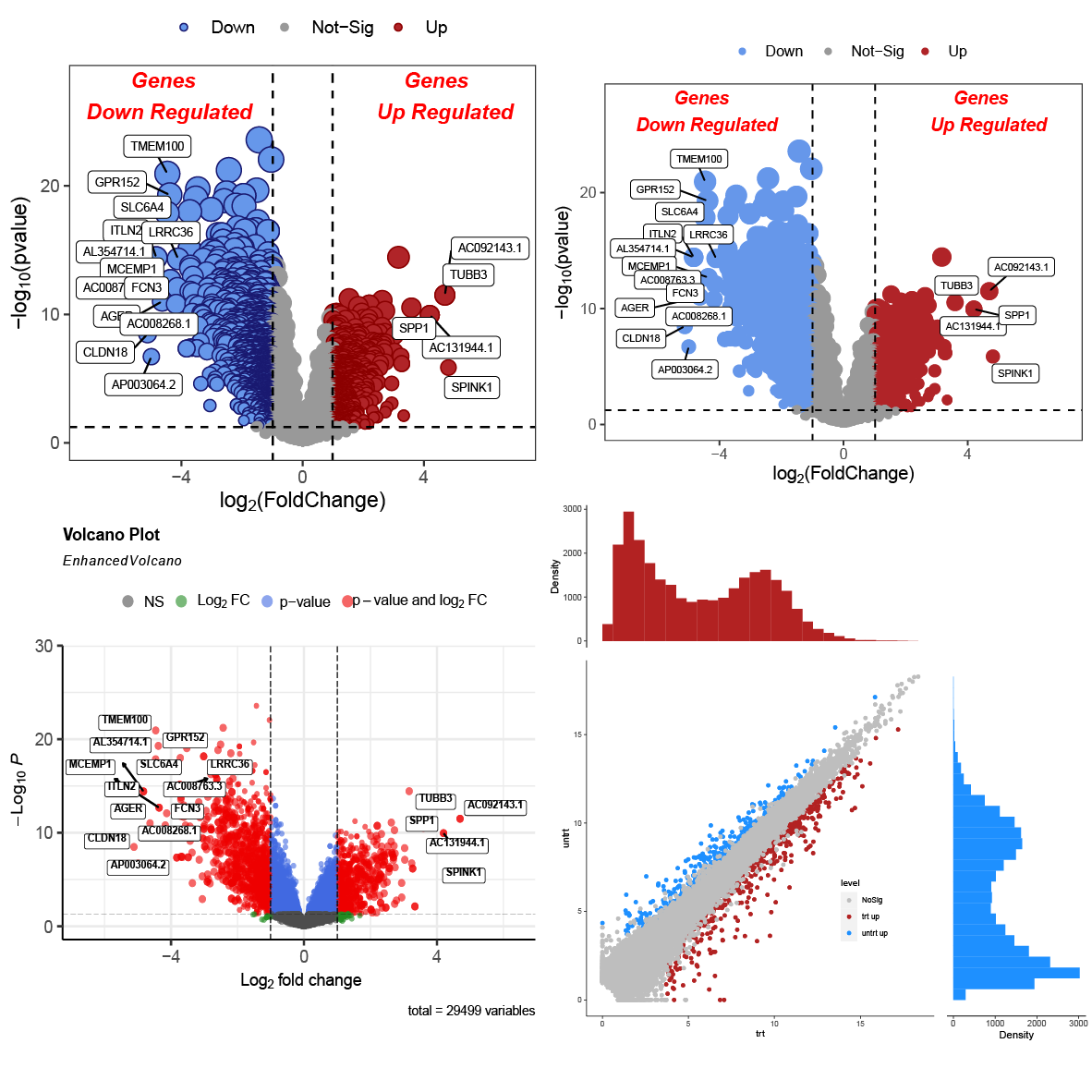
未指定颜色的时候ggplot2会使用其默认配色方案,我们可以通过scale_color_manual()手动指定配色,另外可以在主题中调整图例的位置,这些都可以通过添加图层实现,这样就完成了一副火山图的绘制。
p+scale_color_manual(values=c(&#34;#6697ea&#34;, &#34;grey&#34;,&#34;#b02428&#34;))+
theme(legend.position = &#34;top&#34;,legend.title = element_blank())
出图如下:
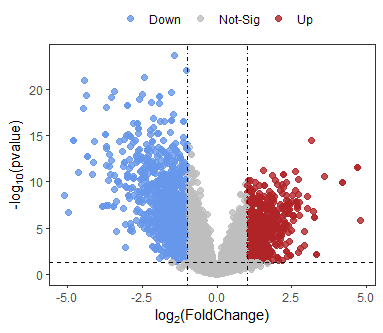
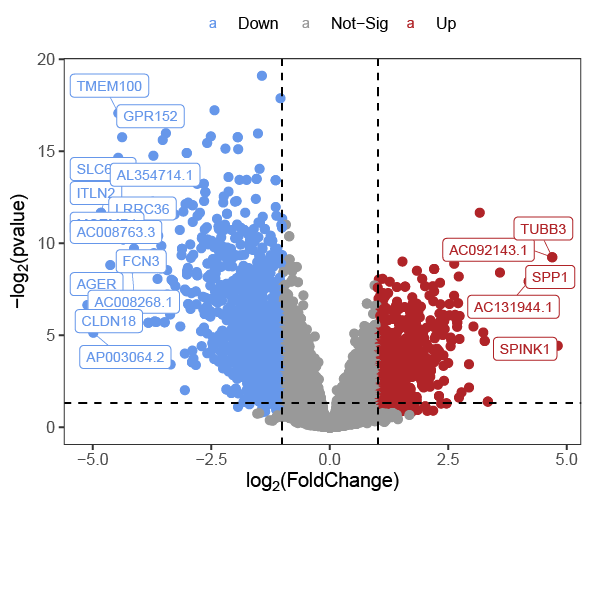
熟悉一些ggplot2的基本用法之后,我们就可以按照自己的想法来美化图片了,我们这里将校正后的p值映射到了点的大小,使用annotate添加上了文本注释,再加了一点点细节,就可以得到下面的图啦,需要这张图的带注释的代码和示例数据的同学可以在公众号回复ggplot2火山图代码拿到哦
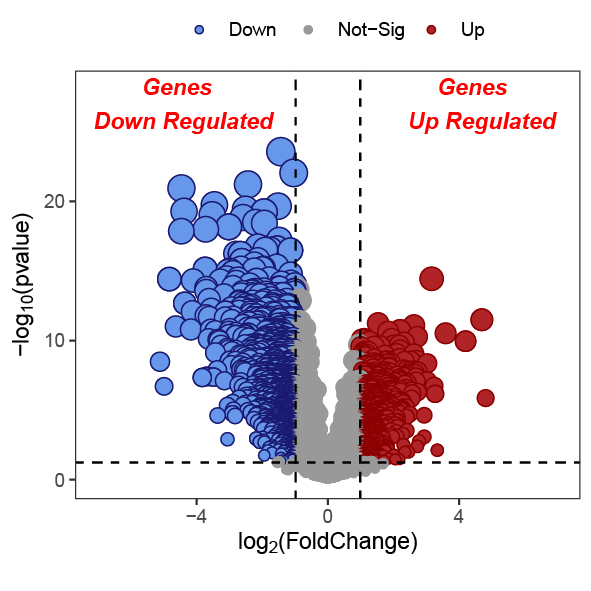
另外肯定有许多小伙伴想要在图中标注自己感兴趣的基因的方法,这里我们使用ggrepel实现,首先加载ggrepel包,然后设置允许重叠次数为无穷,否则图中重叠的点是无法进行标注的。
library(ggrepel)
options(ggrepel.max.overlaps = Inf)
然后就需要找到我们自己感兴趣的基因,我们这里把|logFC|>4的基因设置为感兴趣基因,如果同学们有自己感兴趣的genelist,只需要将其赋值给interested_genes即可,然后新建一列与目标基因进行匹配,这一列输入到geom_label_repel中即可完成标注
interested_genes<-rownames(subset(deg,abs(log2FoldChange)>4))
#判断基因是否在感兴趣的基因内,在则标注为该基因名,否则为空
deg$Label<-ifelse(rownames(deg)%in%interested_genes,rownames(deg),&#39;&#39;)
结果如下:
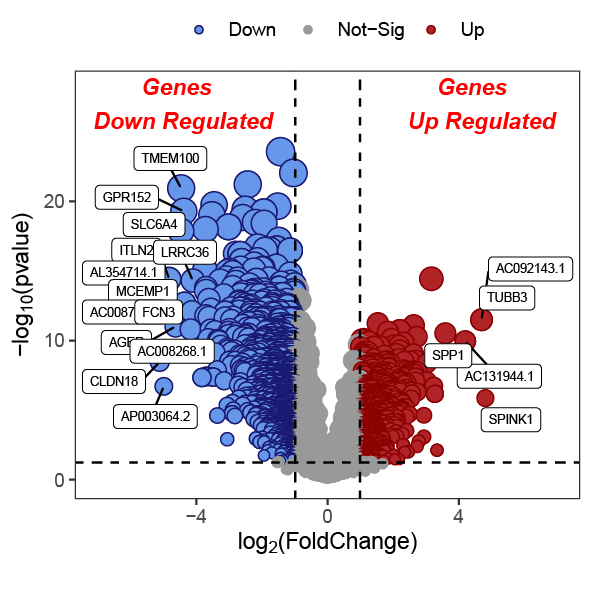
图中点有很多重叠所以边框颜色挤在一起了,这里设置的是同色系的边框,如果是黑色更加不好看,主要是因为数据点太多的原因,在数据点较少的情况下图还是比较好看的。这种情况下可以通过scale_color_manual()将边框颜色设置为与填充颜色一致,这样就去掉了边框颜色:
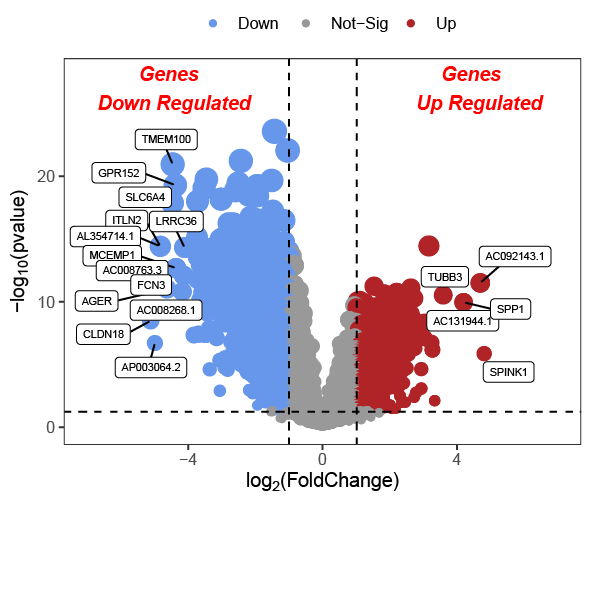
代码和数据已经包含在回复ggplot2火山图代码的内容当中,想要的童鞋可以回复领取。
ggpubr
利用ggpubr包可以使用其中的ggscatter函数实现火山图的制作。
rm(list=ls())
load(&#39;deg.RData&#39;)
deg<-deg[,c(1,4,5,7)]
colnames(deg)[1:3]<-c(&#39;log2FoldChange&#39;,&#39;pvalue&#39;,&#39;p.adjust&#39;)
colnames(deg)
logfc.cut=1
p.cut=0.05
interested_genes<-rownames(subset(deg,abs(log2FoldChange)>4))
deg$Label<-ifelse(rownames(deg)%in%interested_genes,rownames(deg),&#39;&#39;)
deg$logP<- -log10(deg$p.adjust)
library(ggpubr)
ggscatter(deg, x = &#34;log2FoldChange&#34;, y = &#34;logP&#34;,
color = &#34;Group&#34;,
palette = c(&#34;#6697ea&#34;, &#34;grey60&#34;,&#34;#b02428&#34;),
label = &#39;Label&#39;,
font.label = 8,
repel = T,
label.rectangle = T,
xlab = expression(&#39;log&#39;[2]*&#39;(FoldChange)&#39;),
ylab = expression(&#39;-log&#39;[2]*&#39;(pvalue)&#39;),
size = 2) +
theme_test() +
geom_hline(yintercept = -log10(p.cut), linetype = &#34;dashed&#34;) +
geom_vline(xintercept = c(-logfc.cut, logfc.cut), linetype = &#34;dashed&#34;)+
theme(legend.position = &#34;top&#34;,legend.title = element_blank())
出图如下:
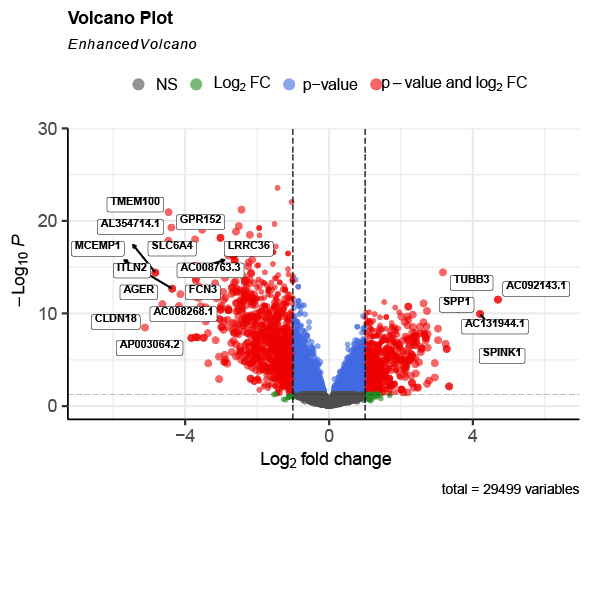
EnhancedVolcano
EnhancedVolcano包能够做出很漂亮的火山图,这里也分享给大家。只需要调用一个函数即可。
BiocManager::install(&#39;EnhancedVolcano&#39;) #安装
library(EnhancedVolcano)
EnhancedVolcano(deg,
lab = rownames(deg),
x = &#39;log2FoldChange&#39;,
y = &#39;pvalue&#39;,
title = &#39;Volcano Plot&#39;,
xlab = bquote(~Log[2]~&#39;fold change&#39;),
pCutoff = 0.05,
FCcutoff = 1,
#pointSize = 4,
pointSize = c(ifelse(abs(deg$log2FoldChange)>2,3, 2)),
selectLab =interested_genes,#指定要标注的基因
labSize = 4.0,
colAlpha = 0.6,
legendPosition = &#39;top&#39;,
legendLabSize = 16,
legendIconSize = 5.0,
labCol = &#39;black&#39;,
labFace = &#39;bold&#39;,
boxedLabels = TRUE,
drawConnectors = TRUE,
widthConnectors = 1,
colConnectors = &#39;black&#39;
)
出图如下:
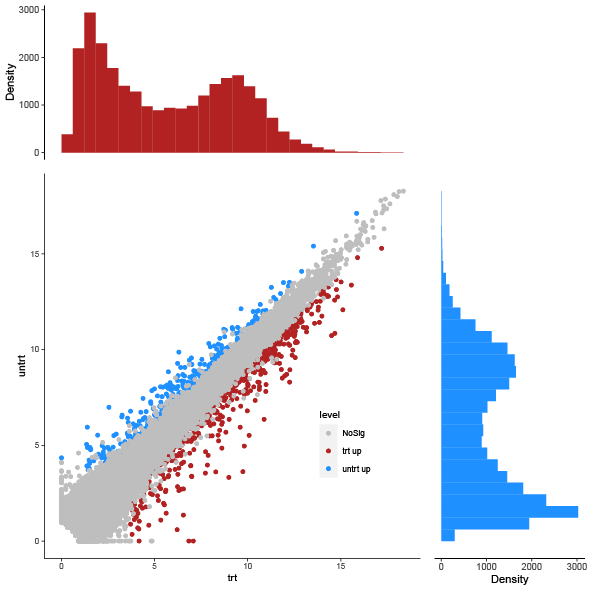
倾斜45度火山图
倾斜45度火山图可以在生信宝典的教程当中找到,示例使用的是DESeq2的差异分析结果数据,有两组的平均表达量值,而limma是没有的,所以这里不做展示,感兴趣的同学可以移步生信宝典的教程。
总结
这篇文章和大家分享了一些火山图的制作方法,主要介绍了ggplot2方法,希望大家喜欢,同时也感谢大家的支持! |
|
 /3
/3 