金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|

聚合酶链式反应(PCR)是一种彻底改变了分子生物学世界及其他领域的技术。在这篇文章中,我们将讨论PCR的简要历史及其原理,强调PCR的不同类型及其应用的具体目的。
PCR原理和历史
1983年,美国生物化学家Kary Mullis在深夜开车回家时,突然有了灵感。他在一张收据的背面写下了最终使他在1993年获得诺贝尔化学奖的想法。这个概念很简单:在实验室的试管中复制在细胞中发生的DNA复制过程。其结果是一样的:在现有的基础上产生新的互补DNA(cDNA)链。
Mullis将Sanger的DNA测序的基础作为他的新技术的起点。他意识到,重复使用DNA聚合酶会引发连锁反应,导致特定DNA片段的扩增。
1976年,他发现了一种恒温DNA聚合酶,Taq,它是从温泉中发现的水热菌中分离出来的[1]。
在发现Taq聚合酶之前,分子生物学家已经在尝试优化循环DNA扩增方案,但他们需要在每个循环中添加新的聚合酶,因为这种酶不能承受DNA变性所需的高温。拥有一种热稳定的酶意味着他们可以多次重复扩增过程,而不需要在每个循环添加新鲜的聚合酶,使整个过程可以扩展,更有效率,更省时。
1985年,Science杂志首次描述了这种使用Taq聚合酶的聚合酶链反应(PCR)[2]。
1993年,第一个经FDA批准的PCR试剂盒投放市场。从那时起,PCR得到了稳步和系统的改进。它已经成为从法医证据分析和诊断,到疾病监测和基因工程的一个游戏规则的改变者。毫无疑问,它被认为是20世纪最重要的科学进步之一。
标准PCR实验概述
PCR用于从被称为模板DNA的起始材料复杂混合物中扩增出一个特定的DNA片段。样品制备和纯化方案取决于起始材料,包括样品基质和目标DNA的可及性。通常情况下,需要将DNA提纯到它的最小扩增浓度。然而,PCR确实需要了解待扩增DNA片段(称为目标DNA)侧面的DNA序列信息。
从实用的角度来看,PCR实验相对简单,可以在几个小时内完成。一般来说,一个PCR反应需要五个关键试剂:
待扩增DNA:也叫PCR模板或模板DNA。这种DNA可以是任何来源的,如基因组DNA(gDNA)、cDNA和质粒DNA。
DNA聚合酶:所有PCR反应都需要一种能在高温下工作的DNA聚合酶。Taq聚合酶是常用的一种,它能在70℃下以60个碱基/秒的速度结合核苷酸,并能扩增长达5kb的模板,因此它适用于标准的PCR,没有特殊要求。新一代的聚合酶正在被设计来改善反应性能。例如,有些聚合酶被设计成只在高温下激活,以减少反应开始时的非特异性扩增。其他的聚合酶则具有“校对”功能,例如,在克隆过程中,扩增序列与模板序列完全一致时,这种功能是非常重要的。
引物:DNA聚合酶需要一个简短的核苷酸序列来指示它们需要启动扩增的位置。在PCR中,这些序列被称为引物,是单链DNA的短片段(大约15 ~ 30个碱基)。在设计PCR实验时,研究人员要确定要扩增的DNA区域,并设计一对引物,一个在正向链上,一个在反向链上,专门针对目标区域的侧翼。
引物设计是PCR实验的一个关键部分,应谨慎进行。引物序列的选择必须以感兴趣的独特DNA为目标,避免与类似序列结合的可能性。它们应该有相似的熔解温度,因为退火步骤对两条链来说是同时发生的。
引物的熔化温度可能受到鸟嘌呤(G)或胞嘧啶(C)与腺嘌呤(A)或胸腺嘧啶(T)相比所占百分比的影响,较高的GC含量会提高熔化温度。调整引物长度可以帮助在匹配引物对时补偿这一点。避免形成二级结构或引物二聚体的序列也很重要,因为这将降低PCR效率,有许多免费的在线工具可用于帮助设计引物。
脱氧核苷酸三磷酸酯(dNTPs):这些作为合成DNA新链的构件,包括四种基本的DNA核苷酸(dATP、dCTP、dGTP和dTTP)。dNTPs通常以等摩尔量添加到PCR反应中,以实现最佳的碱基结合。
PCR缓冲液:PCR缓冲液确保在整个PCR反应中保持最佳条件。PCR缓冲液的主要成分包括氯化镁(MgCl2)、tris-HCl和氯化钾(KCl)。氯化镁作为DNA聚合酶的辅助因子,而tris-HCl和氯化钾在反应期间保持稳定的pH值。
PCR反应是通过将上述试剂加入到一个试管中,并将试管置于PCR仪中进行反应。
PCR扩增包括三组确定的时间和温度,称为步骤:变性、退火和延伸(图1)。
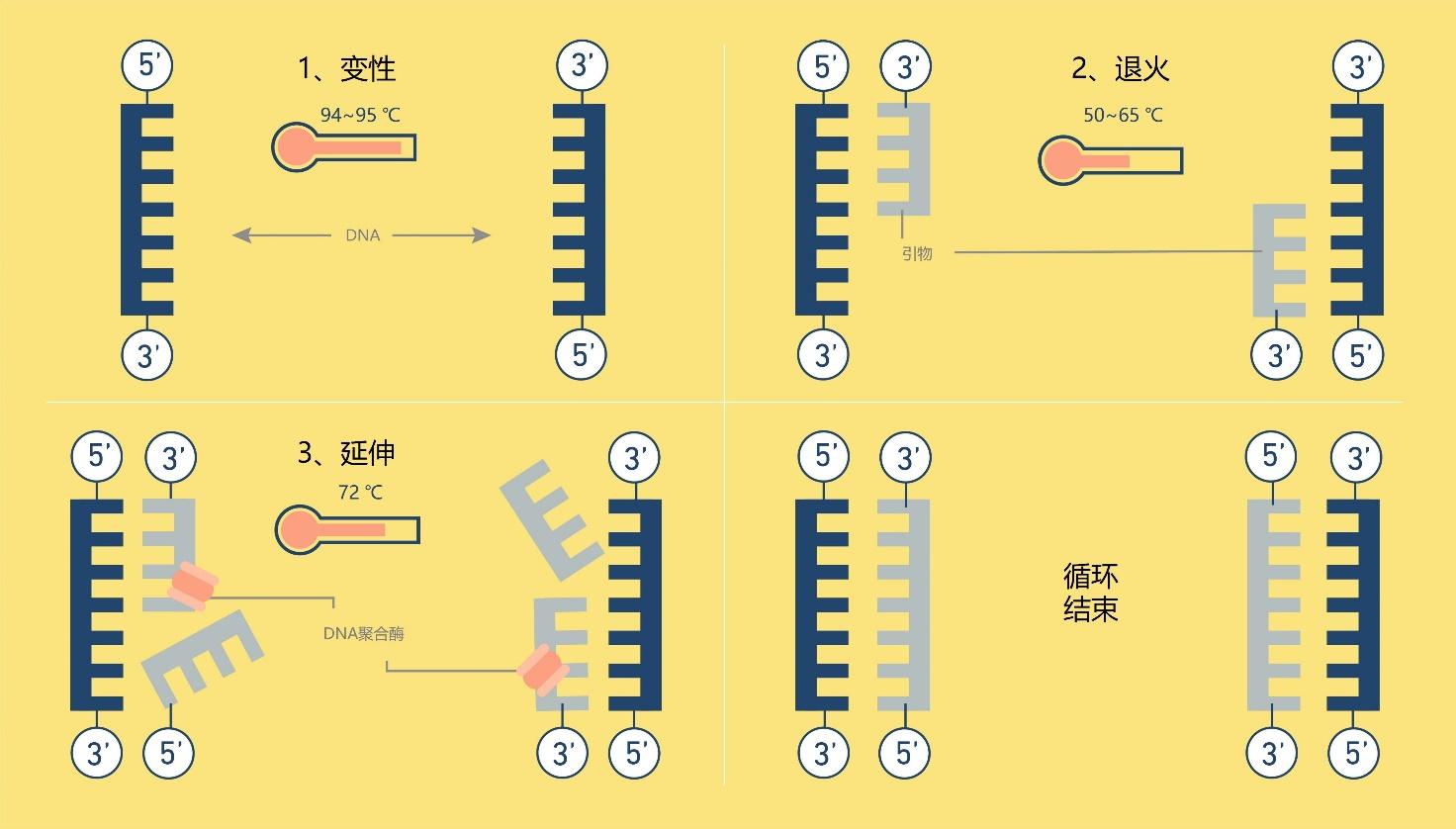
图1 | 一个PCR循环的步骤
这些步骤中的每一个都被称为循环,重复30 ~ 40次,每个循环的DNA数量翻倍,获得扩增(图2)。
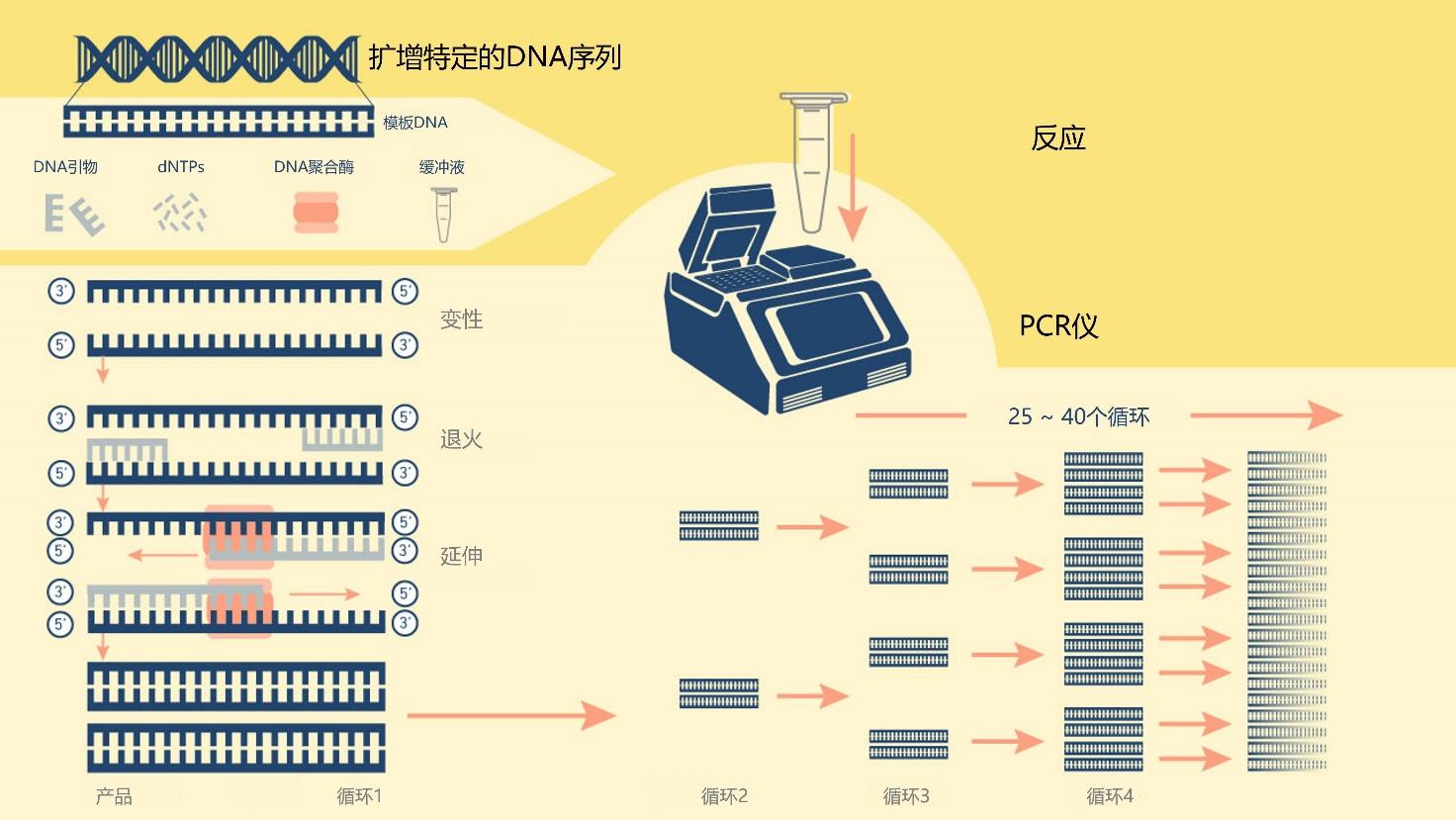
图2 | 通过PCR进行DNA分子扩增的不同阶段和循环
让我们仔细看一下每个步骤。
变性
PCR的第一步,称为变性,将模板DNA加热到95℃,持续几秒钟,随着两条DNA链之间的氢键迅速断裂而分离。
退火
然后将反应混合物冷却30秒至1分钟。退火温度通常为50 ~ 65 ℃,然而,确切的最佳温度取决于引物的长度和序列,每套新的引物都必须仔细优化。
两条DNA链可以在这个温度下重新结合,但大多数不会,因为混合物中含有大量过量的引物,它们在特定的互补位置与模板DNA结合,或退火。一旦退火步骤完成,模板DNA和引物之间将形成氢键。这个时候,聚合酶已经准备好扩展DNA序列。
延伸
然后将温度提高到混合物中存在的DNA聚合酶理想工作温度,通常在72℃左右,如果是Taq,则为74℃。
DNA聚合酶附着在每个引物的一端,合成新的DNA链,与模板DNA互补。现在我们有四条DNA链,而不是一开始就有的两条。
温度回升到94℃,双链DNA分子,其中包括“原始”分子和新合成的分子,再次变性为单链。这就开始了变性-退火-延伸的第二个循环。在这第二个循环结束时,有8个单链DNA分子。通过重复30次循环,开始时存在的双链DNA分子被转化为超过1.3亿个新的双链分子,每个分子都是由两个引物的退火点划定起始分子区域的副本。
为了确定扩增是否成功,PCR产物可以通过凝胶电泳进行可视化,显示扩增子的存在/不存在、大小和大致丰度。根据应用和研究问题,这可能是一个实验的终点,例如,如果确定一个基因是否存在。否则,PCR产物可能只是更复杂的下游调查(如测序和克隆)的起点。
不同类型的PCR
由于其多功能性,PCR技术近年来不断发展,导致了几种不同类型的PCR技术的发展。
一些最广泛使用的是:
定量实时PCR(qPCR)
最有用的发展之一是定量实时PCR或qPCR。顾名思义,qPCR是一种定量技术,可以实时监测扩增过程并在扩增过程中检测PCR产物[2]。
它可以用来确定目标DNA的起始浓度,在许多情况下可以忽略凝胶电泳的需要。这要归功于加入了非特异性的荧光插层染料,如SYBR® Green,它与双链DNA结合后会发出荧光,或DNA寡核苷酸序列特异性的荧光探针,如水解(TaqMan)探针和分子信标。
探针与扩增子内的DNA目标序列特异性结合,并利用福斯特共振能量转移(FRET)的原理,通过一端的荧光分子和另一端的淬灭剂的耦合产生荧光。对于荧光染料和探针来说,随着目标DNA拷贝数的增加,荧光水平也按比例增加,从而可以参照含有已知拷贝数的标准对扩增进行实时定量(图3)。
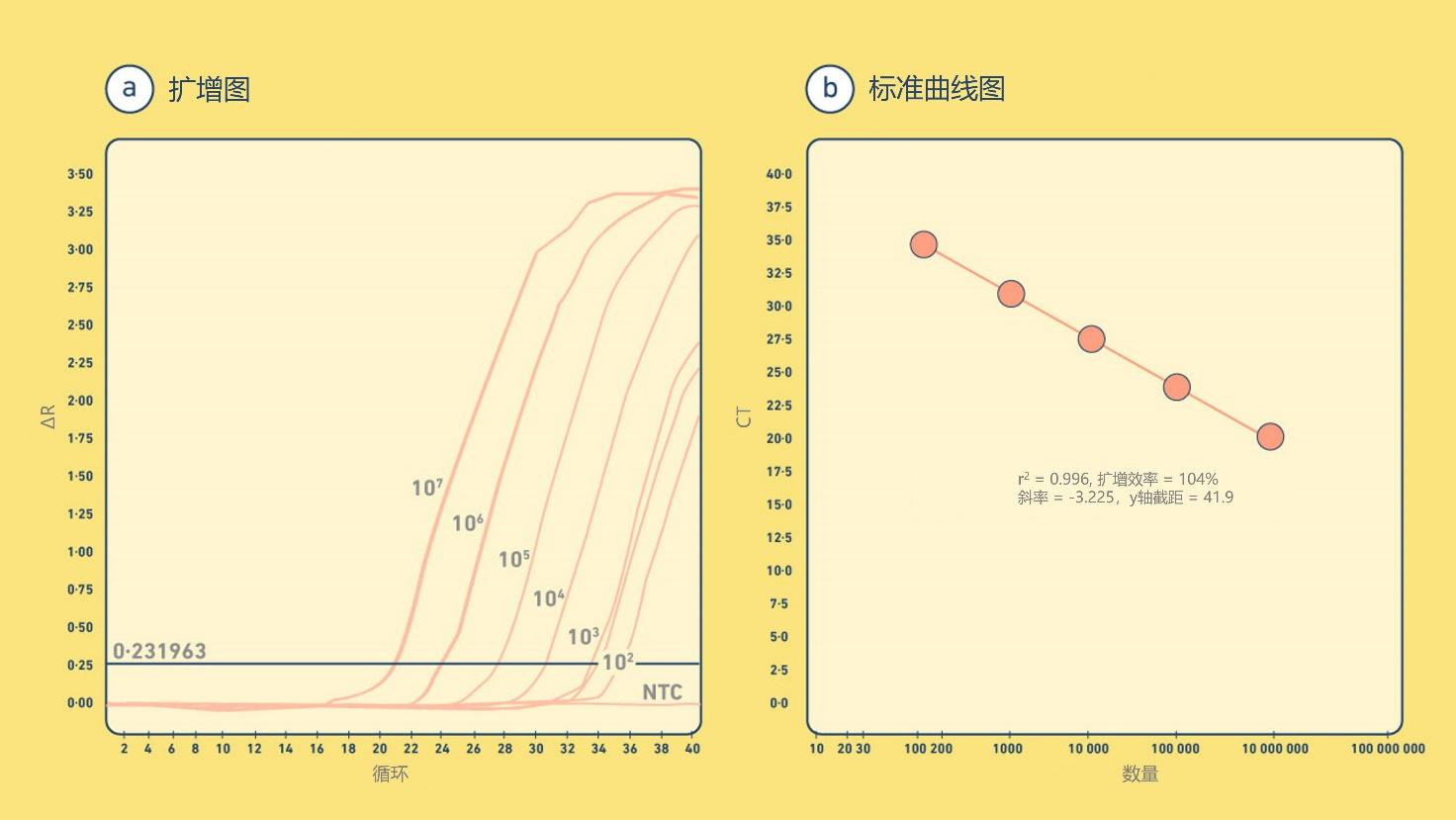
图3 | qPCR扩增图和标准曲线示例,用于对未知样本的拷贝数进行量化。
qPCR使用配备有荧光检测系统的专用PCR仪,在扩增过程中监测荧光信号。
反转录-PCR (RT-PCR)
反转录(RT)-PCR和RT-qPCR是两种常用的PCR变体,能够在临床和研究环境中进行基因转录分析和病毒RNA的定量。
RT是用单链模板RNA制作cDNA的过程[3],因此也被称为第一链cDNA合成。RT-PCR的第一步是在RNA模板和DNA寡核苷酸引物之间合成一个DNA/RNA杂交体。催化这一反应的逆转录酶具有RNase活性,然后降解杂交体的RNA部分。随后,通过逆转录酶的DNA聚合酶活性合成一个单链DNA分子。高纯度和高质量的起始RNA对于成功的RT-PCR是至关重要的。
RT-PCR可以按照两种方法进行:一步RT-PCR和两步RT-PCR。在第一种情况下,RT反应和PCR反应发生在同一试管中,而在两步RT-PCR中,这两个反应是分开的,并依次进行。
反转录-定量PCR(RT-qPCR)
上述反转录也经常作为qPCR的第一步,对生物样品中的RNA进行定量(无论是RNA转录本还是来自病毒RNA基因组)。
与RT-PCR一样,有两种通过RT-qPCR量化RNA的方法:一步RT-qPCR和两步RT-qPCR。在这两种情况下,RNA首先被逆转录成cDNA,作为qPCR扩增的模板。在两步法中,逆转录和qPCR扩增作为两个独立的实验依次进行。在一步法中,RT和qPCR在同一管中进行。
数字PCR(dPCR)和数字滴定PCR(ddPCR)
数字PCR(dPCR)是原始PCR方案的另一种修改版[4]。与qPCR一样,dPCR技术使用DNA聚合酶,利用引物组和探针从复杂的样品中扩增目标DNA。不过,主要的区别在于PCR反应的分区和最后的数据采集。
dPCR和ddPCR是基于限制性稀释的概念。PCR反应被分割成大量纳升体积的子反应(分区)。PCR扩增是在每个液滴内进行的。在PCR之后,用泊松统计学对每个液滴进行分析,以确定PCR阳性液滴在原始样品中的百分比。
一些分区可能包含一个或多个目标拷贝,而其他分区可能不包含目标序列。因此,分区分类为阳性(检测到目标)或阴性(未检测到目标),为数字输出格式提供基础。
ddPCR是最近的一项技术,在2011年开始使用[5]。ddPCR利用水油乳剂形成分隔模板DNA分子的分区。这些液滴基本上充当独立的试管,PCR反应就在其中进行。
微流控PCR
带有微通道和微室的微流控处理系统最近的发展为一系列的实际应用铺平了道路,包括在微流控芯片上通过PCR扩增DNA。
在芯片上进行的PCR得益于微流控技术在速度、灵敏度和试剂低消耗方面的优势。这些特点使微流控PCR对POCT特别有吸引力,例如,用于诊断应用。从实用的角度来看,样品流经一个微流控通道,反复通过反映PCR不同步骤的三个温度区。10 μL的样品进行20个PCR循环只需要90秒[6]。随后的分析可以很容易地在片外进行。
PCR故障排除
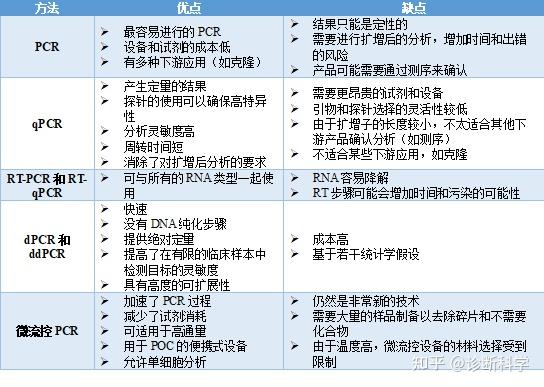
不同的PCR方法都有优点和缺点,对它们所适合的应用产生影响[7]。表1总结了这些优点。
表1 | 不同PCR方法的主要优点和缺点
PCR的应用
PCR已经成为现代分子生物学中不可缺少的工具,并完全改变了科学研究。该技术还为分子生物学领域以外的人打开了细胞和分子过程的研究,因此也被许多学科的科学家发现了用途。
虽然PCR本身是一种强大的独立技术,但它也被纳入了更广泛的技术,如克隆和测序,作为这些工作流程中一个小而重要的部分。
PCR的研究应用包括:
基因转录
PCR可以检查特定时间点上细胞类型、组织和生物体之间基因转录的变化。在这个过程中,RNA从感兴趣的样品中分离出来,并反转录成cDNA。然后,特定基因的原始RNA水平可以从PCR中扩增的cDNA数量中进行量化。
基因分型
PCR可以检测特定细胞或生物体的等位基因的序列变化。一个常见的例子是对转基因生物体进行基因分型,如基因敲除和基因敲入小鼠。在这种应用中,引物被设计用来扩增转基因部分(在转基因动物中)或突变部分(在突变动物中)。
克隆和诱变
PCR克隆是一种广泛使用的技术,通过PCR扩增的双链DNA片段被插入载体(如gDNA、cDNA、质粒DNA)。例如,这使得创建的细菌菌株的遗传物质被删除或插入。定点诱变也可用于通过克隆引入点突变。这通常采用一种被称为重组PCR的技术,其中重叠的引物被专门设计来纳入碱基替换(图4)。这种技术也可用于创造新的基因融合。
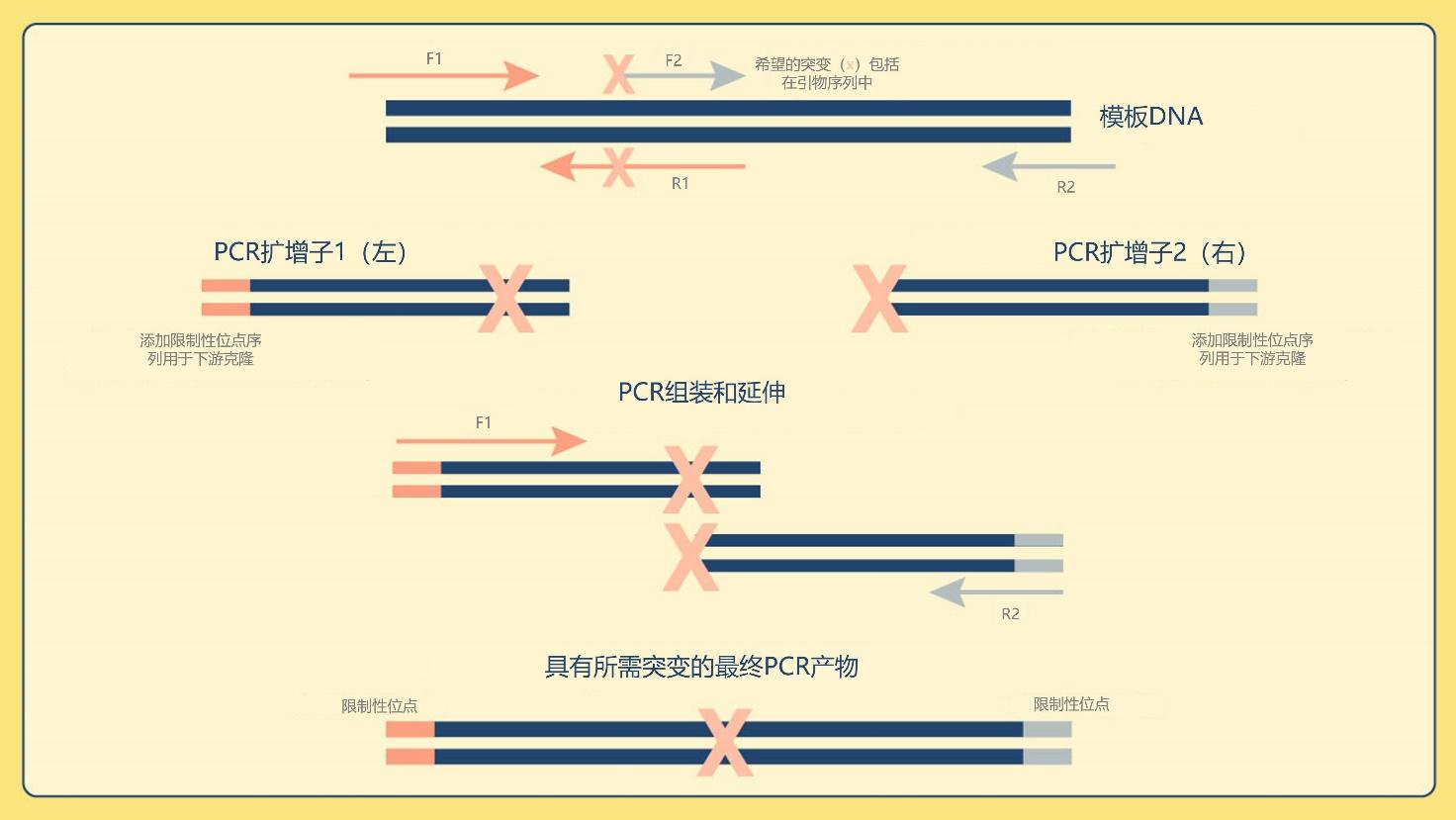
图4 | 描述重组PCR实例的图表
测序
PCR可用于富集测序用的模板DNA。推荐用于制备测序模板的PCR类型被称为高保真PCR,能够保持DNA序列的准确性。在Sanger测序中,PCR扩增的片段随后被纯化并在测序反应中运行。在第二代测序(NGS)中,PCR被用于文库制备阶段,通过PCR富集DNA样本以增加起始数量,并用测序适配体标记以实现多重检测。桥式PCR也是第二代NGS测序过程的一个重要部分。
无论是作为一种独立的技术还是作为其他方法的主力,PCR已经改变了一系列的学科。这些学科包括:
遗传研究
PCR在全球大多数实验室中使用。最常见的应用之一是基因转录分析[9],目的是评估特定基因转录物的存在或丰度。它是通过克隆操纵生物体,动物、植物和微生物,的遗传序列的一种强大技术。这使得基因或部分基因可以被插入、删除或突变,以改变基因标记的表型,阐明基因的功能和开发疫苗,这只是其中的几个例子。
在基因分型中,PCR可用于检测特定细胞或生物体中等位基因的序列变化。它的用途也不限于人类。农业中的植物基因分型有助于植物育种者选择、完善和改进其育种品种。如上所述,PCR也是丰富测序样本的第一步。例如,人类基因组计划(HGP)中的大多数绘图技术都依赖于PCR。
医学和生物学研究
PCR被用于大量的医学应用,从疾病相关的基因突变的诊断测试,到传染病原体的鉴定。PCR在医学领域应用的另一个很好的例子是产前基因测试。通过PCR进行产前基因测试可以识别胎儿的染色体异常和基因突变,为准父母提供关于他们的婴儿是否有某些遗传疾病的重要信息。PCR也可以作为植入前遗传学诊断工具,用于筛选体外受精(IVF)程序的胚胎。
法医学
我们独特的基因指纹意味着PCR可以在亲子鉴定和法医调查中发挥重要作用,以确定样本的来源。例如,从犯罪现场分离出的少量DNA样本可以与DNA数据库或嫌疑人的DNA进行比较。这些程序确实改变了警方调查的方式。真实性测试也利用了PCR遗传标记,例如,确定肉的来源物种。分子考古学也利用PCR来扩增考古遗迹的DNA。
环境微生物学和食品安全
通过PCR检测病原体,不仅在病人的样本中,而且在食品或水等基质中,对诊断和预防传染病至关重要。
PCR是每个领域检测核酸的基准技术,从生物医学研究到法医应用,Kary Mullis将他的想法写在路边的一张收据背面,结果带来了一个革命性的技术。
诊断科学编辑团队收集、整理和编撰,如需更多资讯,请关注公众号诊断科学(DiagnosticsScience)。
参考文献
- Chien A, Edgar DB, Trela JM. Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus. J Bacteriol 1976;127(3):1550-57 doi: 10.1128/JB.127.3.1550-1557.1976
- Saiki RK, Scharf S, Faloona F, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science 1985;230(4732):1350 doi: 10.1126/science.2999980
- Arya M, Shergill IS, Williamson M, Gommersall L, Arya N, Patel HRH. Basic principles of real-time quantitative PCR. Expert Review of Molecular Diagnostics 2005;5(2):209-19 doi: 10.1586/14737159.5.2.209
- Bachman J. Chapter Two - Reverse-Transcription PCR (RT-PCR). In: Lorsch J, ed. Methods in Enzymology: Academic Press, 2013:67-74. doi : 10.1016/B978-0-12-420037-1.00002-6
- Morley AA. Digital PCR: A brief history. Biomol Detect Quantif 2014;1(1):1-2 doi: 10.1016/j.bdq.2014.06.001
- Taylor SC, Laperriere G, Germain H. Droplet Digital PCR versus qPCR for gene expression analysis with low abundant targets: from variable nonsense to publication quality data. Scientific Reports 2017;7(1):2409 doi: 10.1038/s41598-017-02217-x
- Ahrberg CD, Manz A, Chung BG. Polymerase chain reaction in microfluidic devices. Lab on a Chip 2016;16(20):3866-84 doi: 10.1039/C6LC00984K
- Garibyan L, Avashia N. Polymerase chain reaction. J Invest Dermatol 2013;133(3):1-4 doi: 10.1038/jid.2013.1
- VanGuilder HD, Vrana KE, Freeman WM. Twenty-five years of quantitative PCR for gene expression analysis. BioTechniques 2008;44(5):619-26 doi: 10.2144/000112776
*** |
|
 /3
/3 