金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
引言:时隔十年,David Baker团队再次挑战从头设计酶领域。日新月异,这几年在AI蛋白质结构预测的浪潮下,人工设计蛋白质门槛不断降低,近期也出现了各式各样的支架蛋白、小型功能蛋白和自组装单元蛋白的设计工作。但目前酶的从头设计依然是王冠上的明珠,让我们来回顾新酶设计的历史性节点。 一.初生代 | 照葫芦画瓢
在过去10年里,从头酶设计的方法是严重依赖于自然界的蛋白支架模板,在2011/2013年,由Richter & Andy完成的相关的设计。主要使用的技术是RosettaMatch。核心思想为将天然的反应过渡态(酶活中心)移植到其他无关的蛋白质结构支架上,并重新设计底物周围的氨基酸序列,以此来稳定酶“反应中间态”的构象能量。这种照葫芦画瓢的好处是,不需要对酶活催化过程重新设计,但由于能量函数以及设计精度不足,设计的酶也仅仅只有天然酶活性的1%左右,距离工业应用仍然十分遥远。经过几年的努力,连蛋白设计大师也开始在从头设计酶领域越发“沉寂”,设计团队的一些工作也开始转向与酶底物特异性设计相关的领域,毕竟小改酶活口袋还是相对比较容易。
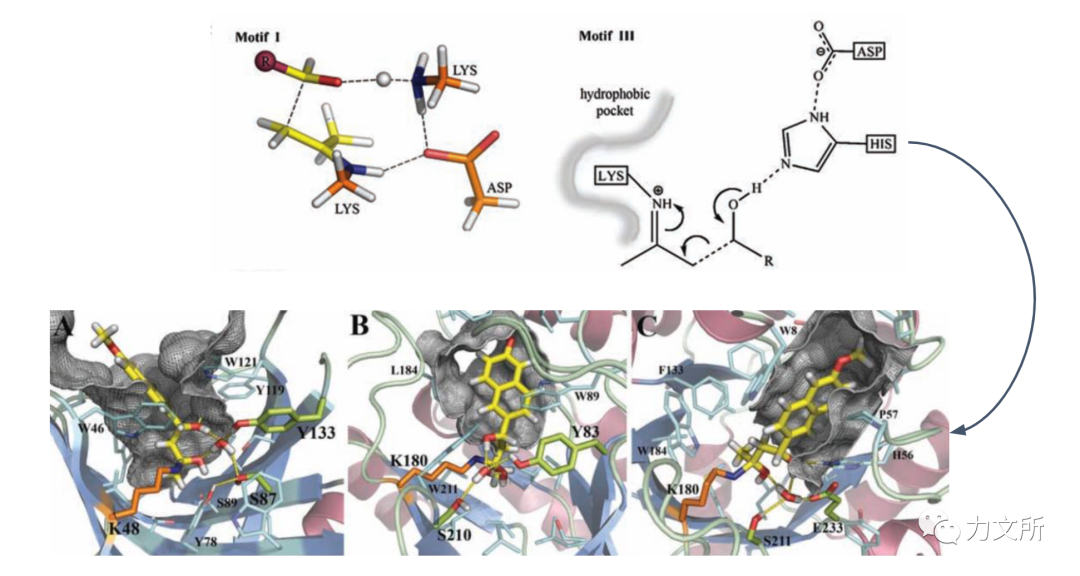
二.次生代 | 酶支架重组设计
由于移植酶活中心的难度较高,后来也出现了一批基于结构域重组的酶设计方法,比较出名的是David Baker的博士Sarel Fleishman近期的一些工作。其核心思想类似于自然界的移码突变,将同一家族中的酶在计算机中进行片段式地拼接,一方面借鉴了进化上的序列空间信息、另一方面也考虑了物理结构上的兼容性。拼接片段有个最大的便利是不需要对酶活中心进行操作,因此设计的酶大多折叠完整。
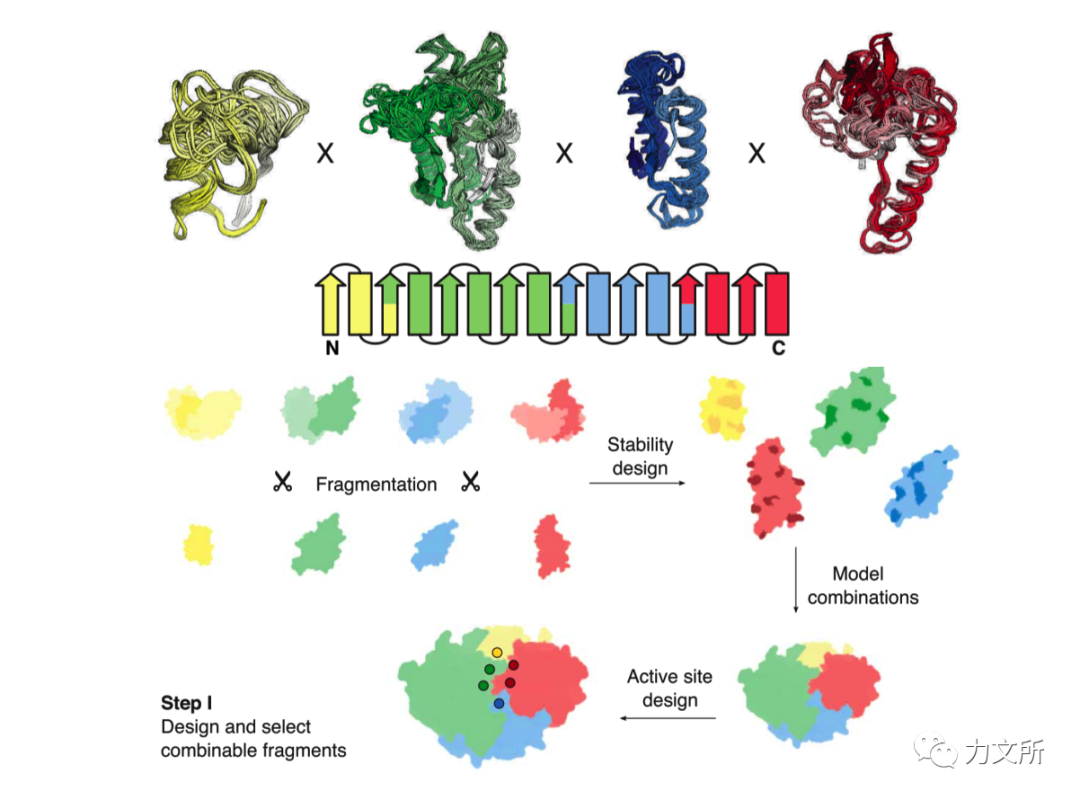
除了设计具有功能的酶意外,一些具有酶活中心移植潜力的支架蛋白设计也受到了关注,比如在2020年,David Baker团队通过Rosetta的二级片段重组的方式,对NTF-2核转运因子结构进行了遍历的生成。基于参数化的方式,探索了各种二级结构片段的组合,得到了上千个全新的支架蛋白。在后续的一些工作中,他们利用这些支架成功地设计出了一些小分子结合蛋白
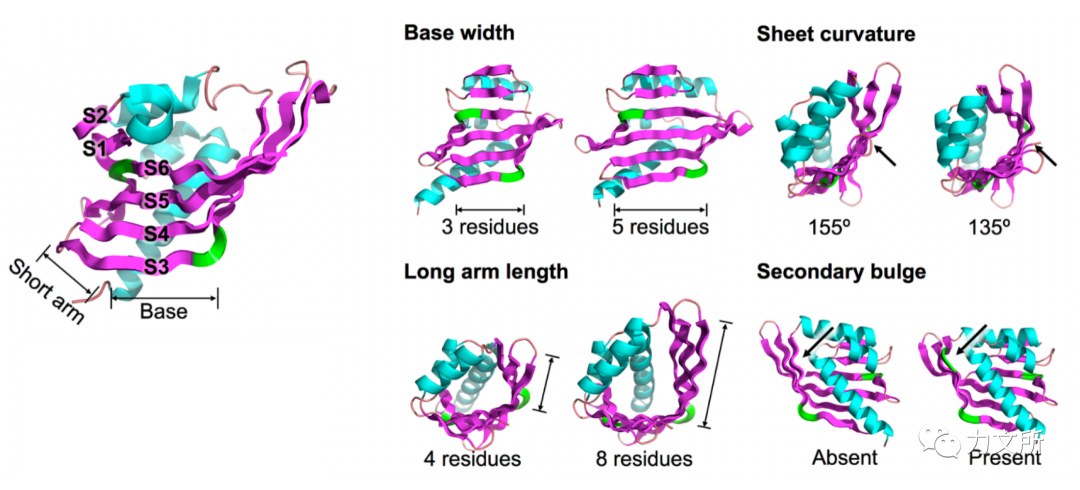
新.深度时代 | 新生酶
从过去的案例可见,新酶设计的2个非常重要的因素: 催化机理研究以及酶支架库质量。在Alphafold2后时代,这两年关于蛋白质幻想设计的工作犹如雨后春笋,其中代表例如trDesign的开创者David Baker的博士Sergey Ovchinnikov。这类方法已经成功设计了一系列的蛋白,这股春风最近也吹到了沉寂已久的酶设计领域。得益于先前在NTF-2核转运因子上的工作,此次David Baker直接使用蛋白质幻想技术生成NTF-2类似结构,但与先前的“自由幻想”以及基于基序的方法不同,此次特定对于蛋白家族进行了约束,这技术被称为“家族特异性幻想”(Family-wide hallucination)。
该算法的核心思想是将家族中保守区域的结构/序列进行锁定,而非保守的结构进行自由幻想,得益于深度幻想的能力,这些非功能化的区域一般具有极度高的稳定性。在进行功能化设计时,首先采用了在2016年提出的RifGen技术,通过Rotamer场和Rosetta能量生成上亿个氨基酸侧链和小分子的复合物构象,后续在通过RifDock将幻想生成的高质量模板和小分子复合物motif进行对接以及氨基酸侧链的进一步优化。(更多设计的实验细节可参考原文献)
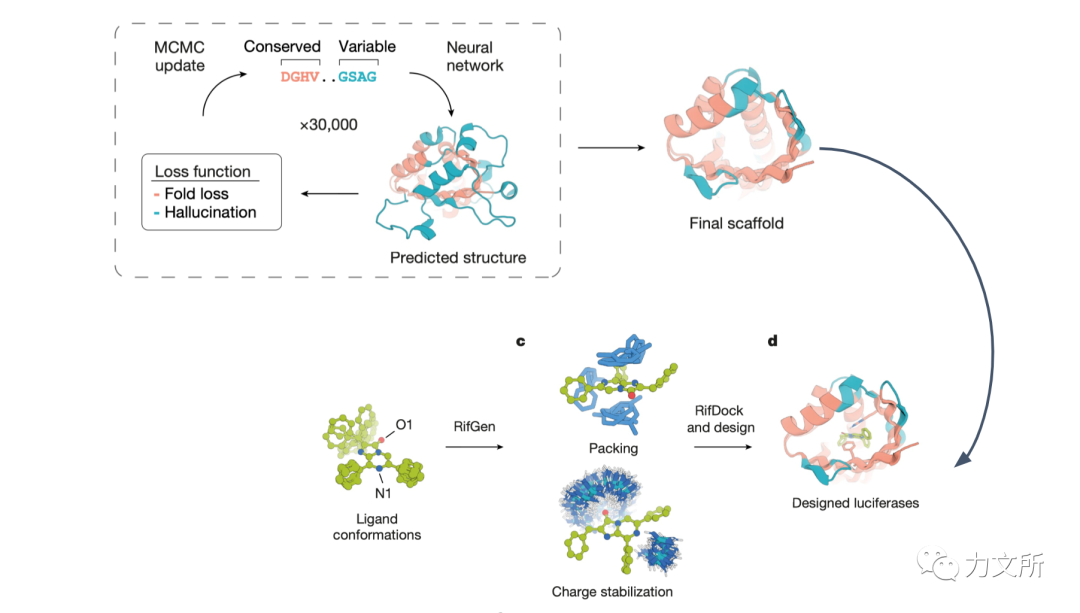
点评
David Baker首次在从头设计酶领域应用了蛋白幻想技术,成功地开发出了2款高度热稳定的新型荧光素酶,此事件可以说是从头设计酶领域的一把“新”星之火,不仅酶支架是全新的,酶催化中心结合模式亦是自然界不存在的。可谓实现了双历史性的突破,可能会在接下来的一段时间内影响业界的风向。在兴奋的同时,我们也看到目前依然存在非常多的挑战,对于目前工业应用的酶大多具有复杂的催化机理,从头设计酶依然需要深刻理解过渡态信息,除此以外催化中心的反应常由多结构域参与,更涉及金属、辅酶等因子。不排除新生设计的酶依然需要通过大量的随机突变库获得“超能”表现。
参考链接:
1. National Academy of Sciences, 115(23), pp.5968-5973. Richter F, Leaver-Fay A, Khare S D, et al. De novo enzyme design using Rosetta3[J]. PloS one, 2011, 6(5): e19230.
2. Jiang L, Althoff E A, Clemente F R, et al. De novo computational design of retro-aldol enzymes[J]. science, 2008, 319(5868): 1387-1391.
3. Kipnis Y, Chaib A O, Vorobieva A A, et al. Design and optimization of enzymatic activity in a de novo β‐barrel scaffold[J]. Protein Science, 2022, 31(11): e4405.
4. Basanta B, Bick M J, Bera A K, et al. An enumerative algorithm for de novo design of proteins with diverse pocket structures[J]. Proceedings of the National Academy of Sciences, 2020, 117(36): 22135-22145.
5. Lipsh-Sokolik R, Khersonsky O, Schröder S P, et al. Combinatorial assembly and design of enzymes[J]. Science, 2023, 379(6628): 195-201.
6. I Anishchenko, TM Chidyausiku, S Ovchinnikov, SJ Pellock, D Baker. De novo protein design by deep network hallucination. (2020) bioRxiv, doi:10.1101/2020.07.22.211482.
原文地址:https://zhuanlan.zhihu.com/p/611767490 |
|
 /3
/3 