金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
这学期在写的cs thesis刚好是做生物信息和机器学习结合的,抛砖引玉一下。作为一个已经五年没学过生物的人,老板还是满巧妙地把问题转换成了类似于自动调超参的问题的(?),需要的前置生物知识不算太多。
生物方面,我们研究的是population genetics. 在研究进化相关问题时,不仅面临数据少的问题,我们常常是不知道什么进化相关的参数导致了我们手上的数据的(evolutionary groud truth),所以这个领域比较依赖模拟出来的数据(simulated data)。近10-20年,生成这些数据的模型生物学家们手动选取一些参数[1][2],比如mutation rates, recombination and gene conversion rates, population size changes, 还有很多很多。由于是手工选取的,生成出的数据往往比较biased, 体现在对于某些summary statistics很match但是对于某些不太行,比如可能site frequency spectrum比较好但是linkage disequilibrium patterns不行[3].
有些人可能会说,直接拿各种生成网络直接一套不就好了,确实也有类似的工作[4]使用GAN来生成population genetics. 但是缺点是这样没法应用到下游任务,因为我们依旧不知道evolutionary ground truth. 把进化相关的参数想成population genetics的label可能会有助于理解。我们不仅需要data, 还需要相关的label(尽管也是假的但是起码和data得保持一致)所以只拿生成网络生成数据并不能解决问题。
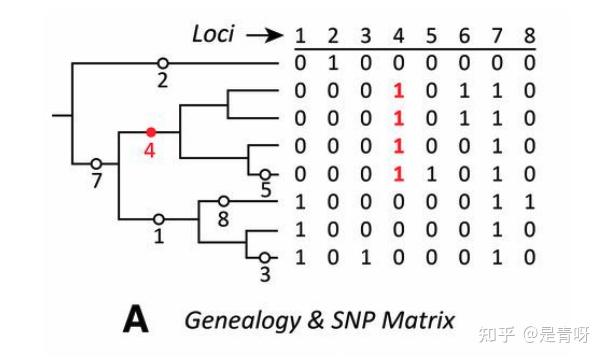
The genealogy of eight haplotypes with eight SNPs from four individuals forming aSNPs matrix which is typically used in the study of population genetics
如图,左边是genealogical tree,右边是SNP matrix[5]。SNP matrix是一种记录单个核苷酸在个人以及整个population中的不同的方法,可以比较方便地拿来当神经网络的input. SNP matrix可以被左边的genealogical tree生成,而genealogical tree可以被上文提到的传统的生成模型生成,只不过需要选择参数。
因此,我们可以用一个GAN,generator生成的不是population genetics data而是参数,这些参数会被喂进一个传统的simulator里面从而生成population genetics,然后discriminator来判断一下这个生成的是真是假[6]。
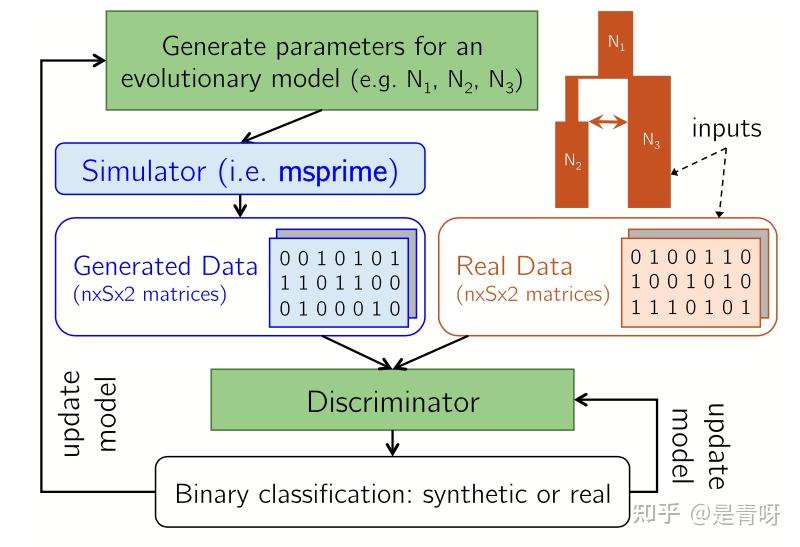
所以这事其实和生物关系没那么大了(?)因为问题就是我怎么生成参数,然后这些参数丢进一个已知的生物学家们搞的模型里面后生成的数据和真实的一样。可以尝试用不同的geneator来看效果,图上这篇工作里面的parameter是选定的只是值在变,也可以尝试允许geneator自己选参数。如果要用上生物知识的话也不是不可以,因为SNP包含方方面面的信息,如果局部特征提取然后再处理可能也有意想不到的效果。总之,在把DNA数据变成matrix之后事情就可以变得比较侧重ML了(。。。 |
|
 /3
/3 