金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
课程:分子生物学
主题:真核生物的RNA聚合酶
细菌只有一种RNA聚合酶,通过置换σ因子而适应环境变化的需要。真核生物则具有三种RNA聚合酶,它们识别不同的启动子。
1 真核生物RNA聚合酶的多种形式
1.1 猜测
核糖体基因(ribosomal)与其他细胞核内的基因存在许多差异:
- 碱基组成(base composition)不同。如大鼠rRNA基因的GC含量(60%)高于其他基因(40%)。
- 核糖体基因显著重复(unusually repetitive)。
- 定位不同。核糖体基因存在于核仁(nucleolus),其他基因存在于核质(nucleoplasm)。
因此,真核细胞的细胞核内至少存在两种RNA聚合酶。
1.2 三种细胞核聚合酶的分离
1969年研究者利用DEAE-Sephadex离子交换层析分离出三种RNA聚合酶。随着硫酸铵浓度增加,析出组分的RNA聚合酶活性呈现出3个峰值。
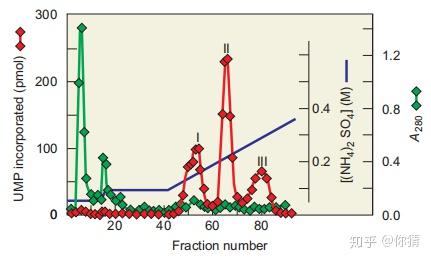
红色:通过检测RNA中标记UMP的掺入量而测得的RNA聚合酶活性。蓝色:硫酸铵浓度。绿色:280nm处蛋白质吸光值。图片来自参考资料[1]
这三种依次析出的RNA聚合酶分别被命名为RNA聚合酶I,II,III(RNA polymerase I, II, III)。
进一步分析表明,聚合酶I主要定位于核仁中,聚合酶II,III主要定位于核质中。
1.3 三类RNA聚合酶的作用
- RNA聚合酶I催化合成大的rRNA前体(precursor)。
- RNA聚合酶II催化合成不均一核RNA(hnRNA),大多数核内小RNA(snRNA)等。
- RNA聚合酶III催化合成tRNA,5S rRNA前体等。
图片来自参考资料[1]
三种RNA聚合酶对α-鹅膏蕈碱(α-amanitin)的敏感性不同,如图所示。RNA聚合酶I对它耐受,而RNA聚合酶II对它极度敏感。
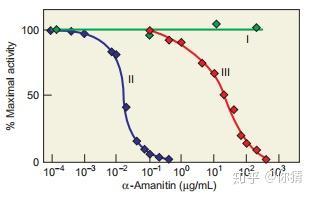
图片来自参考资料[1]
在α-鹅膏蕈碱浓度增加的条件下温育小鼠细胞核合成标记的小RNA,通过离心分离出小RNA,PAGE电泳,测定凝胶的放射性。
结果显示,高浓度α-鹅膏蕈碱抑制5S rRNA和4S tRNA前体的合成,与α-鹅膏蕈碱抑制RNA聚合酶III的模式相匹配。
2 RNA聚合酶的亚基结构
我们很难区分纯化出来的多肽中哪些是RNA聚合酶的真正亚基,哪些是与酶紧密结合的无关蛋白。解决问题的途径有:
- 将各个亚基分离,重新组装成有活性的RNA聚合酶,从而判断哪些亚基是必须的。
- 克隆所有推测亚基的基因,然后使某些基因突变,从而判断哪些亚基是必须的。
2.1 共同特征
- 三种RNA聚合酶都包含两个大亚基及一些小亚基;
- 存在5个共有亚基(Rpb5,6,8,10,12)。
2.2 RNA聚合酶II的结构
目前认为酵母RNA聚合酶II有12个亚基,按大小依次命名为Rpb1~Rpb12。
表位附加法(epitope tagging)可以获得高纯度的RNA聚合酶II:
- 通过基因工程手段在RNA聚合酶II的一个亚基(如Rpb3)上附加一个外源结构域(抗原决定簇标签,epitope tag);
- 在含有放射性标记氨基酸的培养基上培养细胞;
- 添加识别抗原的抗体,免疫共沉淀(immunoprecipitate),纯化;
- 加入强去污剂SDS,使各亚基分离变性;
- 凝胶电泳,放射自显影(autoradiography)检测。
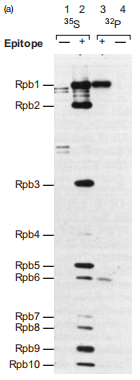
泳道2为由带有表位的酵母细胞中分离的32-S标记的蛋白质,可识别出10个亚基。Rpb11与Rpb9,Rpb12与Rpb10分别存在共沉淀现象而无法辨认。泳道3为由带有表位的酵母细胞中分离的32-P标记的蛋白质,只有两个亚基被标记,表明Rpb1,Rpb6是磷酸化的。
其中,Rpb1,Rpb2,Rpb3组成核心亚基,分别与E.coli RNA聚合酶的β',β,α亚基同源。
- Rpb1结合DNA;
- Rpb2位于或靠近酶的核苷酸连接活性位点。
2.2.1 Rpb1的异质性
最大亚基Rpb1的羧基末端有一段共有序列,为Tyr-Ser-Pro-Thr-Ser-Pro-Ser的七肽重复序列片段,称为羧基末端结构域(carboxyl-terminal domain, CTD)。
小鼠浆细胞瘤RNA聚合酶II的电泳结果显示,最大亚基似乎有三种不同的形式。
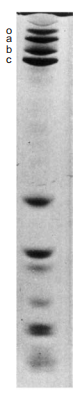
c与Rpb2对应;o/a/b似乎是最大亚基的不同形式。图片来自参考资料[1]
根据基因测序结果推测,a应为母体;
CTD抗体可与a反应但不与b反应,证实了b缺失了CTD;
CTD的两个丝氨酸(Ser)可被磷酸化(phosphorylate)修饰,使a转变为o。
在体内没有观察到b形式的存在,它可能是纯化过程中人为造成蛋白水解的产物。
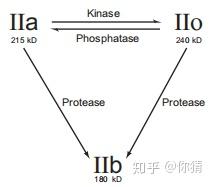
三种形式的互相转化。图片来自参考资料[1]
有充分的证据表明,含a形式Rpb1的RNA聚合酶IIA的作用是与启动子结合,而RNA聚合酶IIO主要执行延伸功能。
2.2.2 三维结构
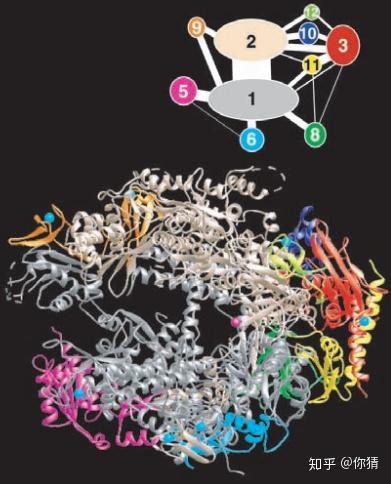
RNA聚合酶II的晶体结构(采用的是缺少Rpb4和Rpb7亚基的突变体,这主要是因为Rpb4和Rpb7亚基容易丢失使得酶不易结晶)。图片来自参考资料[1]
- 催化活性位点位于很深的DNA结合裂隙(deep DNA-binding cleft)中;
- 裂隙由碱性氨基酸残基组成;
- 催化中心存在两个Mg2+,metal A紧密结合,metal B松散结合,可能参与酶与核苷酸底物的结合过程。
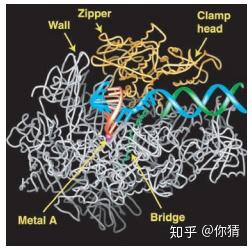
延伸复合体的晶体结构。图片来自参考资料[1]
在延伸复合体中,最大的不同是钳(clamp)环抱着DNA模板和RNA产物,从而避免转录过程中酶与模板脱离。延伸复合体具有以下几个特征:
- DNA-RNA杂交分子转过了一定角度。
- 活性中心的镁离子将单个核苷酸添加到RNA上。
- 桥梁螺旋(bridge helix)穿越酶活性位点附近的裂隙。
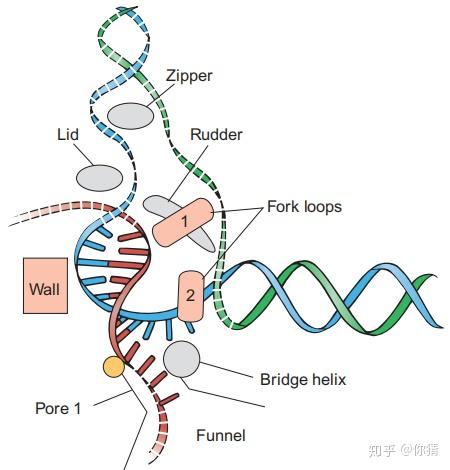
核苷酸与RNA聚合酶II中的关键元件。红色为RNA,蓝色为模板DNA,绿色为编码DNA。图片来自参考资料[1]
RNA聚合酶II中的这些元件十分关键:
- 突出于钳的环结构(the protein loops extending from the clamp):舵(rudder)、盖(lid)、拉链(zipper)。
舵(rudder):引发RNA-DNA杂交体超过9bp时的解离。
盖(lid):维持解离状态。
拉链(zipper):维持模板DNA的解离。
- 孔1(pore 1):将要添加到RNA上的核苷酸进入酶的通道。
- 桥梁螺旋(bridge helix):桥梁螺旋的直线构象允许核苷酸进入活性位点;随着DNA-RNA杂交分子的移动,桥梁螺旋也向着RNA末端弯曲;恢复到直线构象时,核苷酸入口才再次打开。
α-鹅膏蕈碱与桥梁螺旋附近位点的结合会严重抑制弯曲构象的形成,从而阻断移位(translocation),阻断RNA合成。
2.2.3 碱基选择的结构原则
RNA聚合酶II的活性位点有两个核苷酸结合位点:A位点(Addition)和E位点(Entry)。核苷酸先与E位点结合,才能进入A位点,合成磷酸二酯键(phosphodiester bond)。核苷酸在E位点和A位点之间发生了倒转。
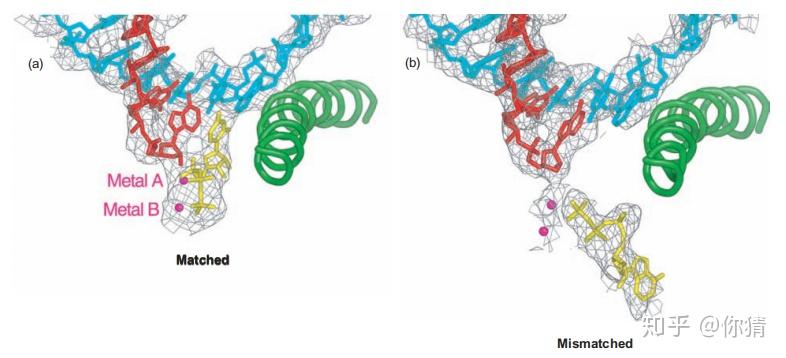
左图:正确配对的核苷酸进入A位点;右图:错误配对的核苷酸位于E位点。图片来自参考资料[1]
活性位点有两个金属离子,metal A永久地结合在活性位点上,metal B进入酶的活性位点后将被结合到正被掺入的核苷酸上。
聚合酶如何辨别4种NTP?只有当正确的核苷酸占据A位点时,Rpb1亚基的触发环(trigger ring)结构才能与底物发生相互作用,稳定底物与活性位点的连接。
2.2.4 Rpb4和Rpb7的功
前面获得的晶体结构缺少Rpb4和Rpb7亚基。12亚基RNA聚合酶II的结构显示,Rpb4/7的存在迫使钳处于闭合状态,因而模板DNA在进入活性位点之前要先行熔解。
Rpb4/7在RNA聚合酶结合关键的通用转录因子过程中发挥重要作用。推测Rpb4/7与初生RNA结合并指导其靠近CTD。
参考资料
[1] Weaver, R. (2011) Molecular biology. 5th ed. New York: McGraw-Hill.(中译本:郑用琏等译,分子生物学(原书第五版),科学出版社)
[2] 周春燕、药立波主编,生物化学与分子生物学(第9版),人民卫生出版社.
原文地址:https://zhuanlan.zhihu.com/p/159313856 |
|
 /3
/3 