金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
免疫共沉淀(Co-IP):最经典方法,在Co-IP中,使用每种蛋白质特异性抗体从细胞裂解物中免疫沉淀两种怀疑相互作用的蛋白质。如果蛋白质相互作用,它们会相互共沉淀,并且可以通过蛋白质印迹或质谱法检测。
酵母双杂交 (Y2H) 测定:Y2H 测定是一种遗传测定,它依赖于两种相互作用的蛋白质将酵母中转录因子的两个互补部分结合在一起的能力,从而导致报告基因的激活。两种蛋白质之间的相互作用可以通过监测报告基因的表达来检测。
荧光共振能量转移(FRET):FRET是一种基于显微镜的技术,它依赖于彼此靠近的荧光分子之间的能量转移。在FRET实验中,两种荧光标记的蛋白质在细胞中表达,并通过监测荧光团之间的FRET信号来检测两种蛋白质之间的相互作用。
生物发光共振能量转移(BRET):BRET类似于FRET,但使用发光蛋白而不是荧光蛋白作为供体。供体蛋白在与受体蛋白相互作用时产生光,可以使用光度计检测。
表面等离子体共振(SPR):SPR是一种无标记技术,用于测量蛋白质结合时表面折射率的变化。在SPR实验中,一种蛋白质被固定在表面上,并通过监测折射率的变化来检测第二种蛋白质与固定化蛋白质的结合。
Pull-down实验:
- Affinity pull-down assays: In affinity pull-down assays, the bait protein is engineered to contain a specific affinity tag, such as a His-tag or a FLAG-tag, which allows the protein to be selectively immobilized on a support containing a corresponding binding partner, such as nickel or anti-FLAG antibodies.
- GST pull-down assays: In GST pull-down assays, the bait protein is fused to glutathione-S-transferase (GST), which can be immobilized on a support containing glutathione beads. The interacting proteins are then identified by western blotting or mass spectrometry.
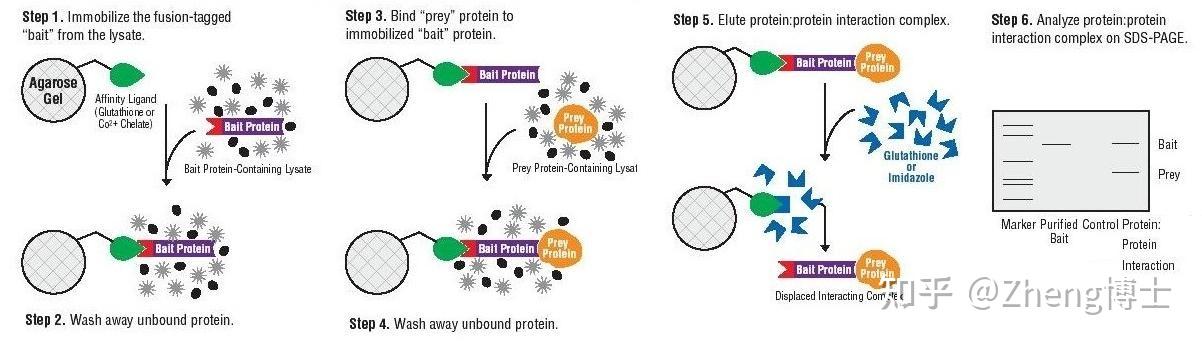
Pull-down实验 |
|
 /3
/3 