金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
众所周知,这几年大火的CRISPR基因敲除技术是受细菌免疫系统启发而来、并应用到哺乳类动物细胞上的一项突破。
这项技术主要由sgRNA定位到一个基因位点上,由Cas9酶在该位点进行DNA双链的切割,切割导致DNA修复通路的激活,使得其它的碱基加入进切割的位点,造成frameshift突变,使得基因无法被表达成功能性蛋白。
CRISPR敲除基因的具体原理是什么呢?
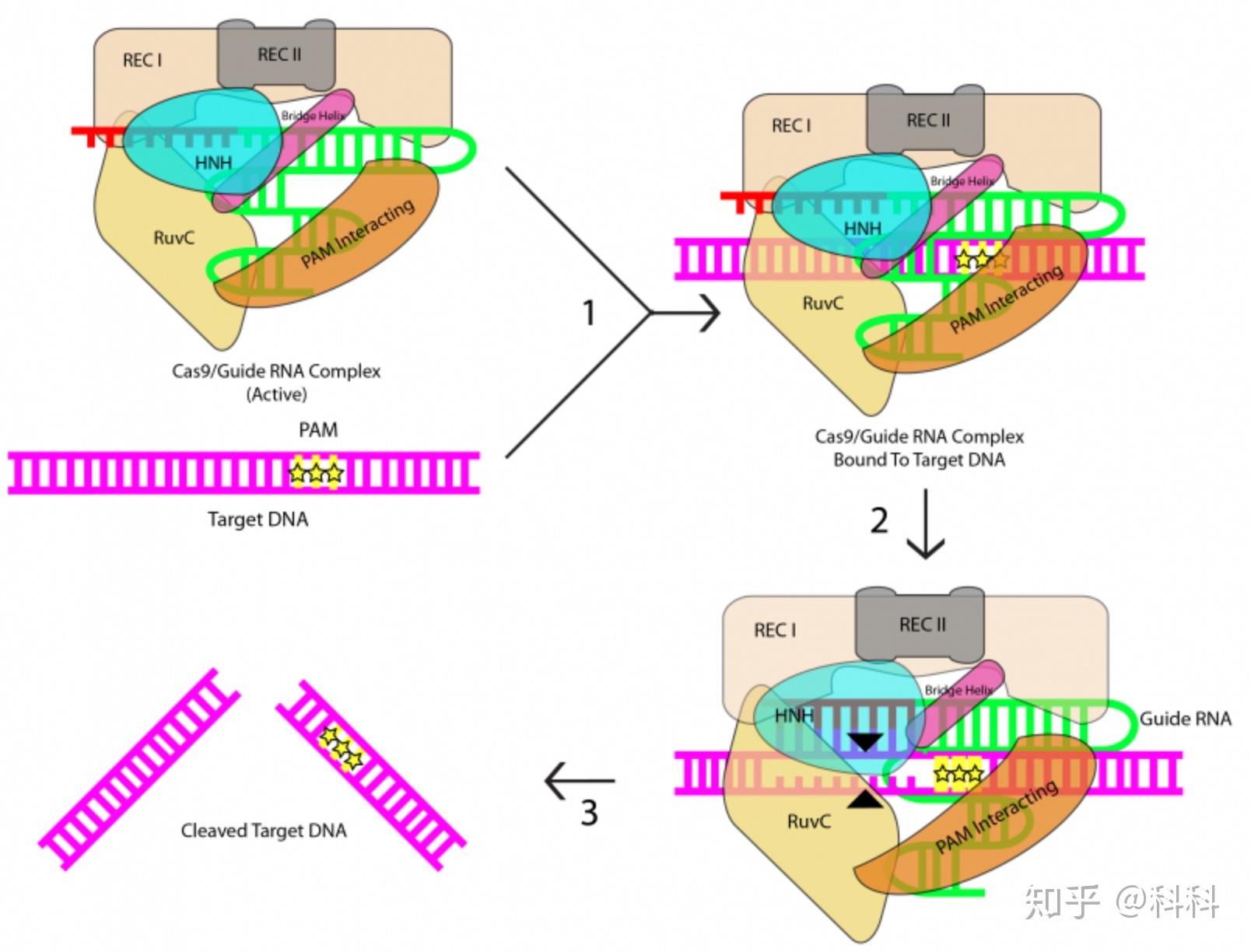
Cas9由的RECI/II, HNH, RuvC, Bridge Helix, Pam Interacting这些domain组成
- 首先,sgRNA和Cas9相结合。Cas9发生了形状上的变化,形成激活状态。
- PAM是target DNA上的一小段序列,不同的Cas9识别不同PAM。
- 拿Streptococcus pyogenesis中的Cas9来说,它识别的PAM是5′-NGG-3′
- 因此在设计sgRNA的时候,需要先找到GG,然后取其旁边的序列。
- 大部分基因序列都有GG。但如果没有GG,就用其它细菌提出来的Cas9来做。
2. Cas9会在PAM上游第三个碱基后切断双链,由Cas9的HNH和RuvC部分来进行剪切。
3. 双链断裂(DSB)之后,会有两种修复方式发生的可能:
- Non-Homologous End Joining (NHEJ)
- Homology Directed Repair (HDR)
Cas9造成基因不被表达是由NHEJ修复通路引起的,NHEJ修复通路如下(右B):
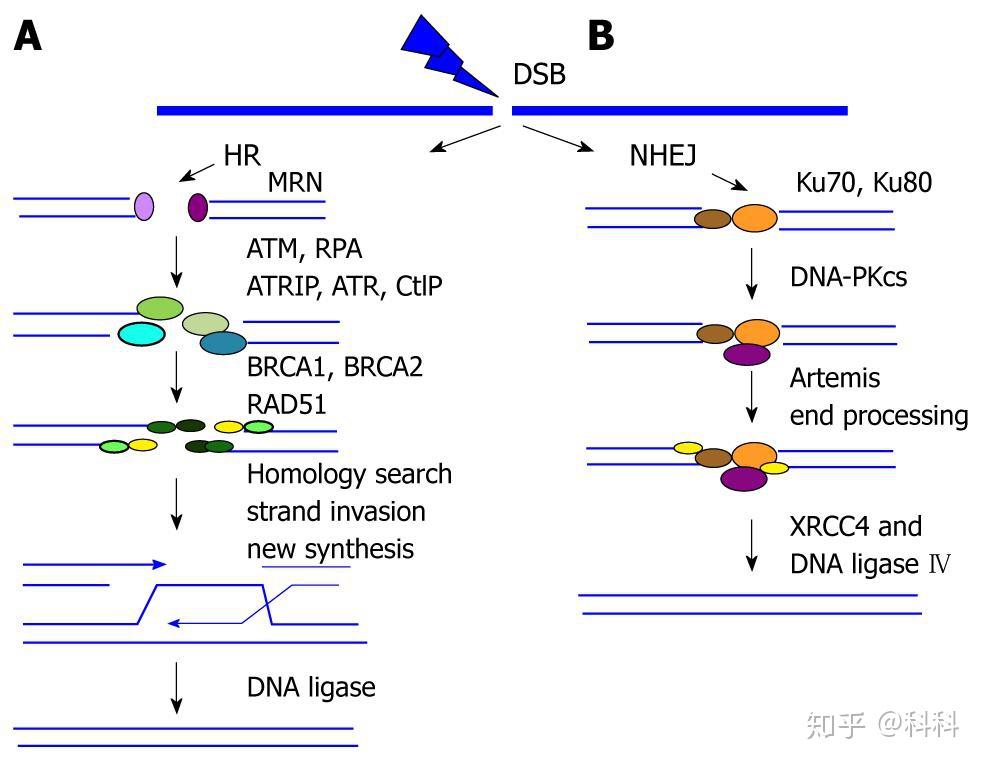
HR vs. NHEJ
- 首先,Ku70/Ku80识别DSB
- 之后,DNA-PKcs 和XRCC4-ligase IV会被招募过来,同时还会来PAXX, XLF,形成一个complex, 这个complex会把DSB修复好
- 这个过程会引发1-10个碱基的插入,由2/3的几率会引发frameshift 突变,导致基因无法被表达,也就是我们说的基因敲除。
<hr/>然而,Cas9造成的DSB并不一定会引发NHEJ,因为DSB end的碱基并没有任何损坏,这种end也叫blunt end,很容易再次粘连在一起。那么突变是怎么引发的?
事实上,即便blunt end再次粘连在一起,sgRNA会再次识别这段序列,然后Cas9会再次切,反复下去,直到发生了由NHEJ引导出的突变,sgRNA才不会识别这段序列。
<hr/>非特异性敲除的改善
sgRNA非特异性结合引起的突变一直是一个问题,那么这个问题是怎么改善呢?
张峰团队想到了一个方法,既然两条链的断裂容易引起变,我们只让一条链断裂不就行了吗?而一条链的断裂又不足以引起位点基因的突变,那么加入两个引起单链断裂的酶不就行了吗?
事实证明,这个想法是有效的,他们成功地把非特异性降低了50到1500倍,并把这个技术称为CRISPR Double Nickase,发在了《细胞》杂志上。
CRISPR DOUBLE NICKASE 敲除的具体原理是什么?
和普通Cas9不同的是,Cas9n (Cas9 Nickase)上有一个D10A的氨基酸突变,这个突变使得Cas9不再导致DNA双链断裂和NHEJ修复(一种会引来突变的修复),而是会引起单链断裂和BER修复(一种不会引起突变的修复),如下图。
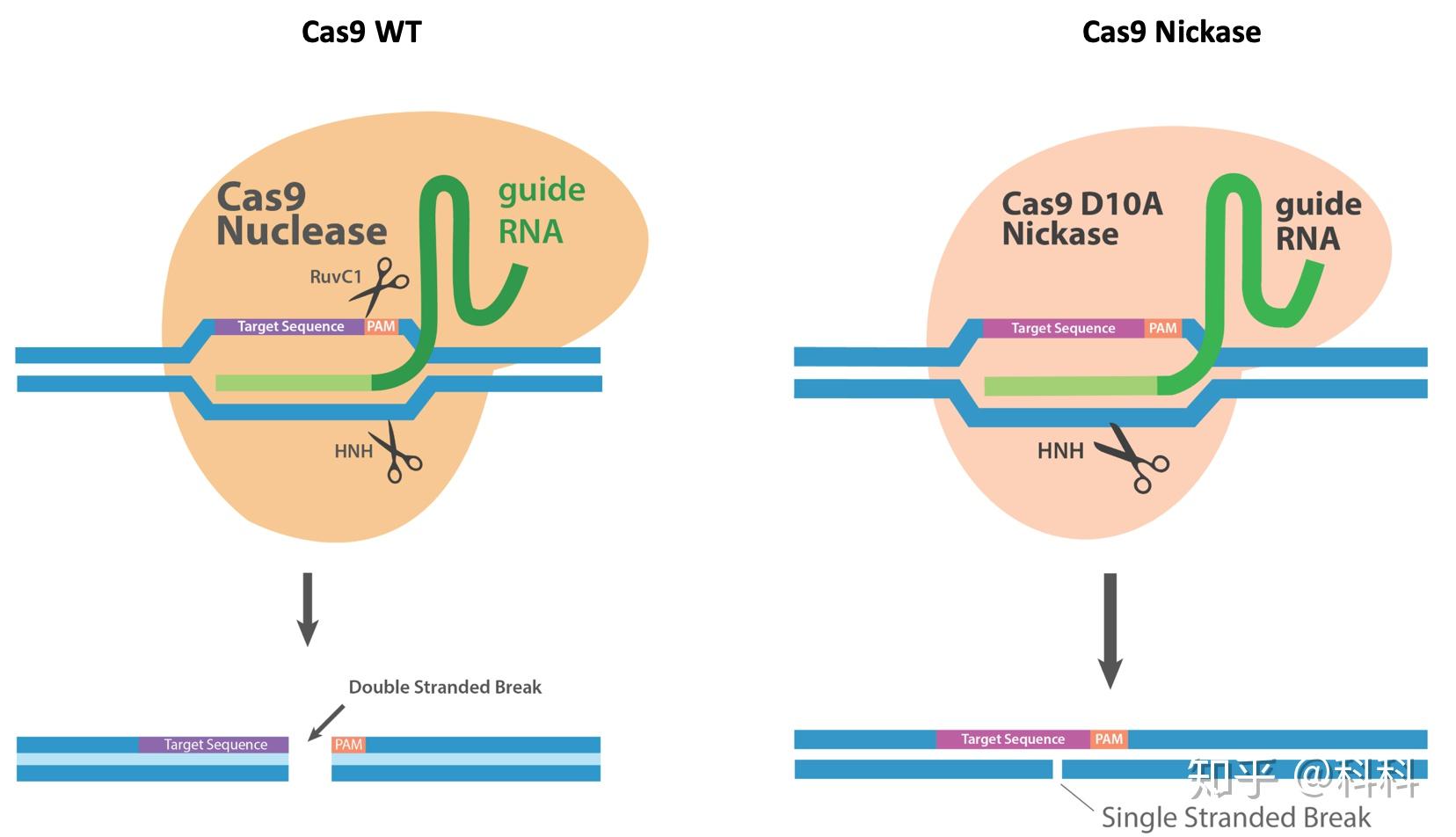
左边WT Cas9可切割双链,右边的Cas9 Nickase只能切除一个链
利用Cas9 nickase只能进行单链剪切的特性,张峰团队想到把两个Cas9 nickase共同作用在一个基因位点上,使其形成双链断裂(DSB),而非特异性结合则不会引起DSB,这样就降低了非特异性突变。
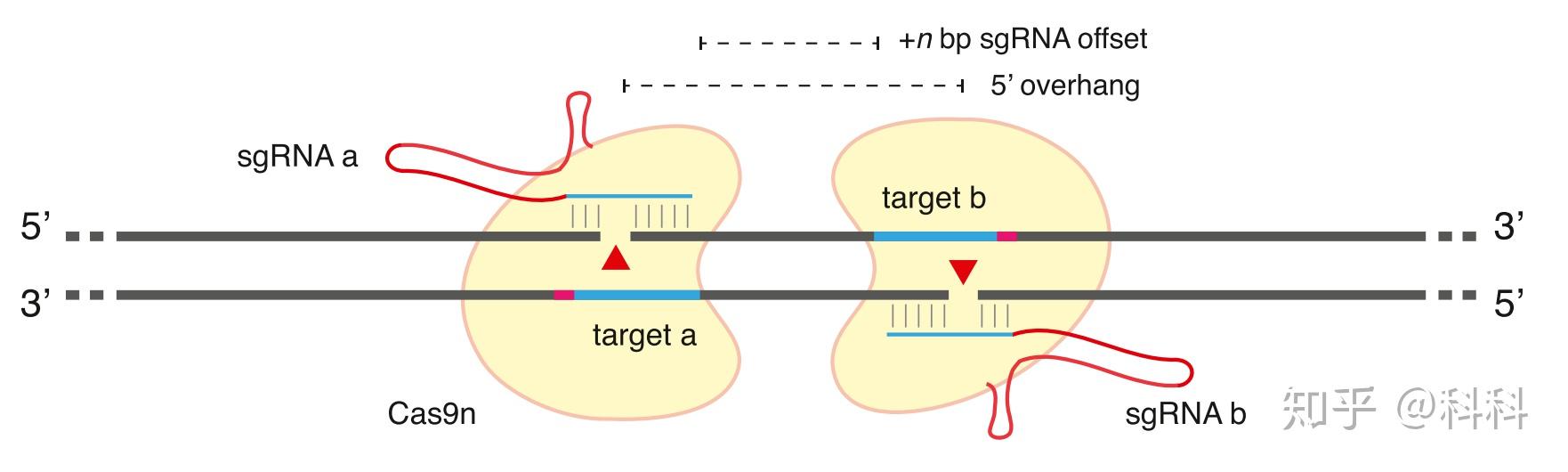
两个只能切割单链的Cas9作用在临近位点上,在该位点上形成DSB
两个cas9的距离要多远效果才最好呢?经过测试,团队发现下图的offset在-8到8bp之间引起的NHEJ最好。超过8bp到100bp效果也可以。

offset=4bp
<hr/>DOUBLE NICKASE 在实验室是如何操作的?
拿Santa Cruz的double nickase plasmids举例,里面一般包含两个plasmids(一对儿sgRNA),一个带puro,一个带GFP。
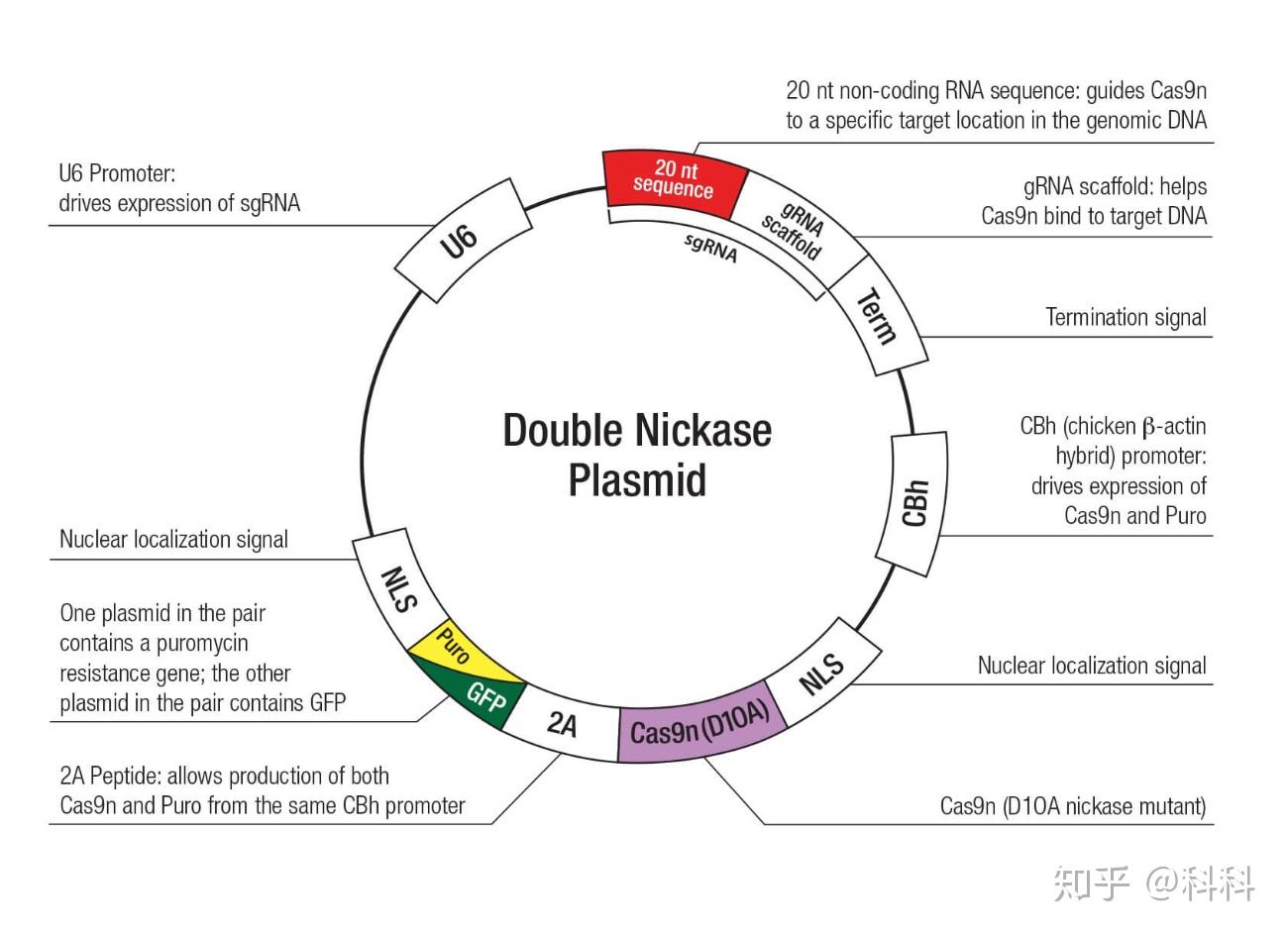
Double Nickase Plasmid
Double Nickase步骤如下:
- ~2e5 cells/ well in 6 well plate
- Transfect 1-3 ug DNA using transfection reagent
- 24-72h later, replace media
- single-cell (1-50 cells/well) sort GFP+ cells in 96 well
- after expansion, treat cells with 2-10 ug/ml puromycin
- 5d later, cell assay: WB/ RT-qPCR to check expression level
<hr/>“CRISPR系统除了用来做基因敲除,是否还有其它的应用呢?”
“当然,除了敲除基因,CRISPR还可以用来激活基因。”
<hr/>如何利用CRISPR系统来激活基因?
sgRNA的基因定位功能简直不要太好,而即便让Cas9的“剪刀”突变了,Cas9依然能和sgRNA结合。既然如此,不妨在突变的Cas9和sgRNA上加入一些促进基因转录的元素?
在Cas9上加入促进基因转录的元素其实并不难,因为Cas9是一种蛋白,只需要在蛋白上面连上一些促进转录的蛋白即可。
那么在sgRNA上如何加入这个元素呢?sgRNA可不是蛋白,它是RNA啊。RNA要怎么样才能连上蛋白?
张峰团队想到了Aptamer(一种帽子形状的RNA)和MS2(一种蛋白)可以互相结合,那么不妨把这个RNA和蛋白的结合应用到sgRNA上?
这项成果最终发表在了《自然》杂志上:
在分析完sgRNA和Cas9的晶体结构之后,他们发现sgRNA有两个loop裸露在Cas9蛋白外,即下图中红色的部分(Tetraloop 和 Stem loop2),它们不参与任何作用。这不刚好可以用来添加aptamer的小帽子吗?
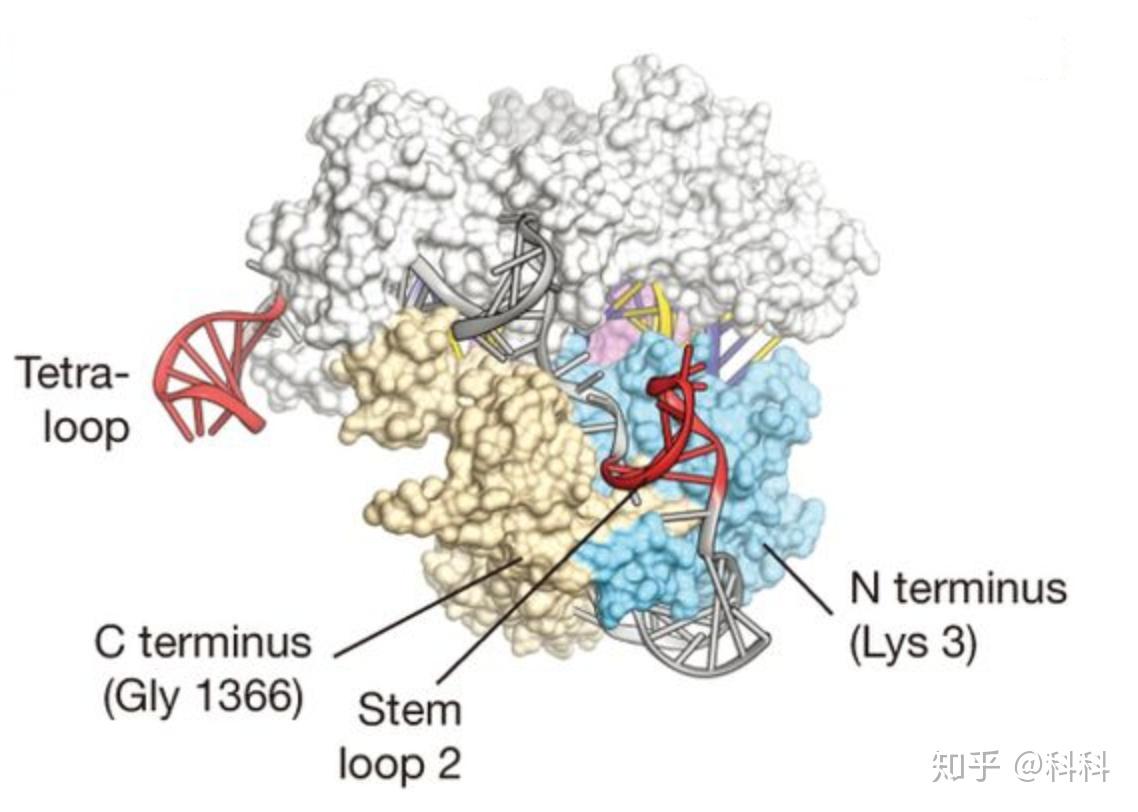
sgRNA两个loop裸露在外,即红色的部分
他们把sgRNA裸露在外且无用的两个loop替换成了aptamer,使sgRNA具有了和MS2结合的能力。这样一来,就可以在MS2蛋白上添加有助于转录的蛋白了。
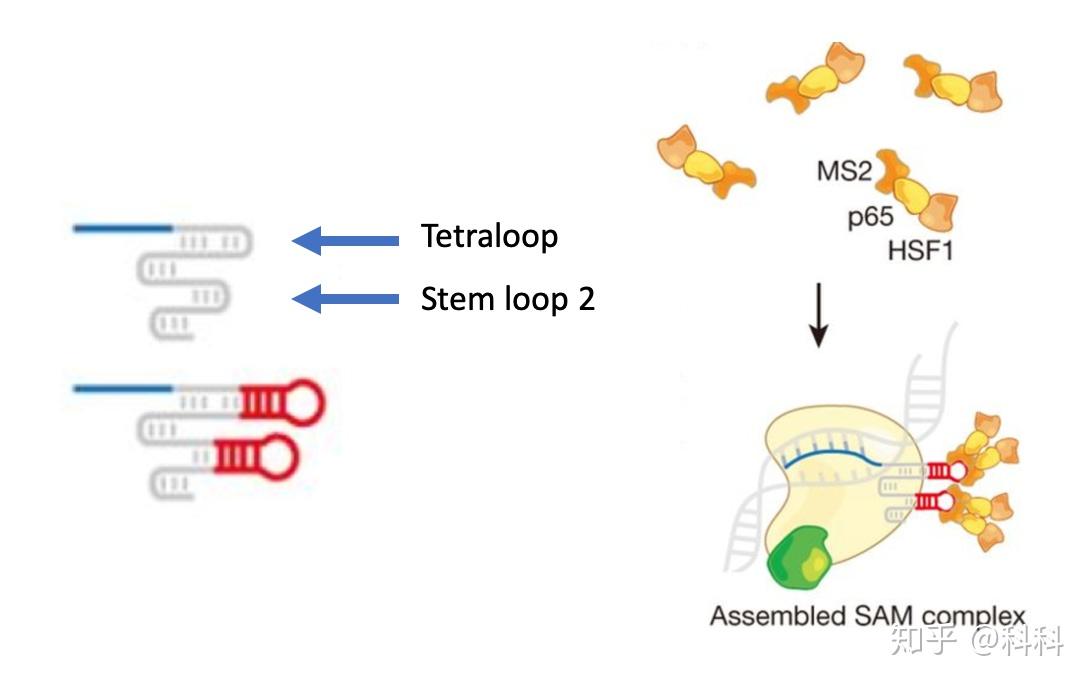
左边两个红色的圆圈(小红帽)就是aptamer,右边橙色的蛋白是MS2。
在他们提出在sgRNA上加帽子的想法前,能做手脚的就只有一个,就是在dCas9(dead Cas9)上安装TAD(Transcription activation domain),比如VP64。那么现在呢,不仅能在dCas9上添加各种元素了,还能在MS2上添加各种元素了。
在他们尝试了各种组合之后,发现VP64:dCas9 和 HSF1-p65:MS2 的组合最好, 于是就有了我们今天的SAM (Cas9 Synergistic Activation Mediators)了。SAM的工作原理如下:
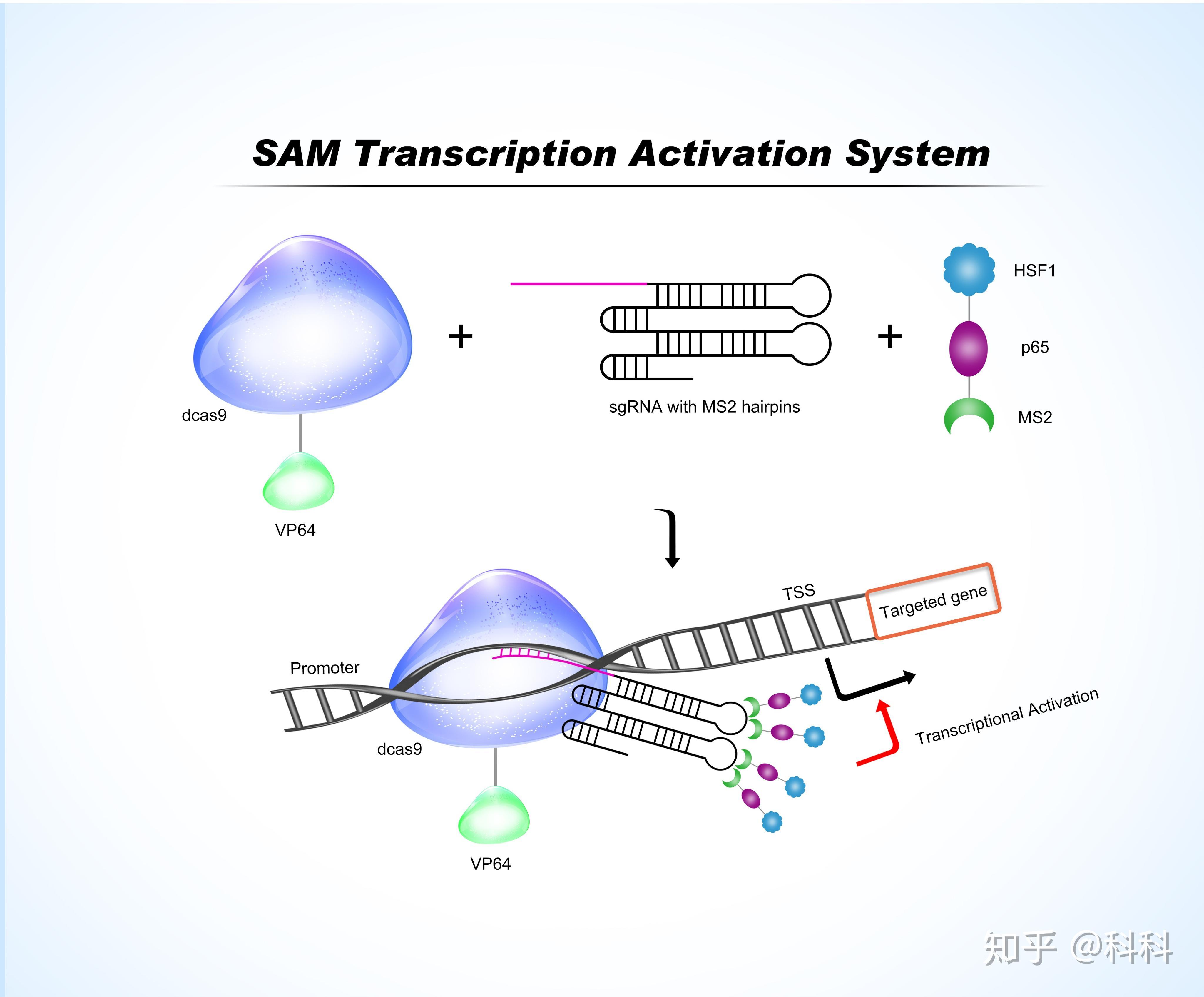
dCas9相连的VP64和sgRNA招募过来的p65和HSF1会共同促进转录
下面是一些关于这个系统中部件的介绍。
- VP64: 来源于VP16, 是它的四聚体,所以16x4=64,就叫VP64了。
- VP是virus protein的缩写,VP16来源于herpes simplex virus (HSV), 是transcription factor, 用于在被感染的细胞内促进一些基因的表达。
- 它主要有两个domain,一个用来帮助结合DNA,一个用于招募PoI II 等负责DNA转录的因子促进转录,叫Transcription activation domain (TAD)。
- 后来人们利用TAD能招募转录因子的特性,只选取VP16的TAD部分(amino acids 437-447: DALDDFDLDML),并把它复制成四份,用GS linker相连,就成了VP64。

VP64 = VP16 x 4
- dCas9: 将Cas9 负责剪切的部分,即HNH和RuvC domain, 进行突变(D10A/H840A),使其不能进行剪切。
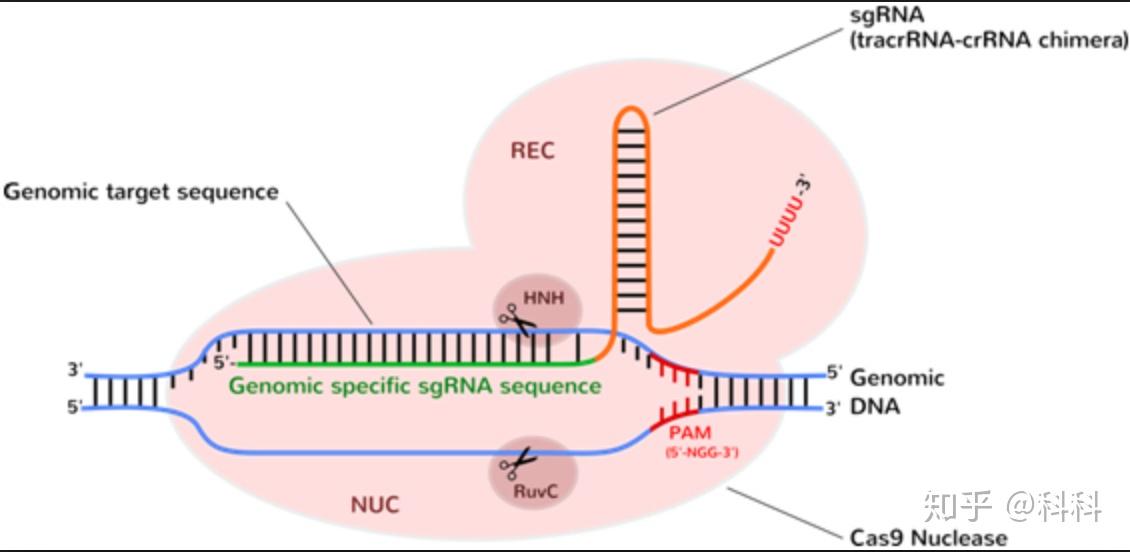
Cas9的两个domain, HNH 和 RuvC负责剪切
- HSF1-p65-MS2
- MS2: 用来和aptamer结合
- p65:选用p65的TAD部分
- HSF1:选用HSF1的TAD部分
不同转录因子的TAD部分相互叠加,有synergistic的效果,要比单一的TAD强。
PS: 目前还有好多转录因子的TAD没有尝试,效果也许会比VP64/p65/HSF1组合更好。
<hr/>
CRISPR 基因激活在实验室是如何操作的?
这里拿Santa Cruz家的activation plasmid举例:
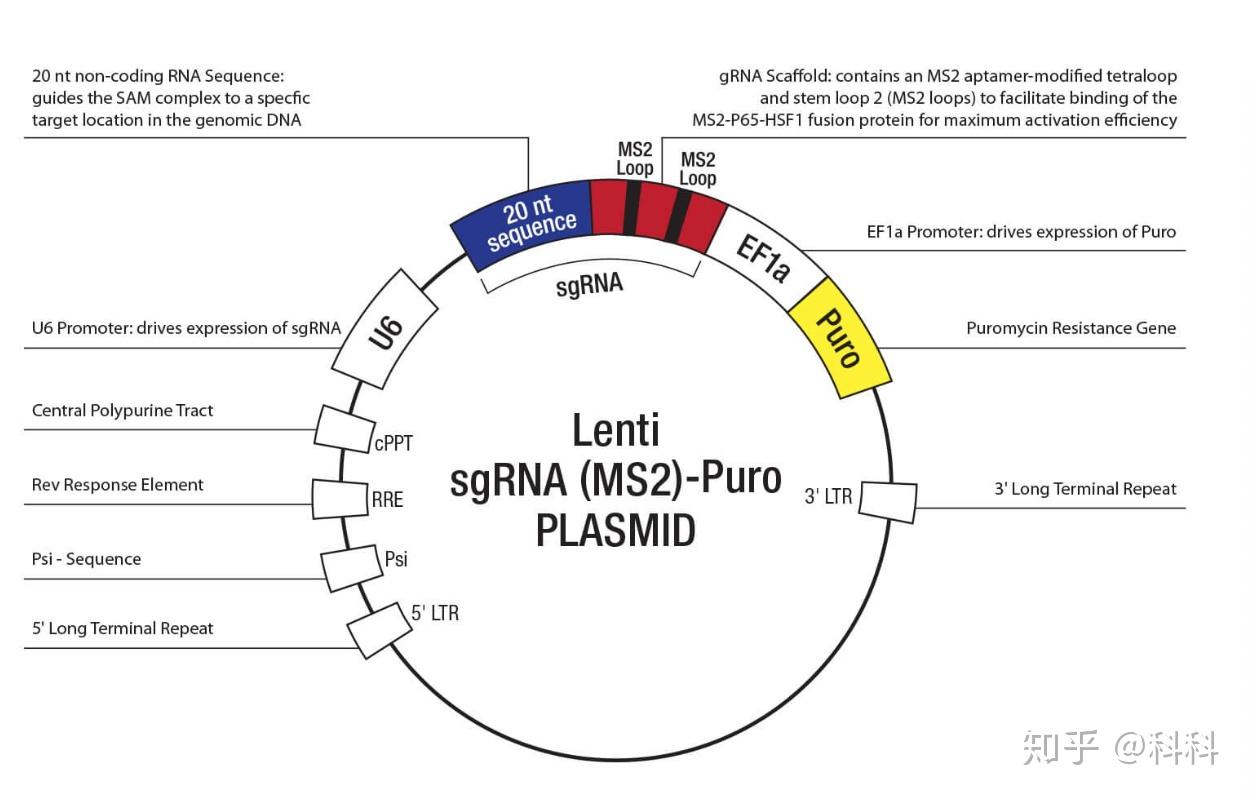
sgRNA plasmid + Puro
Activation plasmids包括三个plasmids,分别是dCas9-VP64, MS2-P65-HSF1,和sgRNA:
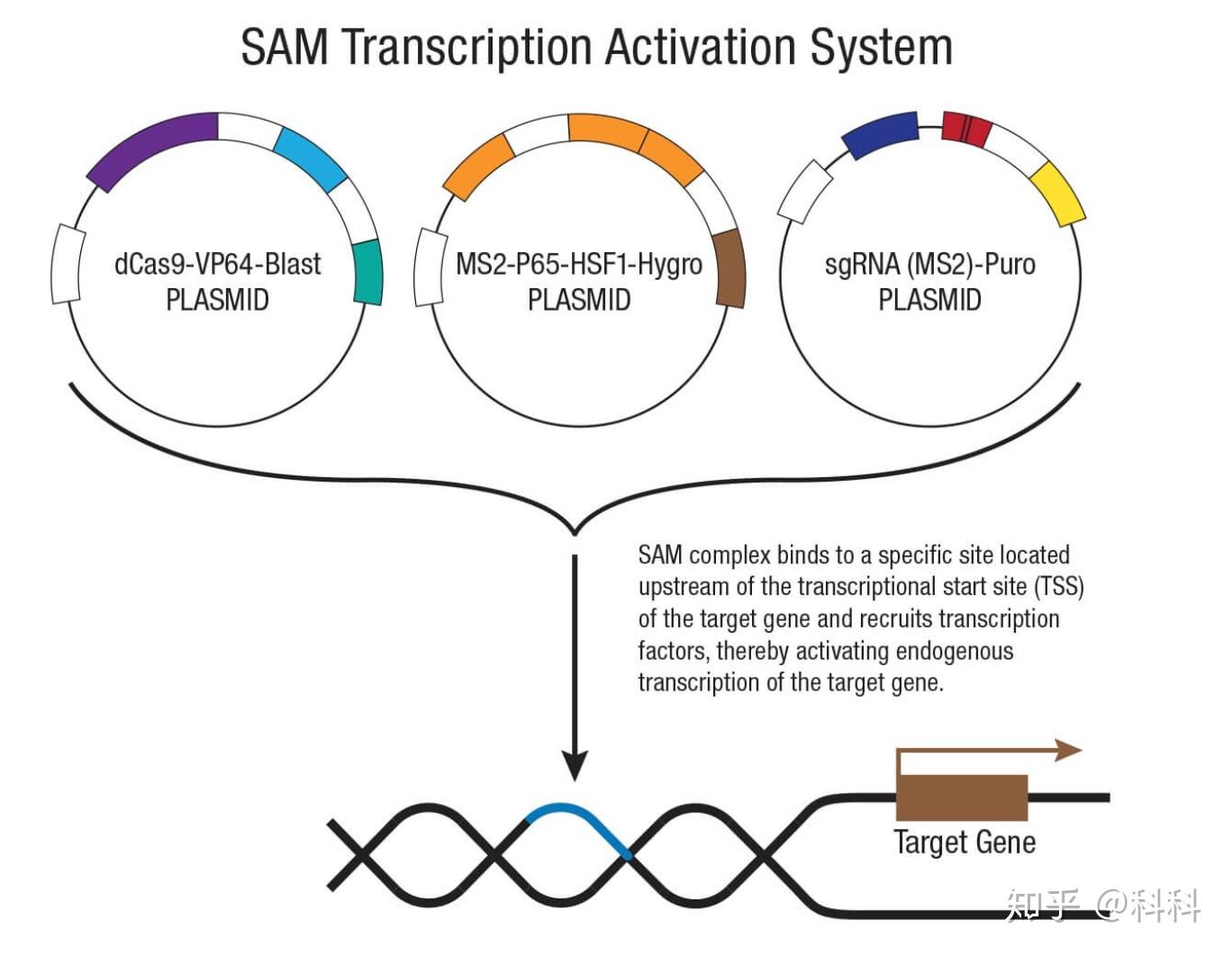
SAM实验步骤:
- ~2e5 cells/well in 6-well plate
- 1-3ug DNA, transfection
- 24-72h later, replace media --> cell assay 48h after transfection
- antibiotics selection, puro: 1-10ug/ml, hygro: 200-500ug/ml, blasticidin: 1-20ug/ml
- replace media with antibiotics every 3 days until resistant colonies are formed
- pick colonies using 10 ul pipet tip under 10x lens microscope
- grow in 96 well and expand
- cell assay again: WB and RT-qPCR
<hr/>以上就是利用CRISPR系统进行基因激活和敲除了。是不是很简单呢?科科。
原文地址:https://zhuanlan.zhihu.com/p/58556895 |
|
 /3
/3 