金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
颠覆生物制造:自动化与创新融合,重塑非传统微生物DBTL【 @金平宇 】
【1】亮点;关键词;生物生产的传统与新兴微生物平台调查;利用模型微生物宿主推进生物生产的生物铸造厂
<hr/>自动化在生物制造厂中的细胞工厂设计和测试
自动化技术正在日益融入到合成生物学和代谢工程中[1]。典型地,构建含有相对简单的合成代谢途径的细胞工厂至少需要十个独立的DNA模块。这些遗传部件必须以多种配置重新排列,以生成产生合理滴定量的所需产品的变体[2],这使得手动构建DNA组合既不切实际又耗时。相反,自动化提供了有效、标准化的细胞工厂设计和构建解决方案。一个主要挑战是快速、有效地测试在给定宿主中植入的大量遗传结构的组合,同时理解每一种修改对最终表型的生物学意义。生物制造厂具有处理大量遗传结构的能力,这些结构在由机器人液体处理器控制的集成分子生物学设施中进行处理,结合高通量分析并由专用软件支持[3][4]。图1A展示了一个用于快速原型设计的DNA结构的生物制造厂的简化方案。学术生物制造厂的一个显著方面是,整个工作流程在DBTL(设计-构建-测试-学习)周期的所有级别上仍未完全自动化或集成。细胞工厂建设的中间步骤,需要大量的优化,大多数情况下在平台外手动(至少部分)执行。此外,自动化在提供有关新陈代谢基本信息的力量到目前为止还没有被充分利用。
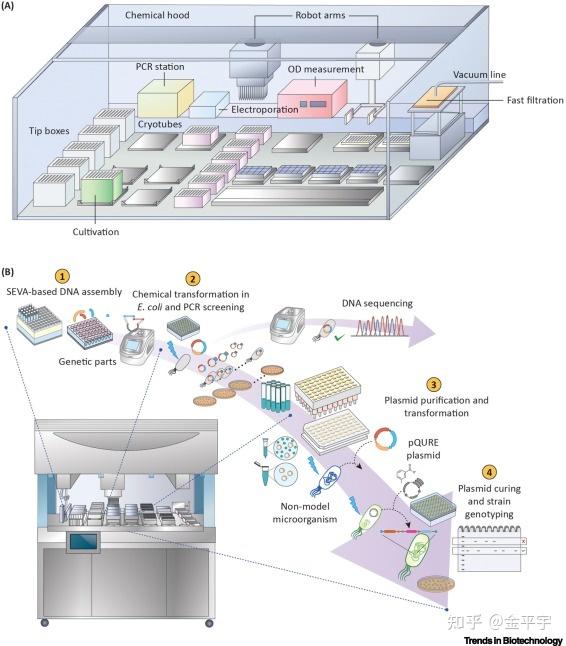
图1:在生物工厂中自动化微生物生理和代谢的基础研究
(A)微生物原型设计的自动化平台的通用方案。根据其功能,不同颜色或位置展示了机器人液体操作组件。该平台包括PCR机(黄色)、电转化站(浅蓝色)、培养设备(绿色)、分光光度计(淡红色)、处理用机器人手臂(顶部)、吸头盒(白色)和快速过滤单元(右边,浅橙色)。所有这些组件都放置在化学罩内。
(B)通过快速质粒构建、转化、质粒纯化、治愈和基因分型介导的自动化基因工程。流水线中的不同步骤如下所示:
1. 通过PCR从标准部件(Standard European Vector Architecture,SEVA)进行遗传部件装配;
2. 将PCR产品转化到化学感受态大肠杆菌后进行测序确认;
3. 质粒DNA纯化和转化到感兴趣的非传统宿主;
4. 质粒消除和单克隆基因型检查。
原则上,整个过程可以在4-5天内完成,并在几乎任何革兰氏阴性细菌宿主上产生菌株变种库。 传统微生物宿主——主要是大肠杆菌的正向工程——利用了生物制造厂在DBTL的“设计-构建”阶段的潜力[5]。最近的两个例子说明了半自动化的方法,用于改造大肠杆菌进行(2S)-黄烷酮和十二烷醇的生物生产。
第一个案例,是通过在自动化管道中组装表达结构的组合库,优化不同黄酮的生物合成过程[6]。设计基因部分以完全兼容通过配体循环反应的DNA组装,通过液体处理机器人平台执行硅藻挖掘工作流程。应用这种半自动化的DBTL程序,使用甘油作为原料,实现了高滴定量的柚皮素(484 mg/l)、松香素(198 mg/l)、毛果杨梅素(55 mg/l,通过咖啡酸喂养)和同型毛果杨梅素(17 mg/l)[6]。
第二个例子,使用基因库和核糖体结合位点(RBS)系统地操作了基因表达强度,以进行十二醇的生物生产[7]。两个连续的DBTL周期被实施,以通过测试从大肠杆菌MG1655衍生的60种工程菌株,增强从葡萄糖形成的产品。在第一次迭代中,通过采用一组预测在强度上存在差异的RBS,调节了不同的载体蛋白(ACP)/酰基辅酶A(CoA)还原酶基因的表达。硫酯酶和酰基辅酶A合酶基因也被结合在这些设计中。接下来,这些实验中生成的蛋白质浓度和产品产量数据被送入一个机器学习算法,以预测进一步的优化步骤。基于硅藻预测的第二次迭代,相对于第一周期的基础菌株,十二醇滴定量提高了21%(0.83 ± 0.13 g/l)。重要的是,Opgenorth及其同事的研究[7]揭示了实施机器学习算法提出的设计的缺点。这些挑战包括:
- 使用现有的RBS,蛋白质含量的可预测性有限;
- 路径蛋白的非目标效应(例如,FadD酰基-CoA合酶的明显毒性);
- 改变单一RBS引起的整个操纵子效应(例如,极性);
- 使用训练集中的密切相关(而不是全组合)结构,掩盖了在完整数据空间中的局部趋势,以及需要大量的数据集,可靠地训练机器学习程序。
另一项近期的研究描述了一种半自动化流程,以优化工程化酿酒酵母菌株产生五环三萜类化合物白桦酸的生产。作者们采用了SCRaMbLE[8],一种体内诱导合成染色体删除的系统,目的是生成一个多样化的菌株库。白桦酸途径模块的设计和构建阶段手工执行,而测试部分则通过实施自动化的高通量筛选方法执行。因此,这篇文章描述了使用酵母细胞工厂进行全自动生物生产过程的最接近的例子之一,具有潜力增加更多步骤以实现完全自动化[9]。另一种真菌物种,假土壤曲霉,已经被深入地、多组学指导的工程应用于糖依赖性合成聚合物前体3-羟丙酸(3-HPA)的生物合成-在生物反应器培养中达到最高滴定量0.88 ± 0.11 g/l[10]。该方法基于蛋白质组学和代谢组学,识别出能够将流动量从预期的3-HPA合成路线引走的上调蛋白。建立了一个依赖于β-丙氨酸的合成途径进行产品形成,通过删除丙酮酸缩酮酶基因(Apald6),负责3-HPA降解,重定向了乙酰辅酶A的流动[10]。
本节讨论的文章提供了关于自动化潜力的原理证明例子。这些开创性的研究也暴露出一个事实,即总体而言,模型生物是生物工厂的首选宿主。只有当细菌宿主的调色板大大扩展到超过大肠杆菌,尤其是用于生产非琐碎化学品时,生物工厂的全部潜力才能得以实现。这项努力需要一个自动化的流水线,以实现对非模型生物的合成工具集的应用,配合基于组学的对其新陈代谢和生理功能的基础知识的扩展。生物工厂如何在这方面成为关键参与者,将在下一节进行讨论。
通过自动化和扩展合成生物学工具箱,为基于非传统宿主的细胞工厂铺平道路
在体内构建和组装DNA模块以及全新途径设计是细胞工厂发展的关键部分。将这些技术扩展到非传统宿主可能标志着从缓慢的驯化过渡到理性设计,克服复杂的代谢网络和有限的微生物生理知识所带来的障碍[11]。然而,为生物技术主力建立的遗传工程方法很难转移到非模型宿主,因为物种间存在功能不兼容性(例如,RecA介导的重组效率低[12])。最近为非模型生物定制的一些尖端技术[13],仍有大量的改进空间,尤其是在准确描述这些替代宿主中的工具方面。将非传统微生物合并到生物工厂流程中,不仅会导致开发新的(可能优越的)细胞工厂,而且还将激发对这些宿主的基础研究。生物工厂可以产生大量的表型和组学数据,如果这些数据被广泛地提供给科学界,就会成为指导学术界和工业界研究的资源。对新近分离的微生物及其工具箱的表征,这是一个曾经需要数十年的努力,现在可能被简化,并在一部分的时间和成本内完成。我们稍后将讨论假单胞菌的基因工程,以示例说明生物工厂可能发挥的作用以实现这些目标。
工具箱优化和大规模多路复用是DBTL循环构建阶段的两个重要方面,这能开启非传统宿主作为细胞工厂的潜力。假单胞菌的基因工程已经针对手动工作流进行了优化,但可以进一步作为自动化设定进行开发。到目前为止,删除P. putida中基因的最快协议包括将自杀质粒共整合到感兴趣的基因座[14]。整合位点和要切除的区域由自杀质粒中两个同源臂(Homologous arm, HA)的序列决定;这些臂可以由用户自由选择,以介导在细菌染色体的任何位置上的同源重组(Homologous Recombination, HR)事件[15]。通过在HA内加入遗传元素,这些特性同样可以在目标位点上整合。I-SceI归巢巨核酸酶,由来自助手质粒的基因提供,在转,引入双链DNA断裂,从而强制进行第二次HR事件,该事件从基因组中移除质粒骨架和目标区域。通过将合成控制整合到质粒复制中,即所谓的pQURE质粒工具集,这种策略得到了升级,该工具集允许通过向培养基中添加3-甲基苯甲酸来严格控制质粒的传播[16]。
然而,这些方法的潜力尚待充分利用。例如,自动化可以用于在多个迭代回合中生成大型构造库。为此,可以采用一个克隆标准,例如,标准欧洲矢量架构(Standard European Vector Architecture, SEVA)[17],以模块化的方式组装复杂的电路,生成与许多革兰阴性宿主兼容的广宿主范围构造,并方便遗传部分的重复利用。
在这里,我们提出了一个快速基因工程的非传统革兰阴性细菌的原型工作流程,通过自动化和并行化的结合(图1B)。这个工作流程包括:
- 并行和自动装配符合标准的模块化DNA部分;
- 电转化E. coli与装配的构造;
- 高通量纯化质粒DNA和转化所需宿主与质粒库;
- 转化自愈合pQURE向量和基因型鉴定结果质粒自由菌株变异体。
这个工作流程中的所有步骤都可以通过集成在机器人平台中的自动液体处理器轻松控制。
虽然在此工作流程中提到的所有技术都已知可以单独应用于P. putida,但需要在其他宿主中进行快速和自动化的功能元素和部分(例如,抗生素标记,基因表达系统,HR机制和效率,转化协议,和反向选择标记)的原型制作以验证其性能。在这样的流程中使用的质粒可以使用广泛使用的技术(例如,USER克隆[18],Golden Gate克隆[19],Gibson组装[20]或SureVector[21])组装,根据选择的方法,最近已经提供了自动化协议[22]。遵循标准化克隆策略,一旦创建,大部分部分都可以用于多个构造,例如,质粒背骨。考虑到在整合质粒中作为HAs所需的特定PCR扩增子的大小相等(约500bp),无论目标如何,这些HA可以以高通量的方式获得,例如,96孔板(图1B中的步骤1)。
对于许多克隆策略,DNA片段的组装可以在相同的布局中进行,即使没有先前的纯化[18]。一旦质粒组装完成,它们就以96孔电转化板格式转化为E. coli进行传播(图1B中的步骤2)。通过菌落PCR后续的Sanger测序验证质粒的正确性。再次,使用标准化引物集用于验证,使得这一步也可以自动化。由于Sanger测序是负担得起的,克隆效率通常很高,这种方法在经济上具有竞争力且快速,不需要DNA纯化或凝胶电泳,从而实现高通量。
图1B中的步骤3说明了在96孔板格式中使用商业可用的DNA提取试剂盒进行质粒纯化。然后通过电转化,化学转化或共轭将纯化的质粒引入到(非传统的)感兴趣的宿主中,恢复后,细胞接种到选择性培养基上选择二倍体。注意,将细菌悬浮液涂布到选择性琼脂板上以分离单个菌落是序列中的关键步骤,但是,实质上缺少完全自动化,高效率的协议以达到此目的。
该协议的最后部分(图1B中的步骤4)涉及通过在二倍体克隆中引入pQURE系统,然后进行质粒治愈来强制进行第二个HR事件。可以实施阴性选择标记(例如,sacB)以促进质粒消除,从而实现更高的通量。序列的最后一步是通过菌落PCR和Sanger测序进行基因型确认,这是在质粒构建阶段进行的。此时,菌株库已准备好移入测试阶段,即通过多组学方法促进的代谢网络分析。
<hr/>Reference:
- Gurdo, Nicolás, Daniel C. Volke, and Pablo I. Nikel. &#34;Merging automation and fundamental discovery into the design–build–test–learn cycle of nontraditional microbes.&#34;Trends in Biotechnology(2022).
|
|
 /3
/3 