金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
本帖最后由 萝卜涨价了 于 2018-9-21 编辑
分子生物学的一个基本技术是,从一个文库的复杂DNA混合物中分离出一个稀有的克隆。从一个高度复杂的文库中分离cDNA或基因组克隆,通常是研究基础生物学过程以及进行应用生物学研究的第一步。对于一个2×109bp的单倍体生物基因组、平均插入大小为20000bp的典型基因组文库来说,一个单拷贝基因的出现概率大约为1/105。与之类似,对于一个衍生于表达许多不同基因的组织或细胞系、高度复杂的cDNA文库来说,一个特定克隆出现的概率也是很低的。要得到一个目标克隆,必须筛选大量的克隆,因此用膜杂交的方法( Benton and Davis1977)或表达克隆法( Wong et al.1985)筛选一个高度复杂的文库,将是一项非常费时费力的工作。
PCR能够以很大的数量级来扩增特定的核苷酸序列( Saiki et al.1985)。当筛选个高度复杂的噬菌体或质粒载体DNA文库时,PCR可以通过增加一个特定序列的丰富度,从复杂的分子克隆混合物中得到一个稀有的DNA序列,因而可以容易地从一个文库中鉴别出一个特定的克隆。这可以通过把原始的文库分为复杂度降低的亚库,再在每个亚库或亚库的组合中筛选特定的DNA序列来实现(图24-1)。含有目的克隆的亚库再进一步细分为更小的库,再对每一个小库使用同样的PCR程序筛选。几轮划分和筛选之后,原来稀有的克隆就大大地富集了,因而可以容易地得到纯的克隆( Israel1993)。
本章介绍了用PCR筛选高度复杂的DNA文库的方法。这种方法可以在短时间内筛选个高度复杂的文库,并且提供了另外一种选择来代替传统的那种费时的、需要噬菌斑或菌落杂交,或者说需要功能基因表达产物的方法。这种筛选技术的优点在于它的速度、灵敏度和简易性。一些分子生物学试剂的供应商,如 MrC geneservice公司,可以提供直接用于PCR筛选的文库。另外,本章所描述的方法需要不止一个能与模板DNA和/或PCR产物正确退火的寡核苷酸,因而增加了真阳性信号的严谨度。
要设计特异和高效的PCR引物,需要知道目的基因的精确序列信息。因为人类、小鼠和其他几个物种的全基因组序列已经测序,这些信息可以用于设计很多物种的任何一个基因的引物。在有些情况下,如用一个物种的已知序列来克隆另一个基因组序列未知的物种的相应基因时,目的克隆的确切DNA序列可能是不知道的。在这种情况下,当用噬菌斑或菌落杂交法筛选时,通常应该使杂交条件的严谨性降低,或用简并的寡核苷酸探针( Goeddel etal.1980; Toole et al.1984)。进行PCR时,由于退火的特异性降低,假阳性信号的出现机会增加,因而使用严谨性降低的退火条件,或者使用简并寡核苷酸引物时会很困难。另外,因为PCR的高度灵敏性,在实验时必须非常仔细,以防止样品的交叉感染产生假阳性结果。然而,如果用于合成引物的序列信息是可靠的,实验设计和实验操作时也很小心,这又不失为一种筛库成功率非常高的方法
一、材料
注意:标记(!)符号实验材料的正确操作请参照附录
1.缓冲液、溶液和试剂
蒸馏水
2酶和酶缓冲液
PCR混合物(可以放在一起冰冻),对于1m反应,包括:200nmo的各种dNTP;1×Vent溶液或等价物;2.5μmol的MgCl2<!>;2nmol的各引物
PCR主要混合物(每个反应必须新鲜配置):1 ul Vent DNA聚合酶或等价物;991上述PCR混合物
3.核酸和寡核苷酸
2个PCR引物寡核苷酸
1个杂交寡核苷酸
PCR正对照:10ng全基因组DNA或能产生阳性PCR信号的起始文库组分(见第⑤步)
4.培养基
LB,1L水中加入:10g蛋白陈、5g酵母抽提物、10 g NaCl,用NaOH调节pH至7.5〈!>
SM,1L水中加入:5.8gNaC、2 g MgSO4(!)、50ml1mol/ L Tris-HCl〈!)(pH7.5)5ml2%的明胶含10mmol/ L MgSO4的LB
5.特殊设备
琼脂糖凝胶电泳设备,包括澳化乙锭〈!>
6.其他
96孔板( Corning Costar)
聚酯封口膜( Nalge Nunc International)
噬菌斑杂交所需材料
7.载体和细菌菌株
用噬菌体載体构建的DNA库
用于扩增文库的细菌菌株〈!
二、方法
在筛选DNA文库之前,需要测定几个参数来建立有效的实验条件。这些参数如下。
①PCR条件。用筛库的引物改变退火温度和循环次数,以得到最大量的特异产物。模板的来源可以是将要筛选的文库(106个噬菌体颗粒)或者是10ng的全基因组DNA。在PCR过程中,噬菌体DNA释放作为模板。因此,在反应之前不需要提纯噬菌体DNA。可以使用的典型引物应该产生0.1~1.0kb的产物(16~24个核苷酸长度,50%G+C含量)。如果需要(比如,当用与不同外显子配对、横跨一个大的内含子的引物时),目的序列可能比1kb长,但产物的量可能会减少,
②确定文库中基因的丰度。用上面所建立的PCR条件,通过改变加入噬菌体的数目定量文库。能产生PCR产物的噬菌体的最小数目就是实验确定的该基因在文库中的丰度。对于一个插入片段平均为20kb的基因组文库来说,要有大于105的复杂度,以确保目的基因至少出现一次。对一个高复杂度的典型文库来说,用104~106个噬菌体做模板,可以显示基因在库中的丰度。加入的含有一个或两个目的基因拷贝的噬菌体的数目,应该用作文库筛选。
旦确定了PCR条件和基因的丰度,就可以按照如下方法进行文库筛选。
D取0.5ml新鲜的、在LB中过夜培养的细菌培养物,与0.5mlSM混合,加入含有文库的噬菌体,室温温育20min。
②加入20ml含10mmol/ L MgSO4的LB,按8×8矩阵分装到96孔板中,每孔100l将板用聚酯封口膜密封,37℃225r/min振荡培养5~6h,对噬菌体进行扩增。扩增之后,噬菌体的浓度应该增加到大概109个/ml。
大概有1/3的培养物能被均分进96孔板,在计算步聚①的加入噬菌体数时应当考虑到这一点
③将一排或一列8个孔中的噬菌体(每孔25μl)用多孔秽液器混合(参看讨论部分的可变样式)。在这一步中,当取掉封口膜和移液时,一定要特别小心,以避免样品交叉污染。如果想避免交叉污染,可以将板子短暂离心,以帮助清除封口膜上的液体。用聚酯封口膜将板子重新封闭,在4℃可以保存1个月。
④用蒸馏水按1:1稀释噬菌体,现在嘘菌体就可以用作PCR模板了。
⑤在24.5μPCR主要混合物中加入0.5μl的樸板(嘘菌体),用上面建立的PCR条件进行PCR。每个实验需要有一个负对照(不加模板)和一个正对照(即能产生阳性信号的10ng的总基因组DNA或一小份的起始文库)。
⑥通过琼脂糖凝胶电泳分析PCR产物。用澳化乙锭染胶并拍照( Sambrook and Rus-sell 2001)
⑦70℃襄空干燥凝胶,变性DNA,然后将寡核苷酸探针(末端用32P标记)用标准的DNA杂交条件与干燥的凝胶直接进行杂交,洗涤,然后放射性自显影[这一步的技术细节可以参看 Israel(1993)]。用澳化乙锭染色很容易就能看到特异的PCR产物时,这一步是可以选做的,下面会有讨论。也可以用标准技术将DNA转移到硝酸纤维素膜或尼龙膜上后再进行杂交。
第⑥步和/或第⑦步取得的据应该能鉴定出含有目的基因的亚库(见讨论部分)。第一轮的筛选现在就完成了,与起始文库相比,阳性亚库中的基因巳经被富集了·后续的筛选循环是①~⑦步的重复,在扩增的亚库噬菌体定量之后就可以进行。
⑧通过噬菌斑法确定阳性孔中的噬菌体的量( Sambrook and Russell2001)
⑨用约为上一轮数量1/30的噬菌体侵染细菌,开始下一轮的筛选
⑩重复②~⑨步
24.1讨论
对于一个只得到一个阳性克隆的筛选,阳性孔应该在产生阳性信号的行和列的交叉处(图24-1)。对
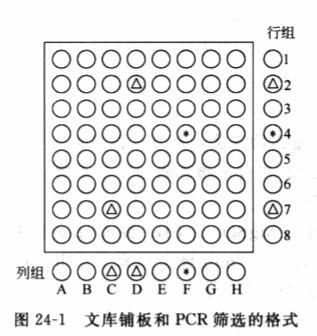

于产生两个或更多阳性克隆的筛选来说,对产生阳性信号的行或列的每个孔中的噬菌体进行第二次PCR,将最终确定阳性孔。在下面的部分和图24-1中将会讨论筛选文库的其他方式
根据特异性PCR产物的量和纯度,琼脂糖凝胶上的结果将足以鉴定出阳性克隆。如果要得到更高的敏感度和特异性,可以按步骤⑦详述的那样,用与PCR引物之间的序列特异配对的放射性或荧光标记的探针,与胶上的PCR产物杂交。这一步也可以不做,特别是在筛选的第二阶段和第三阶段。图24-2(a)是一个琼脂糖凝胶电泳的初步筛选结果,它是通过溴化乙锭的染色才观察到的。因为PCR产物非常复杂,并且特异产物的量很少,需要用杂交来鉴别阳性克隆[图242(b)]。随着第二和第三轮筛选,PCR产
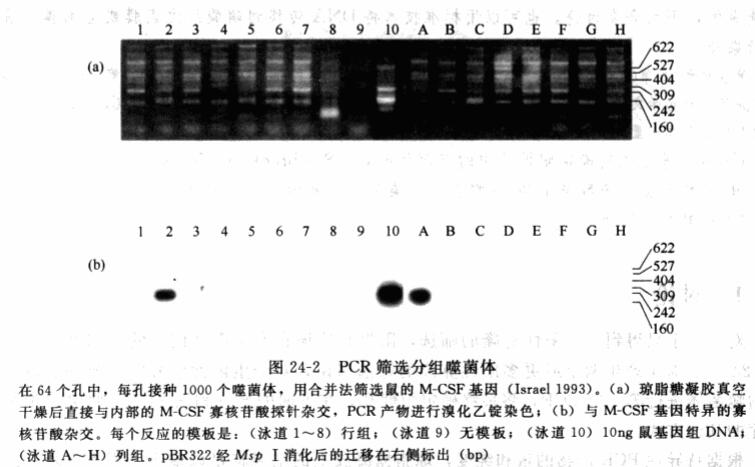
物得到富集,PCR产物的纯度和量增加(比较图242(a)第2泳道和图24-3(a)第9泳道)。这时,杂交就可以不做了。当分离到含有目的基因的纯的噬菌体时,PCR总产物的大部分将是正确的PCR产物(图24-3)
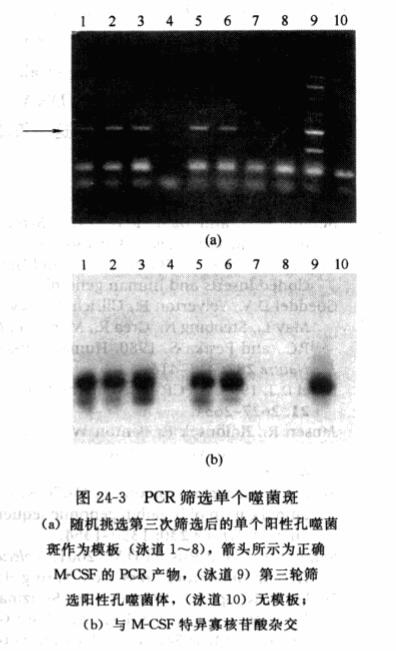
在每一轮筛选之后,基因的丰度就可以估计了,方法可以是上面操作步骤中所提及的阳性亚库的PCR定量法,或者是噬菌斑杂交法。该丰度在每一轮筛选中都会增加,并且可以用来检测富集步骤的效率。一旦基因得到了有效扩增,就可以通过对单个噬菌斑进行PCR或通过噬菌斑杂交的方法得到纯的噬菌斑。
在特定的条件下,可以对该技术进行几种改进,使得克隆更加有效,或满足其他用途的需要。
这些改进如下
24.1.1改变压库的合并模式
用8×8矩阵模式合并,只是一种可以使用该技术的模式。在一个极端的情况下,可以利用每个孔的噬菌体分配噬菌体库以及做PCR。随着实验技术越来越自动化,以及96孔和384孔PCR板及热循环仪的产生,噬菌体的扩增和PCR都可以用多孔移液器和同样规格的板子来进行,这就使得这方法更加可行。但是PCR产物仍然需要用凝胶电泳进行分析,如果不用合并策略,个体样品的数目就会多很多。一个解决的方法就是,对扩增的噬菌体仅按照一个方向合并(也即仅按排或列合并)进行PCR,然后再对阳性亚库每个孔的噬菌体进行第二次PCR。这一方法的优点是分析每个亚库仅需要8个PCR反应,分析单个的孔需要另外8个PCR反应,这样就可以知道阳性样品的精确位置。但是,这种策略需要连续做两次PCR,使得整个过程需要的时间增加了。
24.1.2筛选非噬菌体载体上的基因
DNA文库,尤其是cDNA文库,通常用的是质粒载体。这里所介绍的PCR筛选技术,直用于从一个质粒载体构建的文库中克隆一个cDNA基因( Israel1993)。要筛选质粒文库,含质粒的细菌要在多孔板上长16h,以确保PCR过程中细菌的数目足够多,在PCR反应中可以代表文库中的所有克隆。其他的步骤与噬菌体文库筛选的步骤是一致的。用YAC载体构建的cDNA文库和基因组文库,也可以用类似的PCR进行筛选,只需将YAC进行适当的改进,以便在酵母中可以有效扩增
24.1.3从载体延伸法
当序列信息很有限时,或筛选一个含有靠近插入区一端特定序列的克隆时,其中的一个引物可以与载体的序列互补( Jansen et al.1989)。由于一个PCR引物与文库中的所有噬菌体退火,这种改进将产生大量的不正确的产物。另外,当扩增的序列长度大于2kb时,PCR产物的量将会大大降低。所以,一个特定克隆在库中显示出的丰度将会降低。已经有了关于获得长PCR产物的方法( Cheng et al.1994),这就使得该方法更加可行。
本章介绍了筛选高度复杂的DNA文库的基本PCR程序以及几种改进。如果拥有不错的实验试剂和仔细的实验技术,这一策略应该能稳定地筛选到阳性结果。
|
|
 /3
/3 