金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
基于CRISPR/Cas系统的诊断技术自问世以来,其对无仪器核酸检测技术的需求推动了多种等温扩增技术和快速检测工具的发展。尤其以Cas12和Cas13为代表的单Cas酶反应体系更是获得快速发展,除在基因编辑领域已有诸多应用外,在诊断领域的发展同样迅速。本篇文章主要针对基于CRISPR/Cas技术的不同系统在诊断领域的应用进行简单介绍。
CRISPR/Cas9诊断系统:在基因编辑和诊断领域,Cas9是最早被应用和开发的。2016年,James Collins博士领导的国际小组通过把Cas9和PCR进行结合开发了一种针对寨卡病毒分型的特异性检测方法(图1)[1]。但由于存在着Cas9只能靶向DNA、蛋白体积过大、脱靶效应等方面的不足,同时检测时间长且需要变温反应,该系统在诊断产品开发方向并未得到广泛应用。
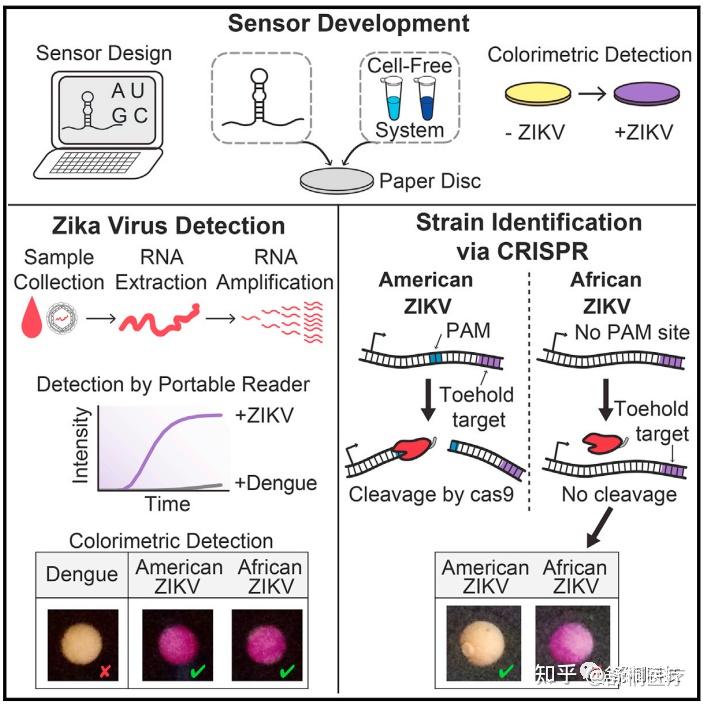
图1 基于Cas9开发的寨卡病毒检测系统
与Cas9不同,Cas12或Cas13除了能够在识别目标序列后启动靶向切割活性,还具有非特异性性切割体系内DNA或RNA的能力(即“反式切割活性”)。利用该反式切割活性,使带有荧光基团的报告分子释放荧光,构成了以Cas12或Cas13反式切割活性为基础的CRISPR/Cas诊断技术:其中以SHERLOCK诊断系统、DETECTR诊断系统、HOLMES诊断系统以及CDetection诊断系统为代表。
SHERLOCK诊断系统:2017年,张峰及其同事提出了一种结合靶基因预扩增技术(RPA或RT-RPA)与Cas13a酶活的诊断平台——SHERLOCK诊断系统(图2)[2]。该系统表现出的单分子检测灵敏度及单碱基的特异性是CRISPR诊断领域中具有里程碑意义的重要工作,然而其只能定性而不能定量检测,同时在扩增与检测之间需要开盖转移易造成气溶胶污染。因此第二代SHERLOCK v2技术增加Csm蛋白和多种Cas蛋白,并将纸基传感器引入系统,使结果可视且更快捷。此外,整个SHERLOCK v2反应一步进行,采用Cas13酶一步法(边扩增边检测)完成整个检测过程。研究证明,SHERLOCK可以检测Zika病毒、登革热病毒、各种致病菌和DNA中的单核苷酸多态性(SNPs)。以该技术为基础开发的新冠检测产品在2020年5月获得了新冠检测的EUA,成为全球第一个注册获批的CRISPR诊断试剂。
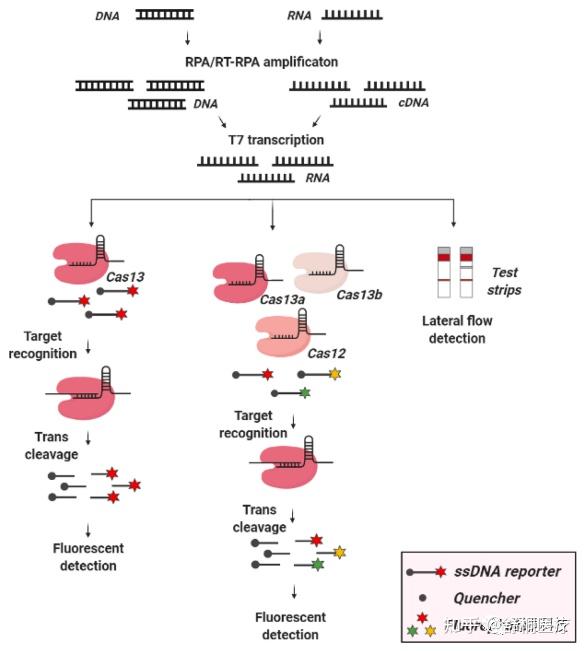
图2 CRISPR-Cas诊断平台的原理图—SHERLOCK/v2
DETECTR诊断系统:Cas12a(Cpf1)是一种CRISPR-Cas突变体,与Cas9类似,它也能切割双链DNA,而Doudna实验室发现了Cas12a切割非特异性(反式)单链DNA的能力,并利用这一能力创建了一个称为DETECTR的DNA检测平台[3]。当靶标核酸存在时,通过恒温扩增(RPA)特定产物,并通过Cas12a和sgRNA复合物进行特异性识别,同时激活Cas12a反式切割活性,将FQ-ssDNA报告探针切割并发出荧光。
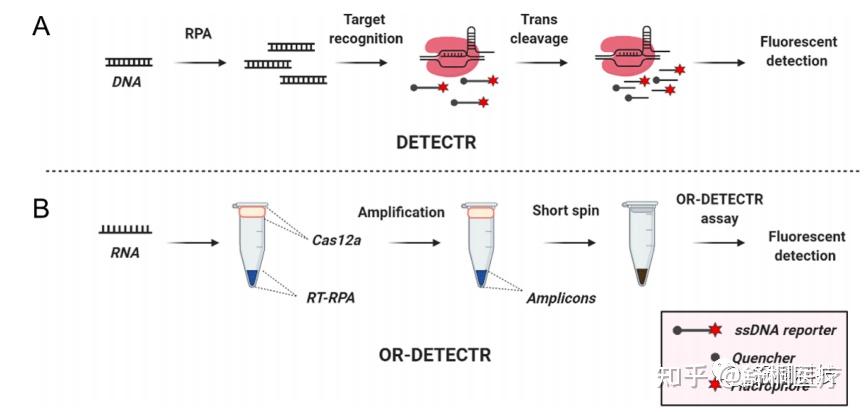
图3 CRISPR-Cas DETECTR和OR-DETECTR诊断平台的原理图
DETECTR系统从流程上更有优势,不仅步骤上更少,而且所用到的探针为ssDNA探针,合成便宜且保存稳定。为了解决两步法的气溶胶污染以及提高检测灵敏度和特异性,同样将恒温扩增与基于Cas12a的CRISPR检测整合在一个系统中,将检测时长从2个小时缩短至30分钟内。
HOLMES诊断系统:同样是利用Cas12a的反式切割活性,国内王金教授团队通过PCR步骤实现模板扩增开发了HOLMES系统(图4)[4-5]。随后,该团队又基于LAMP-Cas12b开发了HOLMES v2系统,并开发了One-Pot一步法CRISPR诊断体系,同时,HOLMES v2可与digital PCR体系联用实现对靶标核酸的绝对定量。测试数据显示,Cas12b的反式切割活性对ssDNA的亲和力要好于dsDNA,具有更宽的反应温度范围,而且灵敏度与Cas12a相当,同时该团队系统优化了Cas12a的反应系统,定义了其反式裂解单位(transU)[6]。在专利方面,王金教授与张峰教授达成了中美市场的专利“交叉授权”合作,共同推进CRISPR诊断产业的快速发展。
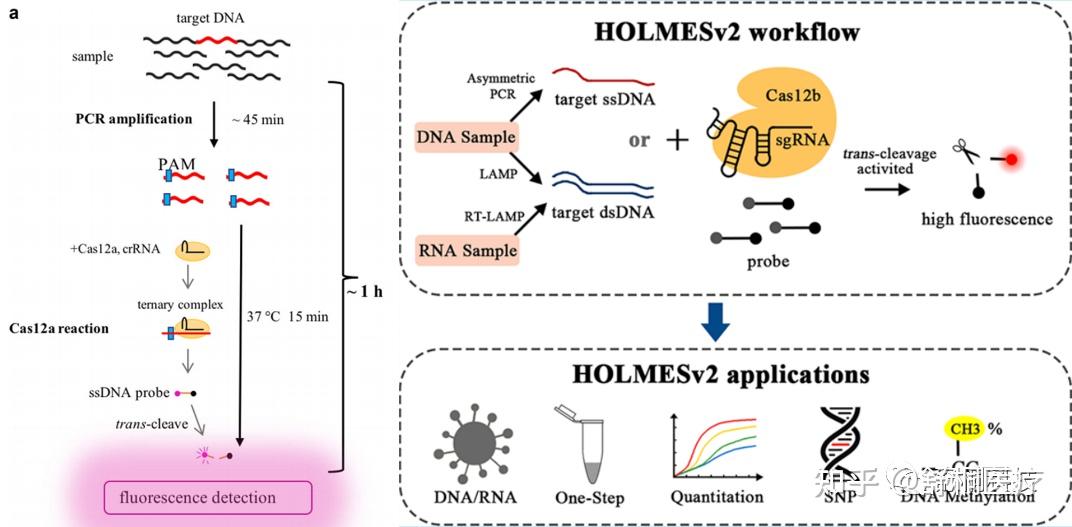
图4 HOLMES/v2诊断系统
CDetection诊断系统:由中国科学院周淇院士团队开发的具有独立知识产权的CDetection诊断系统,采用具有较宽反应温度范围(25-60℃)和pH稳定性(1-8)的AaCas12b,通过AaCas12b-sgRNA复合物特异性识别靶标基因后,激活其反式切割活性,高效的切割体系内ssDNA荧光报告分子,从而发出荧光信号,该系统能够适应不同环境下的检测条件。使用RT-RAA和Cas12b优化系统成分和反应条件,该团队开发了一种Cas12b辅助的one-pot检测平台(CDetection v2),能够在30分钟内快速检测SARS-CoV-2,而不与其他病毒交叉反应[7-8]。
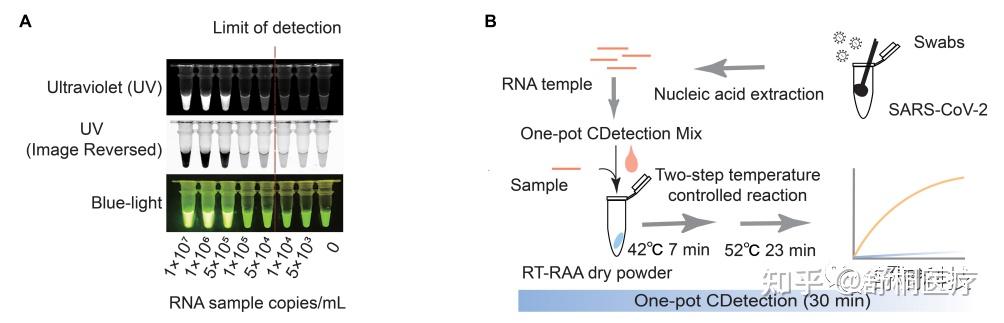
图5 CDetection v2结果的可视化
基于CRISPR的分子诊断技术简单、快速,搭载恒温扩增则无需复杂仪器,不同特性Cas蛋白的发现,丰富了检测核酸类型的选择,从dsDNA到ssDNA再到RNA,均可作为CRISPR/Cas分子诊断系统的靶序列,结合各种可视化反应,为分子即时诊断的应用带来更多发展可能(表1)。
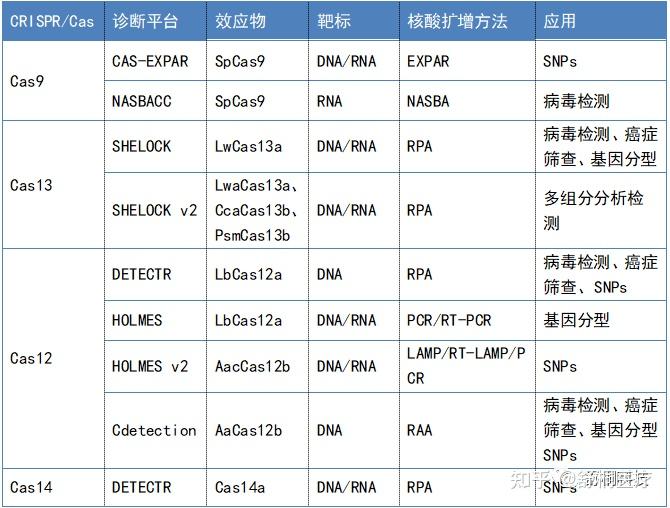
表1 基于CRISPR/Cas系统的诊断技术
珠海舒桐医疗科技有限公司长期致力于基因编辑领域,研发团队潜心研发,具备完善的CRISPR/Cas分子克隆平台以及相关蛋白纯化技术,有着丰富的体外诊断经验以及研发管线。今年2月,舒桐自研高保真CRISPR-FrCas9获国际专利授权,本项专利CRISPR-FrCas9是目前已知的切割效率最强、脱靶效应最低、特异性最好、PAM限制最少的CRISPR,显著优于SpCas9。此外,珠海舒桐医疗是国内首家提供脱靶检测服务的公司,可提供GUIDE-seq体内脱靶检测、AID-seq体内脱靶检测等多种服务,欢迎垂询。
关于舒桐
珠海舒桐医疗科技有限公司深耕基因编辑技术10余年,是一家以基因编辑为核心技术的生物医药服务企业,可承接脱靶检测、细胞株定制、病毒包装等CRO及CDMO业务。目前,舒桐科技拥有3000㎡的GMP研发实验室及生产车间;拥有博士后科研工作站及澳门理工大学教学科研实习基地。研发团队熟练掌握各类基因编辑技术,包括基因编辑、碱基编辑及先导编辑等,成功完成如基因敲除、点突变、大片段插入、过表达等细胞系构建项目;开发了多种具有自主知识产权的脱靶检测技术,建立了高通量的sgRNA筛选平台;建立了工厂化新型CRISPR挖掘体系,已开发出多种切割效率高且低脱靶的新型CRISPR。公司目前已申请基因编辑、脱靶检测、新型CRISPR及药物递送等专利70+项,授权50+项。同时,公司具备先进的CGT原料规模化生产能力,产品质量达到领先水平,可为科研机构及生物医药企业提供安全高效的一站式服务和原料供应。舒桐科技坚持以“创新、敬业、融合、开放”为核心价值观,致力于利用基因治疗技术造福人类健康,让生命更美好。
如需获得更多信息,请咨询我们:
电话:
400-6309596
15022705442(同微信)
企业官网:
http://www.generulor.com
产品订购/技术支持:
service@generulor.com
参考文献:
[1] Pardee K, Green AA, Takahashi MK, et al. Rapid, Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 2016 May 19;165(5):1255-1266.
[2] Gootenberg JS, Abudayyeh OO, Lee JW, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 2017 Apr 28;356(6336):438-442.
[3] Chen JS, Ma E, Harrington LB, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 2018 Apr 27;360(6387):436-439.
[4] Li SY, Cheng QX, Wang JM, et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discov. 2018 Apr 24;4:20.
[5] Li L, Li S, Wu N, et al. HOLMESv2: A CRISPR-Cas12b-Assisted Platform for Nucleic Acid Detection and DNA Methylation Quantitation. ACS Synth Biol. 2019 Oct 18;8(10):2228-2237.
[6] Lv H, Wang J, Zhang J, et al. Definition of CRISPR Cas12a Trans-Cleavage Units to Facilitate CRISPR Diagnostics. Front Microbiol. 2021 Nov 29;12:766464.
[7] Teng F, Guo L, Cui T, et al. CDetection: CRISPR-Cas12b-based DNA detection with sub-attomolar sensitivity and single-base specificity. Genome Biol. 2019 Jul 1;20(1):132.
[8] Wang X, Chen Y, Cheng X, et al. CDetection.v2: One-pot assay for the detection of SARS-CoV-2. Front Microbiol. 2023 Mar 23;14:1158163. |
|
 /3
/3 