登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
绝大部分单细胞转录组课题都是想看已知的重要的单细胞亚群是否有比例变化或者说是否有通路激活或者抑制情况,把它们来跟表型联合起来。
而稍微看几个自己领域内的单细胞文献就知道已知的单细胞亚群主要是什么了,比如我前面介绍过 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的fibo 和endo进行细分,并且编造生物学故事的。
有生物学意义的单细胞亚群如果是有十几个,就会面临一个问题,大家的比例相差悬殊,尤其是肿瘤里面的淋巴细胞,尤其是b和t细胞浸润非常严重,这样的话如果我们想研究微环境里面的基质细胞比如成纤维和内皮细胞就会面临细胞数量不足的困境。那么,这个时候预先把样品进行单细胞流式细胞仪筛选就是一个很好的策略,比如2021发在NC的文章:《Investigating immune and non-immune cell interactions in head and neck tumors by single-cell RNA sequencing》,就是18个 头颈癌Head and neck squamous cell carcinoma (HNSCC) 病人的单细胞转录组数据集,数据集链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE164690
可以看到每个病人都会有多个10x的单细胞转录组数据 :
GSM5017021 HN01_PBL cells
GSM5017022 HN01_CD45+ cells
GSM5017023 HN01_CD45- cells取样策略如下所示:
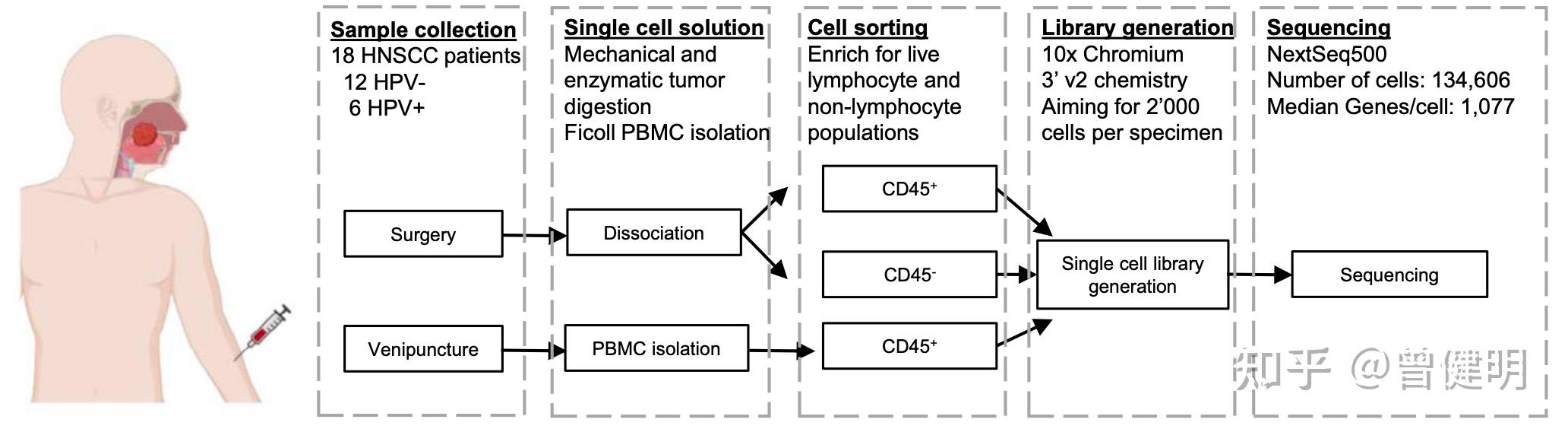
取样策略
每个病人都会有肿瘤部位,样品经过了CD45的筛选后区分成为两份去做单细胞转录组,少部分病人也抽血液做了PBMC单细胞转录组。
如果我们这个时候下载其表达量矩阵文件,GSE164690_RAW.tar 有点大,是718.2 Mb,把它拆分成为了3个单细胞转录组项目,各自独立降维聚类分群就会发现其实并不是很纯粹的免疫细胞和非免疫细胞。
首先看CD45+ cells,理论上都应该是免疫细胞,但是很明显的是里面确实有少量上皮细胞和基质细胞 ;
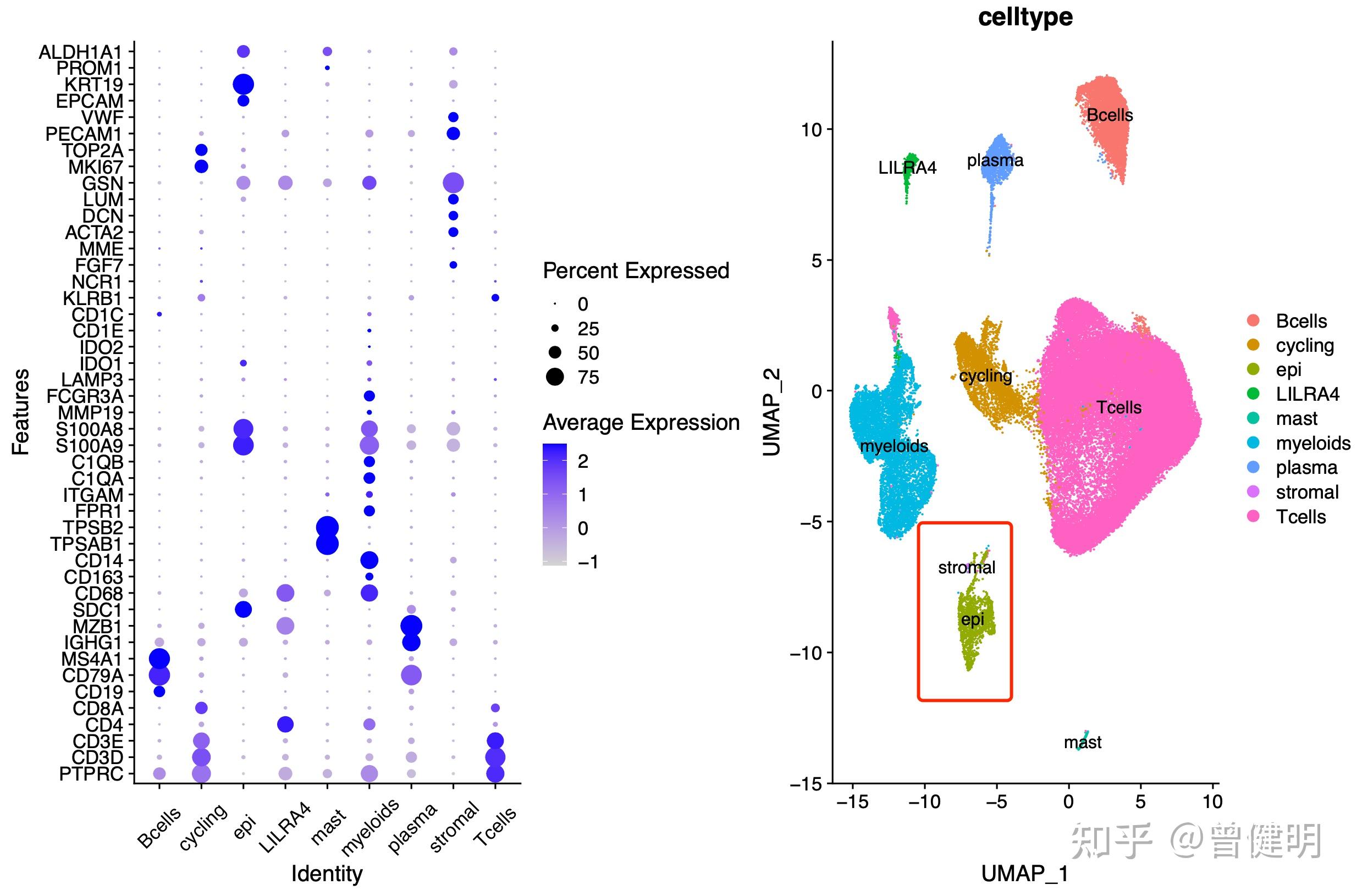
理论上都应该是免疫细胞
从这个单细胞转录组里面的mRNA的表达量来看,不小心混入在里面的上皮细胞确实并不会表达CD45,但是为什么流式细胞筛选就被混入了呢?
让我们再看看 CD45- cells ,就更严重的混入了:
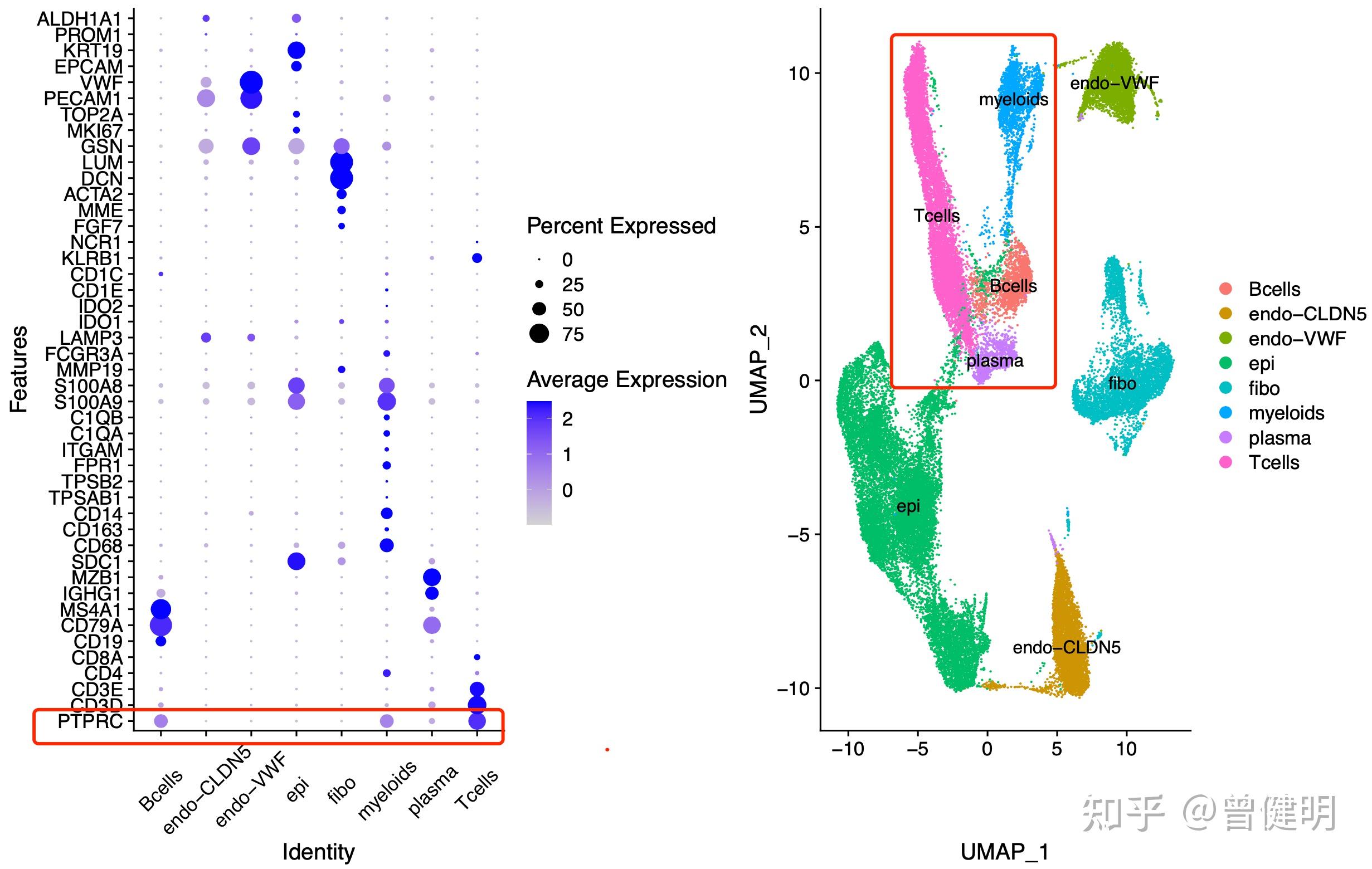
更严重的混入了
我们可以独立计算一下每个样品的混入情况,以及整体的混入情况,这样大家有一个大概的认知,后续再遇到这样的流式细胞筛选过的数据集也不要担心。
首先让我们看看免疫细胞里面的混入情况:

image-20220925203625215
发现,其实免疫细胞的筛选并没有问题,仅仅是因为其中一个病人HN17的混入,假如拿到作者原始的实验数据,其实就可以去看其流式细胞图片,是不是这个病人实验环节有错误啦。
再看看非免疫细胞里面的混入情况:
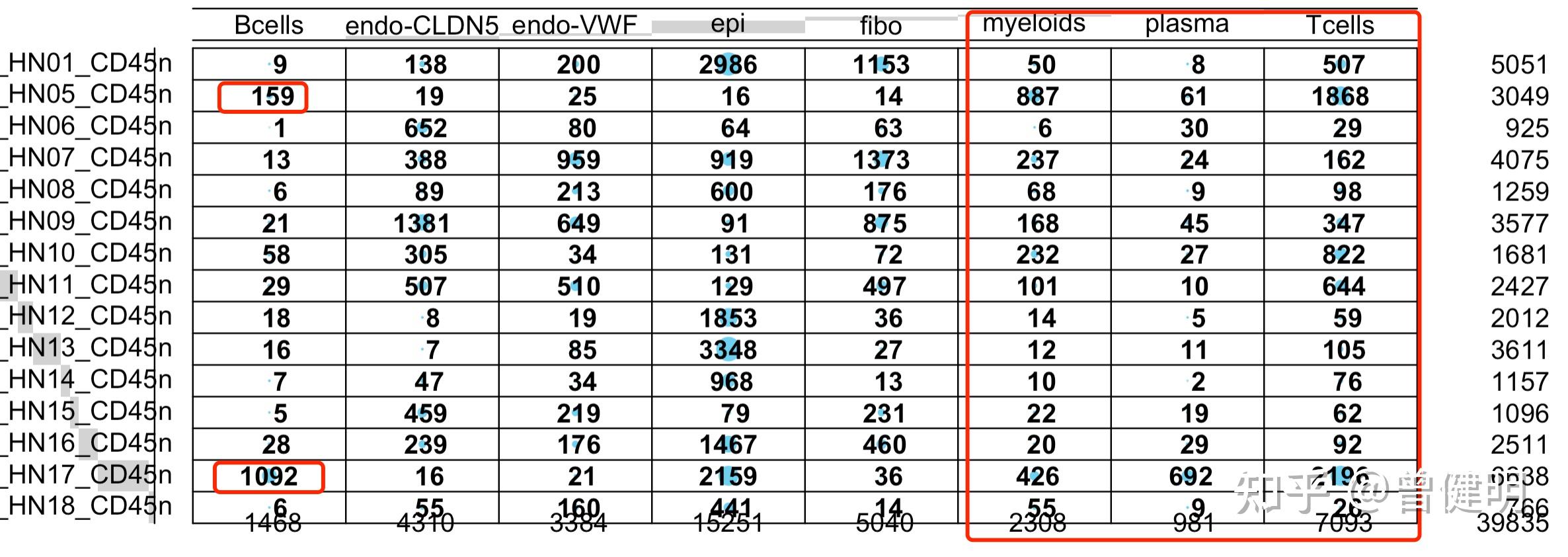
非免疫细胞里面的混入情况
可以看到这个HN17仍然是有问题,与此同时,HN05问题也不小哦。
当然了,我们仅仅是拿其中一个文章来举例说明了这个流式细胞筛选不能达到百分百的纯,并不是一个正经的学术研究,如果大家感兴趣把它发展成为一个课题,也可以联系我哈。需要具备基本上单细胞转录组数据处理建议,到降维聚类分群即可。
而且需要有基本的R可视化能力哦,因为总是有人嫌弃seurat标准流程的默认出图并不好看,详见以前我们做的投票:可视化单细胞亚群的标记基因的5个方法,下面的5个基础函数相信大家都是已经烂熟于心了:
- VlnPlot(pbmc, features = c("MS4A1", "CD79A"))
- FeaturePlot(pbmc, features = c("MS4A1", "CD79A"))
- RidgePlot(pbmc, features = c("MS4A1", "CD79A"), ncol = 1)
- DotPlot(pbmc, features = unique(features)) + RotatedAxis()
- DoHeatmap(subset(pbmc, downsample = 100), features = features, size = 3)
常规的降维聚类分群,参考前面的例子:人人都能学会的单细胞聚类分群注释 ,走seurat流程进行单细胞降维聚类分群,这样的基础分析,有基础10讲:
原文地址:https://zhuanlan.zhihu.com/p/665539955 |
 /3
/3 