金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
CRISPR技术虽然是现阶段生物界非常新而且非常热门的一门技术, 在各大门户网站上有很多文章已经对于此项技术进行了详细的介绍. 但是本篇文章本着简洁, 通俗易懂的理念, 将细致的介绍CRISPR的相关概念并且阐述作者自身对于CRISPR这门技术的理解. 适合刚刚接触CRISPR系统的人进行学习探讨.
在本文中, 我将介绍什么是CRISPR 系统, 其结构特点和作用机制. 同时也会概括性的介绍CRISPR 在现阶段的应用, 和gene drive(基因驱动) 技术.
(1) CRISPR/Cas: 强大的基因组编辑工具
精准, 简便的对于基因或者基因组的信息进行编辑对于生物学家了解生物体内的代谢过程, 生物体内基因的功能研究和现代基因治疗都有着至关重要的作用. 之前的基因组编辑技术, 例如锌指蛋白 (Zinc Finger, ZFNs)和转录激活因子样效应物
(Transcription activator-like effector, TALENs) 对于基因组编辑的发展做出了不可磨灭的贡献. 在这两种技术当中, 基因编辑的工具在基因组上的定位依靠蛋白质与DNA之间的相互反应, 而对于基因组的剪切过程依靠Fokl蛋白. 图一中, 彩色的圆形或椭圆形代表了合成的锚定蛋白 (Fig.1). 但是锚定过程依靠DNA 与蛋白质相互作用的一个显著的缺点就是当要锚定新的基因位置时,需要对于锚定蛋白进行重新设计和合成, 这大大的加大了基因编辑的工作量和难度, 使这些技术较难适应高通量的基因组编辑工程.
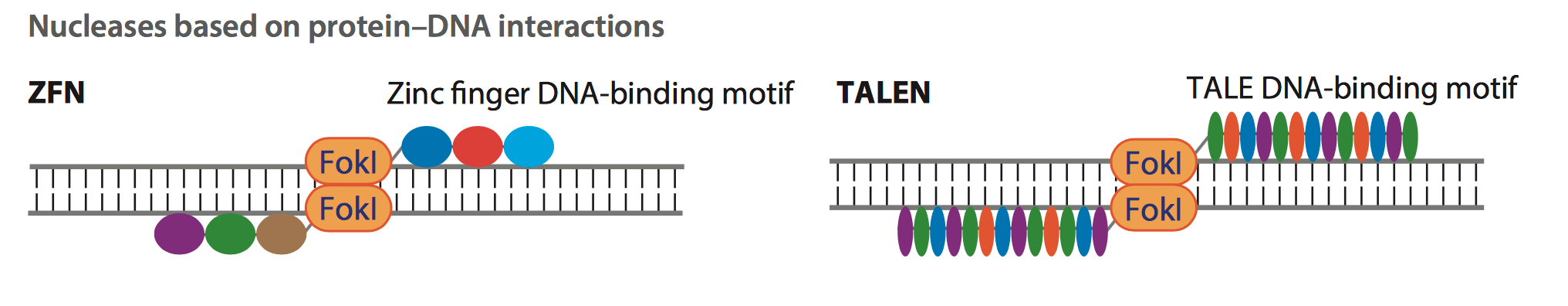
Figure 1: Zinc Finger, ZFNs and Transcription activator-like effector, TALENs) technologies.
CRISPR/Cas系统作为一个革新性的强大基因组编辑工具的出现改变了这一境况, 可以说是颠覆了基因组编辑这个领域. CRISPR的全名是 (clustered regularly interspaced short palindromic repeats), 中文意思是成簇的, 规律间隔的,短回文重复序列. Cas 所指的是
(CRISPR-associated). 与上述的工具之间的不同之处是, CRISPR/Cas系统对于基因组上基因的定位利用RNA 与DNA 之间的相互作用, 对于新基因位置的锚定只需要一小段新的RNA序列, 这个特点大大的减少了新和成蛋白的工作量, 且特异性是很高的. 具体的机制在下文会有介绍. CRISPR/Cas系统之所以成为基因组编辑界的新宠是因为这个工具具有很多其他工具不具备的优点, 例如, 合成简便, 使用方便, 低费用,特异性高等.
(2) CRISPR/Cas系统的结构
CRISPR/Cas系统是细菌体内的获得性免疫系统, 是用来对抗侵略细菌的外源DNA, 质粒和噬菌体的. 与普通的原核生物的general的免疫系统不一样, CRISPR/Cas系统是获得性免疫系统, 这就意味着这个免疫系统是具备“记忆性”的. 他可以记住入侵过的外源DNA 和噬菌体, 并在他们再次入侵的时候切断他们基因组, 被切断的基因组将变为线性,无法进行复制和表达,并被细菌体内的酶降解掉. 这个免疫系统虽说简单, 但是却非常实用且强大.
CRISPR 系统是一个什么样的结构呢. 简而言之, CRISPR/Cas系统由两大部分组成: 第一个部分是编码Cas相关蛋白质的基因(在Fig.2中的白色方块箭头), 这些Cas蛋白在获得外源基因片段和剪切外源基因上都起着重要的作用. 第二大部分被称为CRISPR array, 这个部分中包含了repeat序列和spacer序列, 这两种不同的序列是间隔开来的, 本着两个repeat序列夹一个spacer序列, 正如Fig.2所示, 黑色菱形代表repeat序列, 不同颜色的方形则代表了不同的spacer序列. Repeat序列在同一细菌中的碱基组成和长度是相对保守的, 基本不变. 在不同的细菌之间会有些许差异. Spacer序列则是用来锚定目的外源基因的, 所以spacer的序列碱基组成差异较大, 因为他们来自于不同的外源基因. Spacer 基因当中包含着被锚定基因组中的特异性高的保守序列, 确保在之后转录出的RNA 可以与被锚定基因组精确配对. CRISPR array之前通常会有一个富含A-T的leader sequence, 这个序列中包含启动子, 是用来启动repeat 和spacer序列转录的. CRISPR array 不包含阅读框(ORF, open reading frame).
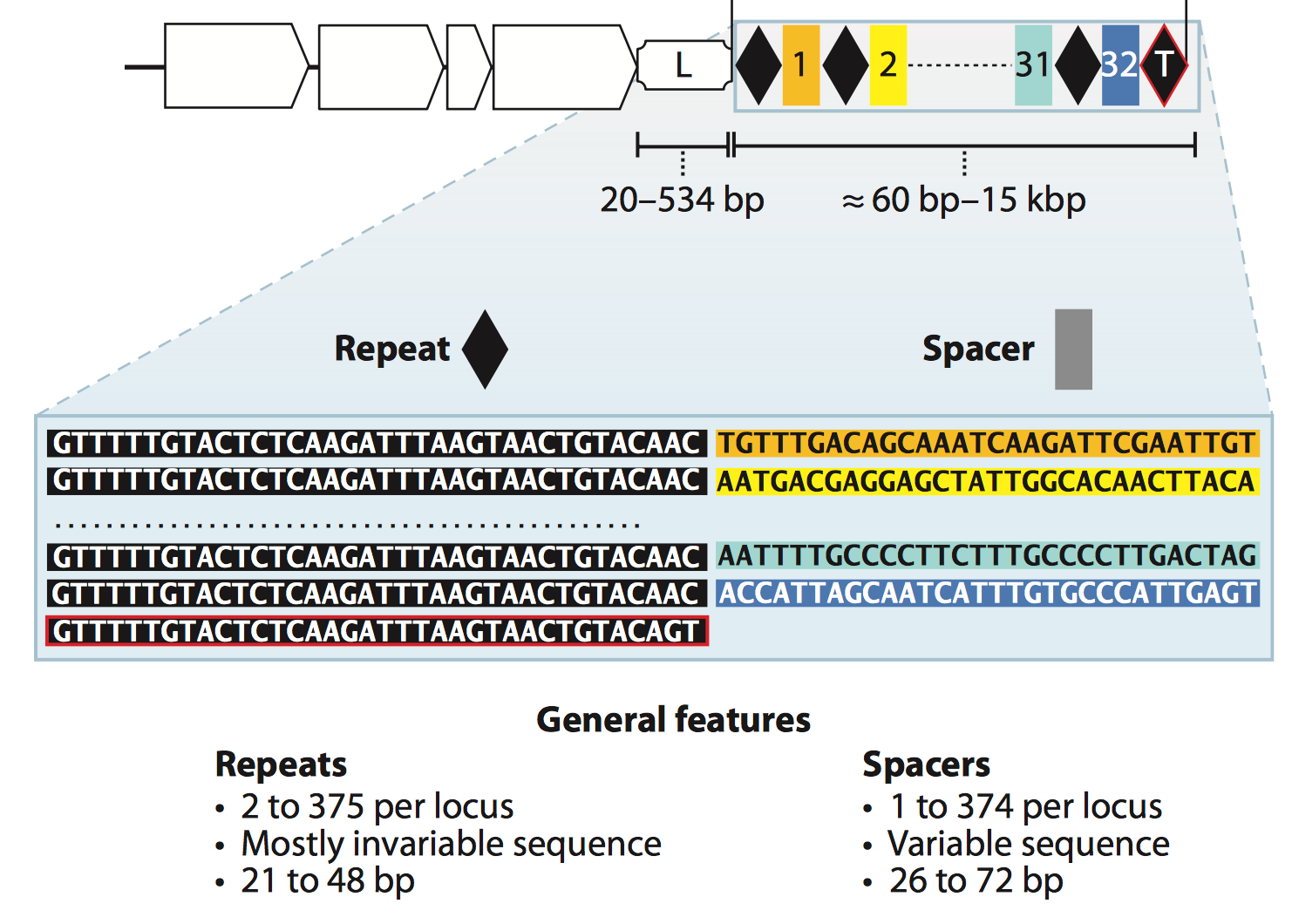
Figure 2. General CRISPR/Cas system locus.
(3) CRISPR/Cas 系统的作用机制
CRISPR/Cas的作用机制可以分为两个主要的部分: Adaptation和Interference. 第一个大部分Adaptation根据我的理解可以又分为两个小部分, 分别是Spacer序列的获取和CRISPR RNA
(CrRNA)的合成加工. 下面对于作用机制的解释基于Fig.3.
1. Adaptation: Spacer的获得
当噬菌体或者外源基因侵入到细菌体内后, 其基因组中的protospacer会被CRISPR/Cas系统中的cas相关基因进行识别而剪切. Cas蛋白对于spacer的识别获取基于其序列下游的PAM (Protospacer adjacent motif) 序列, PAM 序列在spacer获取和CRISPR系统的体外设计中都起着至关重要的作用. 不同的CRISPR/Cas系统的PAM识别序列也是不同的. 当Cas相关的蛋白选择spacer后, 会把其基因剪切下来, 并插入到leader序列和相邻的repeat的中间, 形成新的spacer. 这样, 下次同样的外源基因入侵时, 就可以对其基因组进行剪切了. Spacer的前面是需要有repeat的, 这和后面形成城成熟的CRISPR RNA 很重要. 为了不影响读者对于机制一个大概的理解, 新插入的spacer前是如何形成新的repeat序列的, 作者将在Appendix1中作介绍.
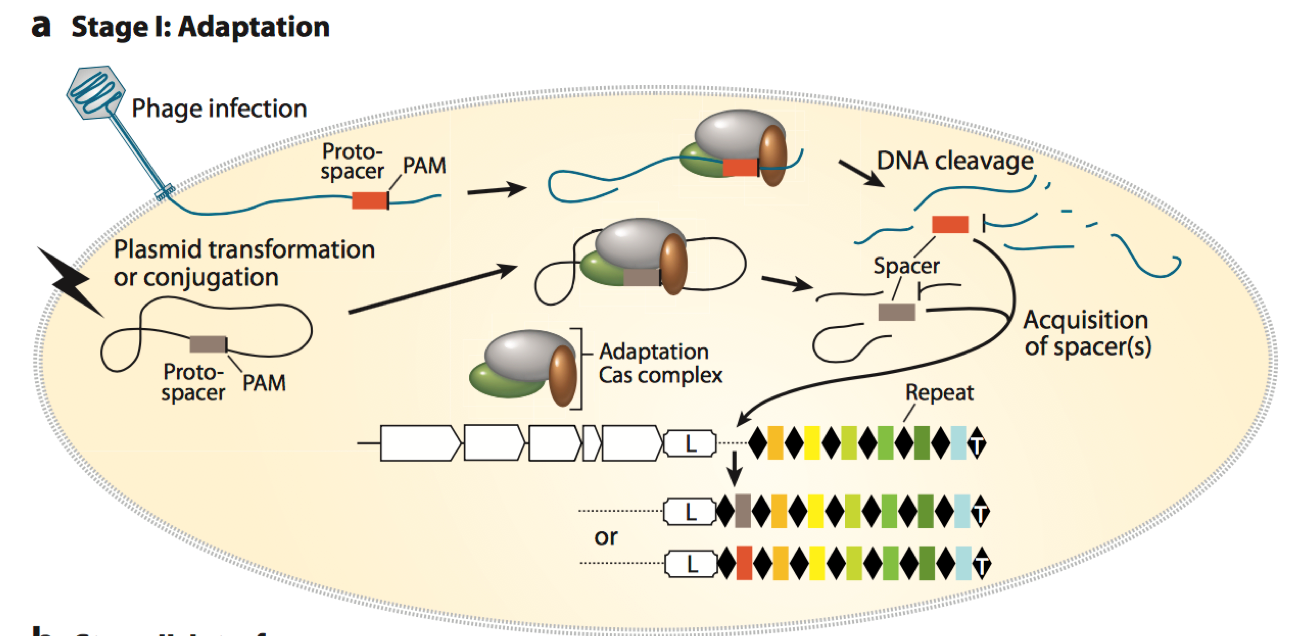
Figure 3. Adaptation process, including spacers acquisition and crRNA biogenesis.
2. Adaptation: CRISPR RNA (CrRNA) 的形成
之前提到过, leader sequence中具有启动子, 可以启动后面CRISPR array的转录, 这个转录是连续的. 因此转录出的RNA 产物是一条长链, 其中包含了CRISPR array中所有的spacers和repeats, 这条长链RNA 被称为precursor transcript (pre-crRNA). 如Fig.4所示, 长链pre-crRNA会随之被细菌体内的管家基因表达的酶或者Cas相关的蛋白(取决于CRISPR系统的差异)所加工剪切, 使之称为成熟的, 含单一spacer的crRNA. 转录出来的spacer RNA 序列是和目的锚定基因互补的, crRNA 可以引导Cas相关的蛋白去剪切目的基因组中的基因.
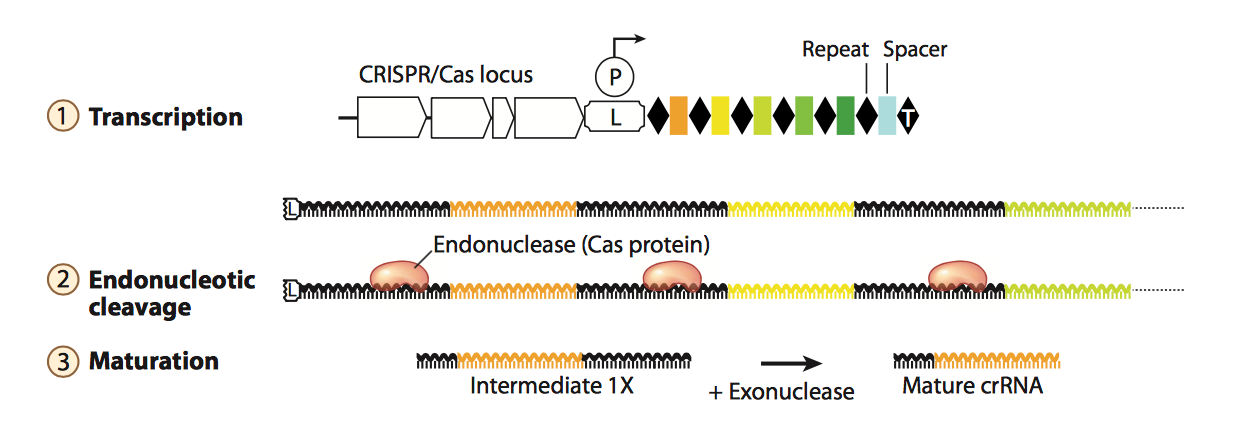
Figure 4. crRNA biogenesis
3. Stage II: interference
在成熟的单一spacer的crRNA形成之后, 其会与Cas相关蛋白和其他的RNA 组分组成一个复合物, crRNA 可以与外源基因中的基因互补配对, 并引导Cas蛋白或蛋白复合物对外源基因片段进行剪切. 正如fig.5和fig.6所示. crRNA和Cas相关蛋白质组成的复合物是根据不同种类 CRISPR系统而不一样的. 在最常用的type II系统中, crRNA会与noncoding trans-activating CRISPR RNA (tracrRNA)互补配对再与Cas9蛋白形成复合物进行DNA 剪切. 这个在后面会介绍.
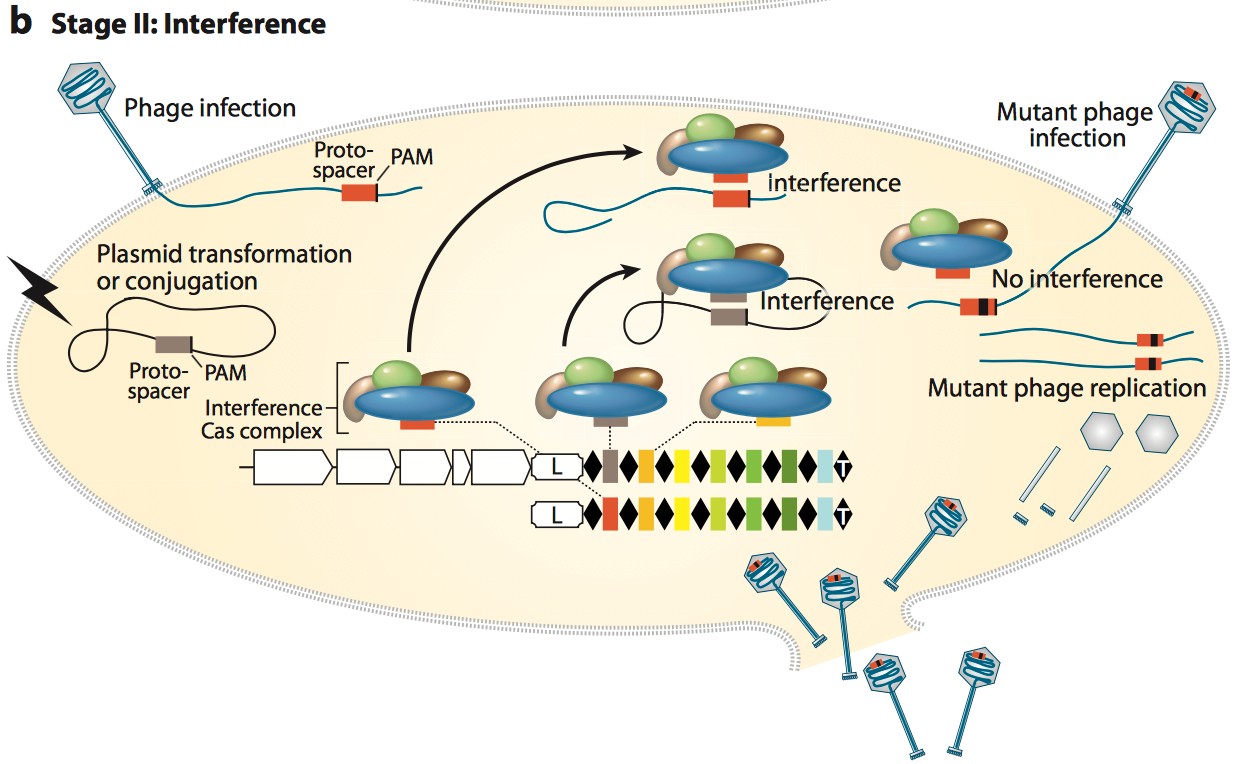
Figure 5. Stage II interference.
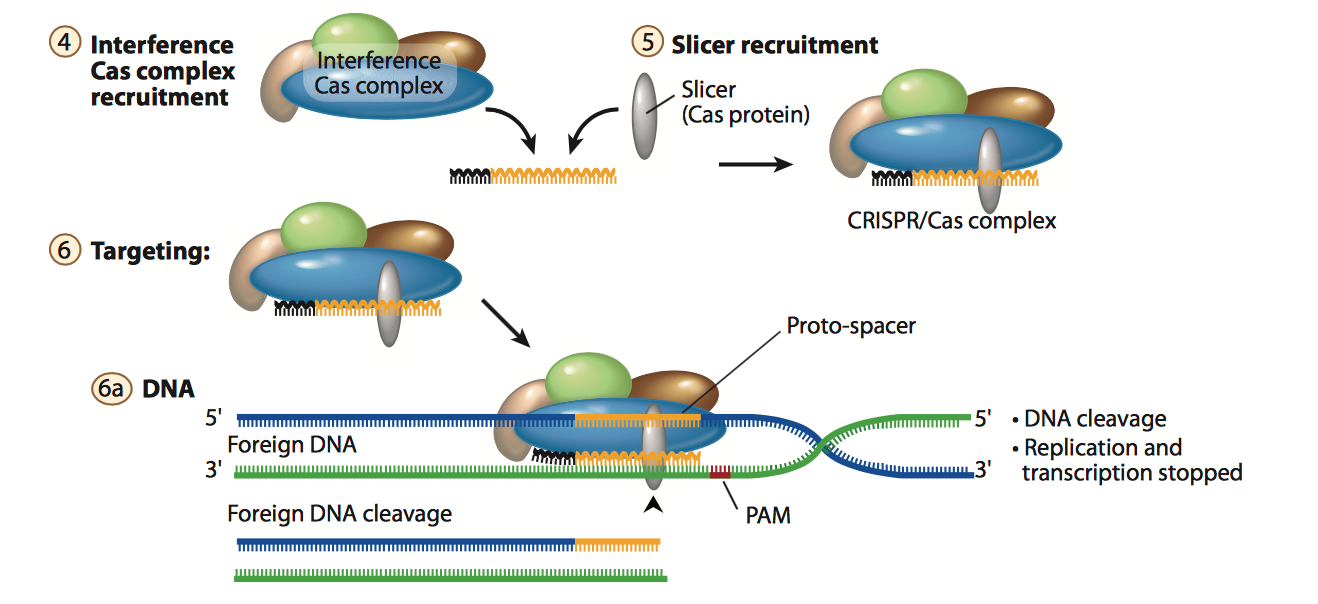
Figure 6. schematic of interference stage.
(4) 不同的CRISPR 系统
CRISPR/Cas系统大体可以被分为两类, Class 1 (包含了type I, III, and IV), 和Class 2(包含了type II 和type V和type VI). 在Class1 中, 对于外源基因组的剪切需要一个大的Cas蛋白复合物(由不止一种Cas蛋白组成)和引导RNA. 在Class2中, 对于外源基因的剪切只需要一个单一的剪切蛋白, 例如 TypeII中的Cas9蛋白和TypeV中的cpf1蛋白. Fig.7中清晰的表明了在何种阶段, 有何种Cas相关的蛋白参与.
在人工合成Cas系统时, 最常用的是Class2 CRISPR 系统, 因为对于DNA 的剪切只需要单一的Cas9 蛋白或者cpf1 蛋白, 非常简单便捷.
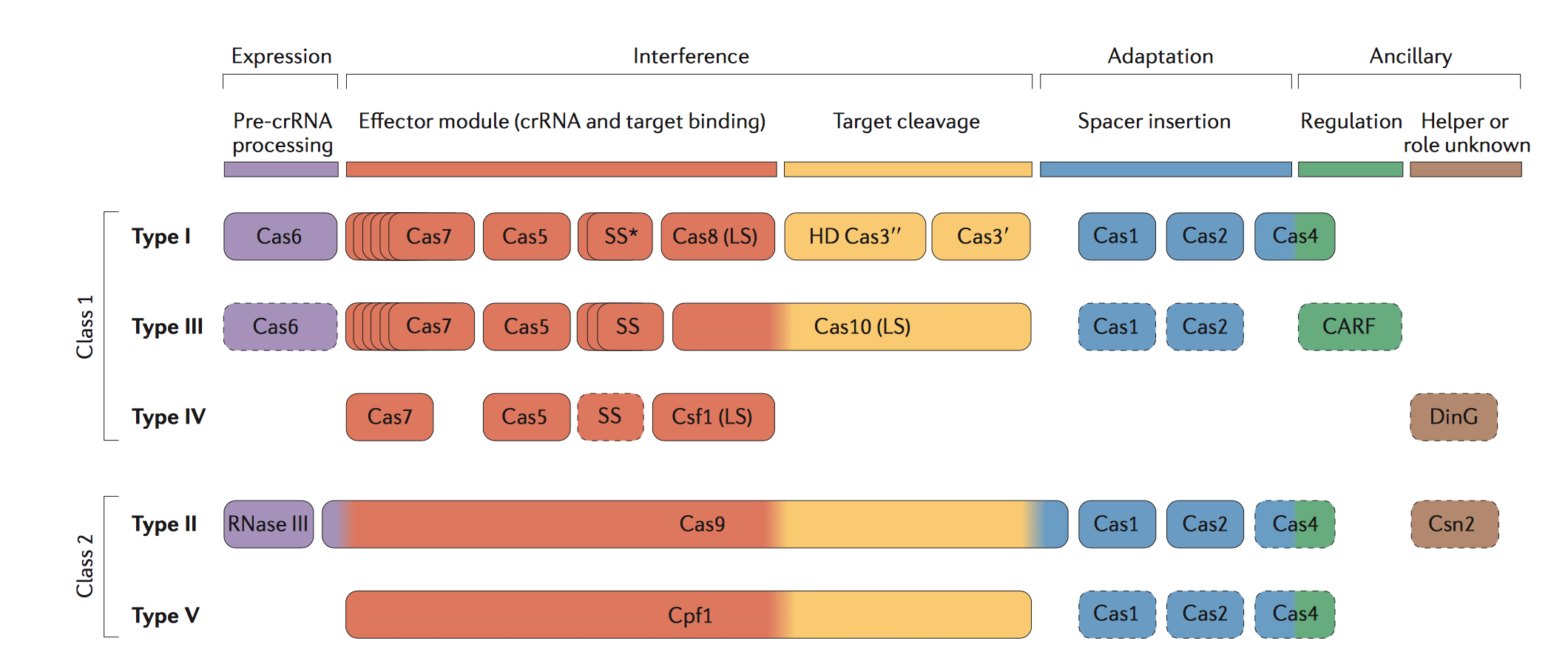
Figure 7. functional classifications of Cas proteins.
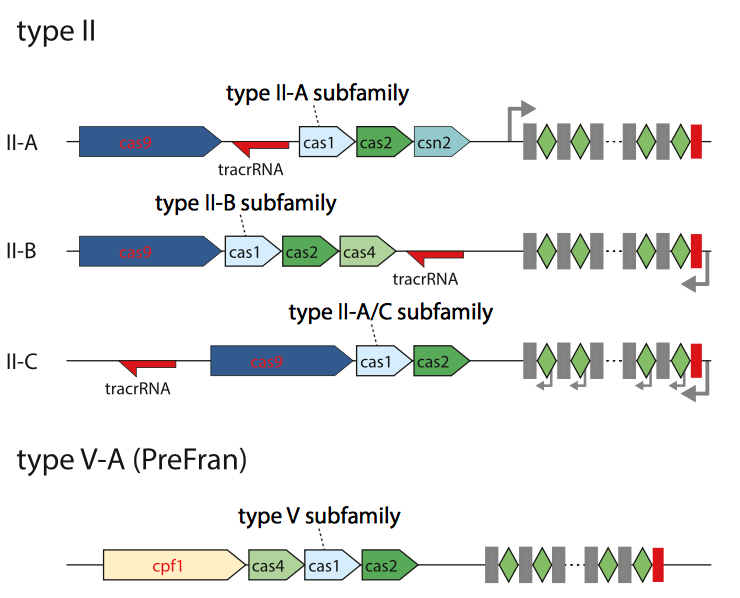
Figure 8. Genomic architectures of the known and newly identified Class 2 CRISPR-Cas systems.
Fig.8 示意了type II 中的三个subtype和Type V中CRISPR 系统的基因结构. 各中Cas蛋白的作用可以对照Fig.7去看. 其中的tracrRNA在上一节中有所介绍. 在对DNA 进行剪切的时候tracrRNA 会和CrRNA配对, 形成guided RNA引导Cas9去剪切DNA. 其中详细的结构示意图在Appendix2中显示.
(5) Type II CRISPR 系统体外的合成策略和应用原理
第三章节中介绍的CRISPR作用机制是细菌体内的作用机制, 是应用CRISPR技术的基础. CRISPR 之所以被称为是一个强大的基因组编辑工作是因为他操作简单, 合成也非常的便捷. 上面提到了TypeII CRISPR 系统是最常用的系统. 那么在这一节中我就以type系统为例介绍CRISPR的应用.
首先我们先看Fig.9, 这是一个非常清晰的示意图. 在实践应用中, 我们会先选择需要进行锚定剪切的基因, 基因的选择要基于PAM 序列, 之前介绍过. 那么type II 的PAM 序列就是NGG. 也就是说, 在目的剪切的基因上, 与剪切的基因片段后必须要有一段PAM序列, 这对后面Cas9的识别和剪切是必要的. 再选择好欲剪切的基因片段后, crRNA则是被选择的这一片段. 随后tracrRNA是必须的, 因为其要和crRNA形成特殊结构后才可以引导Cas9蛋白去剪切. 由Appendix2可以看出, tracrRNA与crRNA配对的部分是repeat基因, 所以在实际应用中, 一个tracrRNA就可以和不同的crRNA (包含目的基因的protospacer和repeat序列)相配对了. 而sgRNA (single-guided RNA) 是体外人工合成的, 可以引导Cas9区剪切目的基因.
Ps: 在实际操作用, 可以不需要linker loop. (如Appendix 2 所示), 因为如果使用linker loop, 对于有每一个spacer都要转录一次tracrRNA. 如果一个系统中有多个spacers, 则按照Fig.8 的基因结构合成即可.
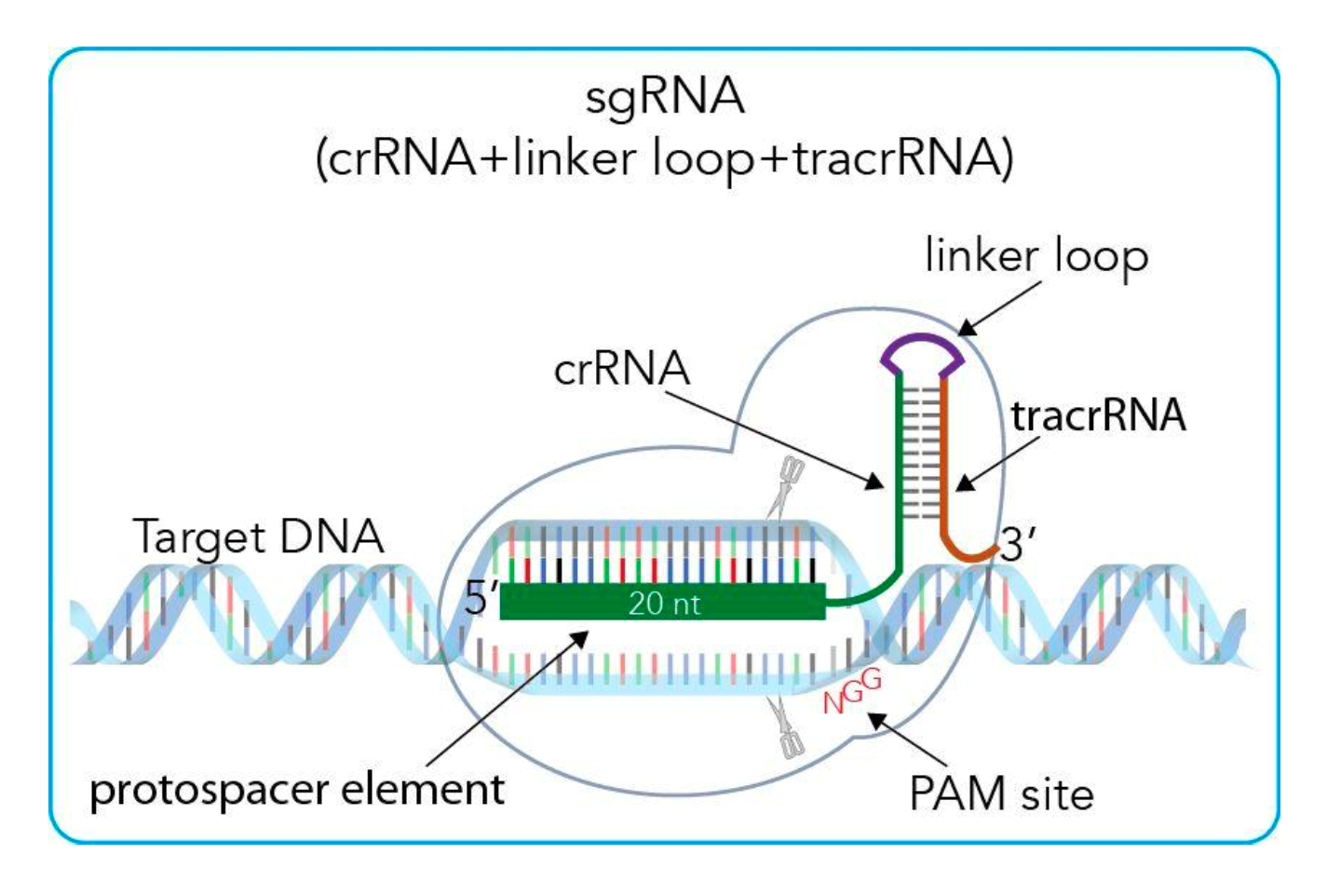
Figure 9: Schematic of artificial type II CRISPR systems
那么当把基因剪断了之后, 该怎么办呢? Cas9蛋白可以引发DNA 的双键断裂, Cas9中包含了HNH , RuvC-like 和PAM序列交流活性区. HNH 和RuvC会各切DNA 的一条链, 造成双键断裂. 正如Fig.10所示. 如果对HNH和RuvC活性区域进行突变使之无法行驶剪切的功能的话就会产生Dead Cas9 (dCas9). dCas9 只可以提议性的附着到特定的基因位置上 (依据sgRNA 上的序列), 而不可以行驶剪切功能. dCas9在CRISPR的各类应用中起着很重要的作用.还有一种方法就是只对HNH或RuvC的其中一个位点进行沉默突变, 形成和Cas9 nickase, 这样就可以用两个不同的nickase对基因组进行锚定和单链剪切, 造成粘性末端.
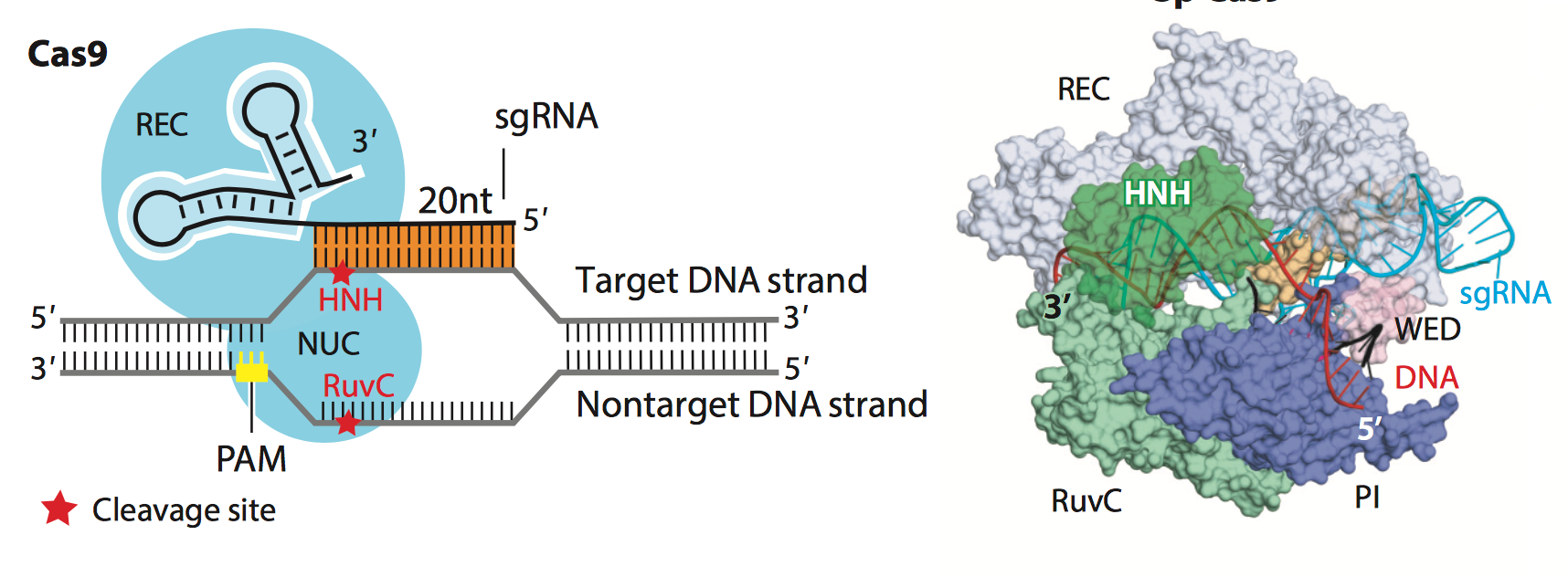
Figure 10. Schematic of main domains of Cas9 protein.
如Fig.10 所示, 单一的CRISPR系统可以造成平末端的DSB (double
stranded break), 两个Cas9 nickase 系统可以造成粘性末端的断裂. 当DNA 的双链被剪切了之后, 有两种修复方式: 第一种就是NHEJ, 不同源的末端修复. 这是生物体内自发的SOS修复, 紧急连接断裂的DNA 双链, 但这种连接方法是随机的, 油价可能造成碱基的插入, 删除, 造成阅读框的移码. 而另一种是HDR, 同源定向修复, 提供一个两端序列和断裂序列相同的donor DNA小片段, 这个DNA小片段可以与断裂的基因进行同源重组, 由此形成目的性的插入基因, 删除基因等功能 (红色标出的片段是目的插入片段). 由此可以完成基因组中基因的定向改变, 插入和删除.
Fig.10中的第三个图则是运用了dCas9 (dead Cas9). 并在Cas9蛋白上附着上FokI 酶, 这样两个dead cas9 就可以实现基因的定位而不可能剪切, 而FokI 酶行驶剪切效用. 这样的双锚定功能会大大较低CRISPR 系统的脱靶效应 (off-target).
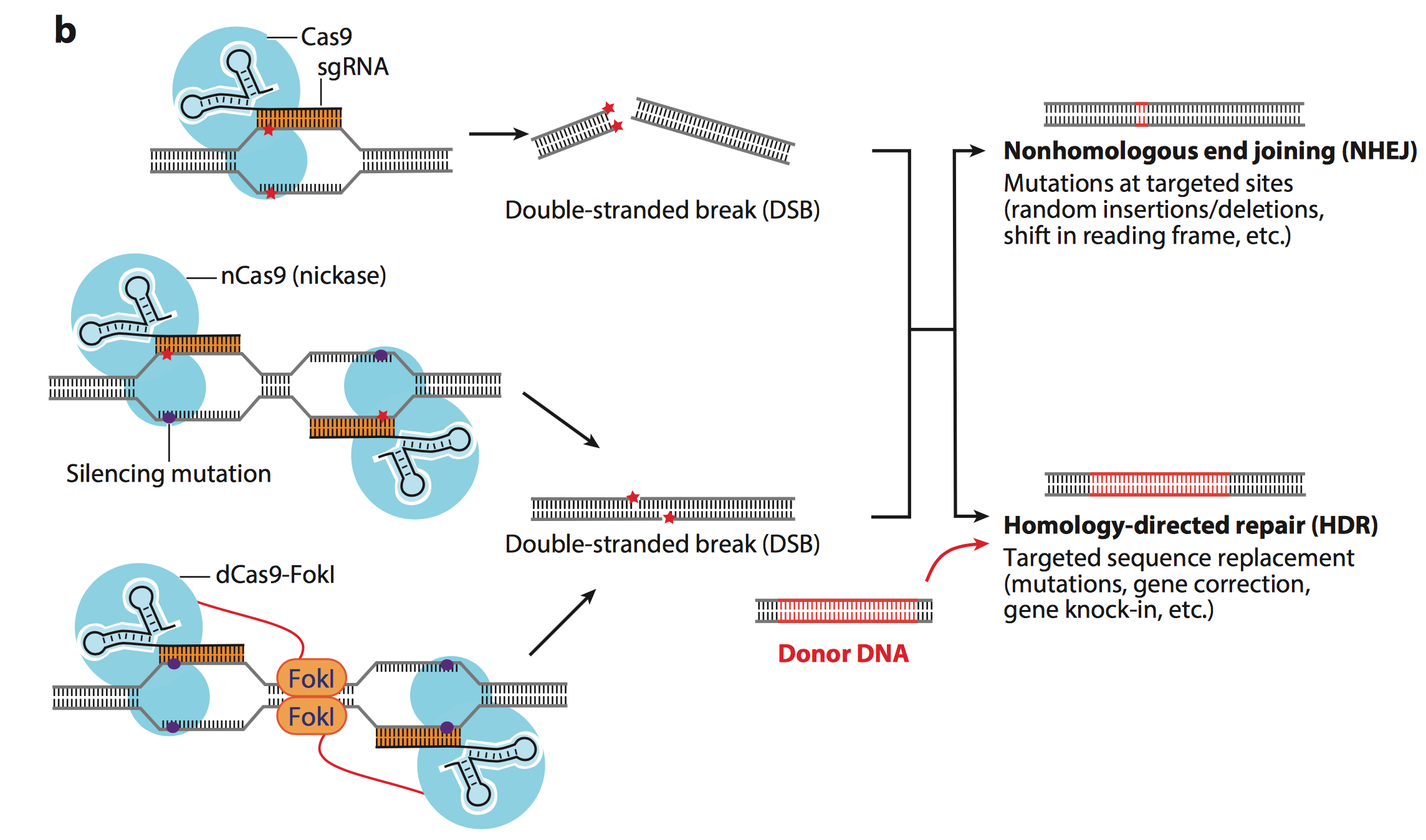
Figure 11. Cas9 in genomic editing.
(6) CRISPR系统的不同应用
CRISPR系统最先的用途就是造成DNA 的双键断裂, 在使用NHEJ 或者HDR的方式进行定性,目的基因的编辑. 但很快, CRISPR系统就应用到了众多领域. 例如激活或抑制转录反应. 下面以Fig.12 为例做介绍. 图A呈现了正常的Cas9蛋白和dCas9蛋白. 图B的第一幅图将 w subunit与dCas9 蛋白融合, 这样Cas9蛋白可以定位到特定的位置, w subunit可以召集转录因子, 由此激发转录过程, 第二幅图, dcas9可以结合到RNA聚合酶的下游, 阻挡聚合酶继续转录从而抑制转录. 图C中则同通过融合VP64去激活哺乳动物细胞的基因转录和通过融合KRAB去抑制转录. 而图D则更为先进, 相比于图C中只融合一个VP64激活因子来说, 这里的方法是融合一个多肽(scFV peptide), 其中包含了VP64, 而多肽之间是可以结合的, 这样就大大的提升了激活因子VP64的召集率, 一个dCas9蛋白最多可以召集24个VP64, 大大提升转录效率. CRISPR系统在转录的激活与抑制上还有很多应用, 这里不详细介绍, 有兴趣的读者可以去自行阅读下方的参考文献学习.
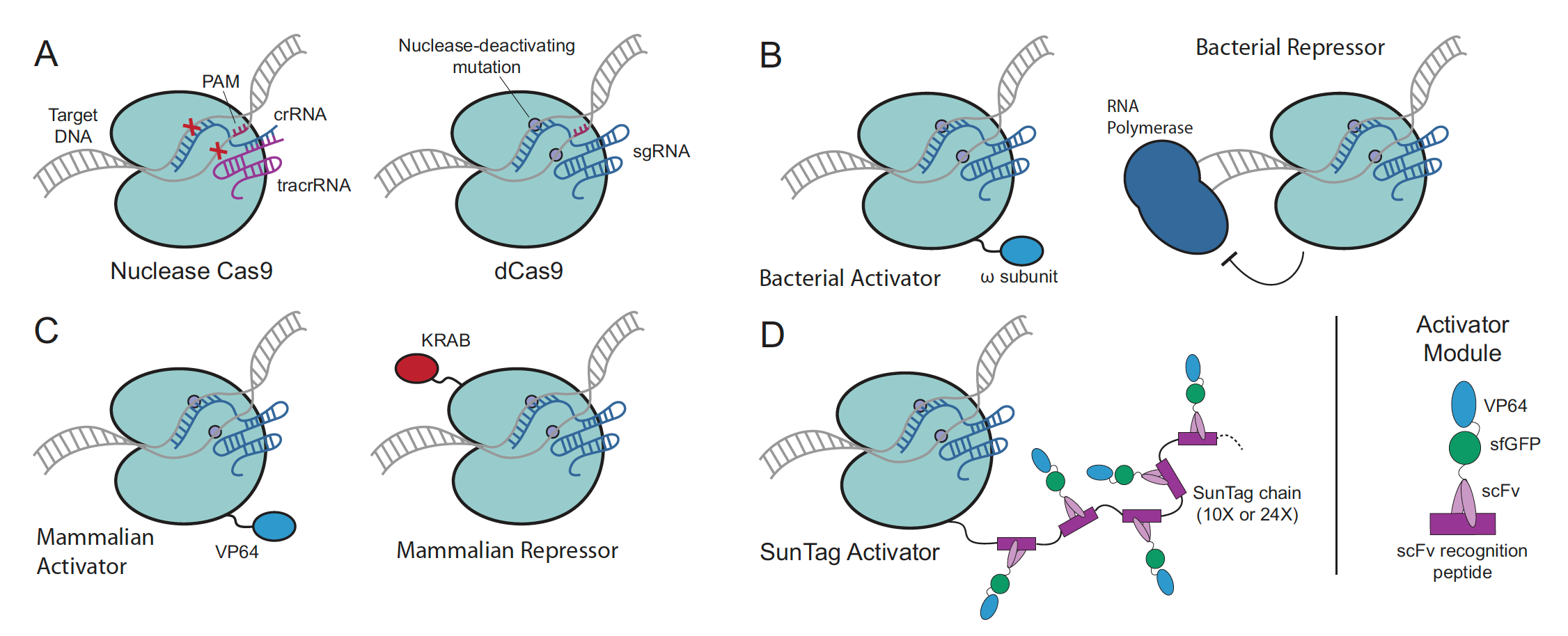
Figure 12. Engineered CRISPR interference systems and different applications.
除了转录的激活和抑制之外, 还可以把荧光蛋白融合到dcas9上, 这样可以通过guide RNA 的引导去定性结合特殊的基因位点, 从而便于观察. 这对于观察一个目的基因的表达强度和表达历程是非常有用的. (Fig.13)
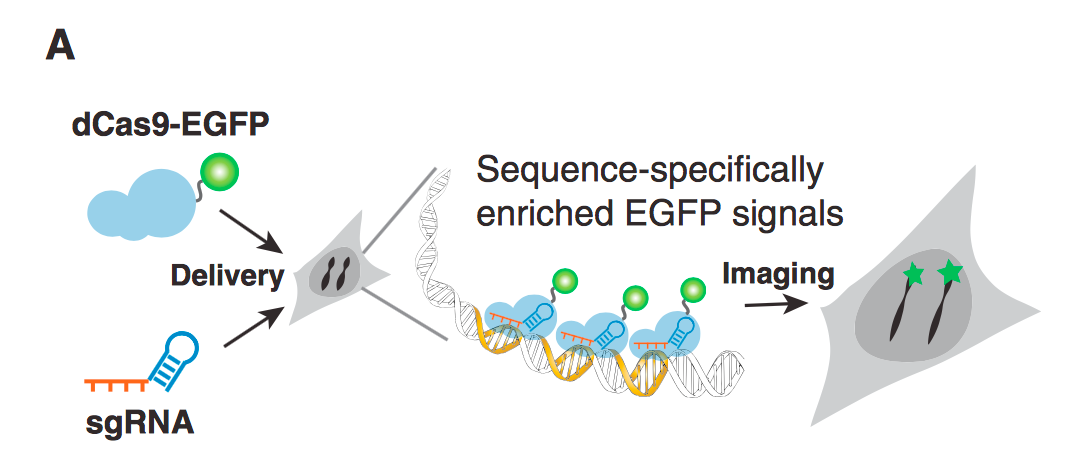
Figure 13. Overview of CRISPR imaging. Sequence-specific enrichment of fluorescence signals by sgRNA-directed dCas9-EGFP allows the imaging of genomic elements in living cells.
(7) Gene drive (基因驱动)技术
一个非常典型的例子就是基于CRISPR 技术上的Gene drive技术. 这个技术可以理论上完全改变一个生态圈中的野生型(Wild type)物种. 哈佛大学医学院的Wyss机构率先提出的通过基因驱动技术可以扭转生态中具有携带疟原虫相关基因的野生型蚊子, 换句话说就是使用CRISPR 技术让所有可以让蚊子携带疟原虫的相关基因消失 , 理论上在几年时间中就可以根除疟疾. 具体的理论设想如下:
1. 首先对蚊子野生型蚊子的染色体基因(与携带疟原虫相关)进行编辑
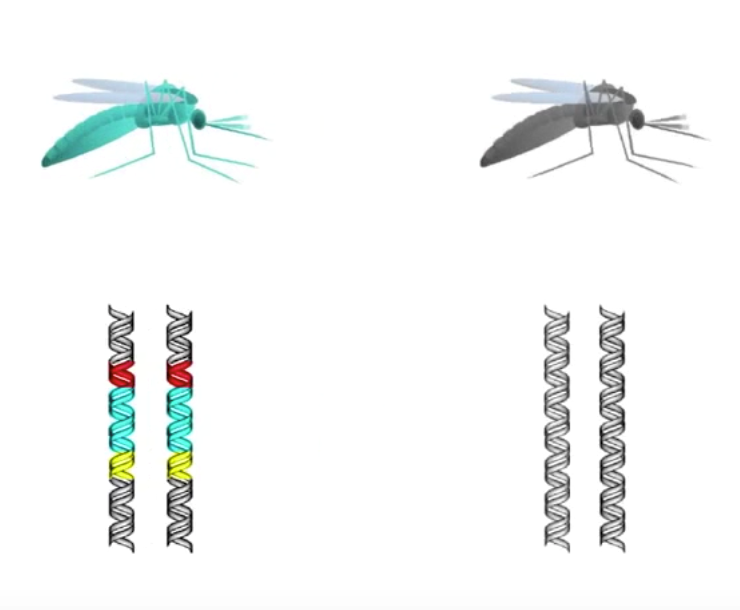
蓝色的蚊子是携带gene drive的CRISPR系统的, 灰色的蚊子是野生型. 其中, 蓝色的基因代表与携带疟原虫相关的基因的位置(蓝色代表已经编辑过, 无法携带疟原虫), 在野生型文字中的基因位置是一样的, 因为他们是等位基因. 野生型内的基因是可以使蚊子携带疟原虫的. 红色的基因代表sgRNA, 黄色的基因代表Cas9蛋白.
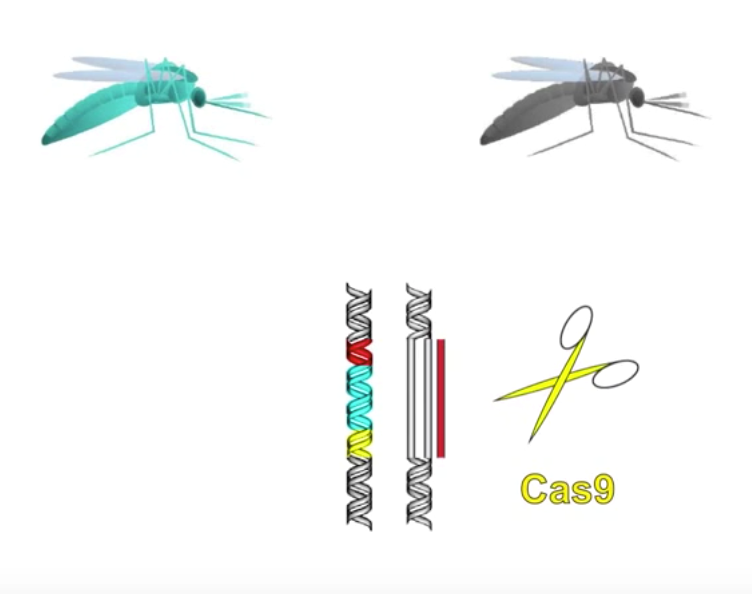
当携带gene drive的蚊子和野生型蚊子相交配后, 两个同源染色体配对. 但是, 蓝色蚊子的一条染色体可以表达出sgRNA和Cas9, 红色sgRNA可以引导Cas9区剪切野生型中与携带疟原虫相关的基因有关的基因, 从而这条野生型的染色体DNA发生双键断裂.
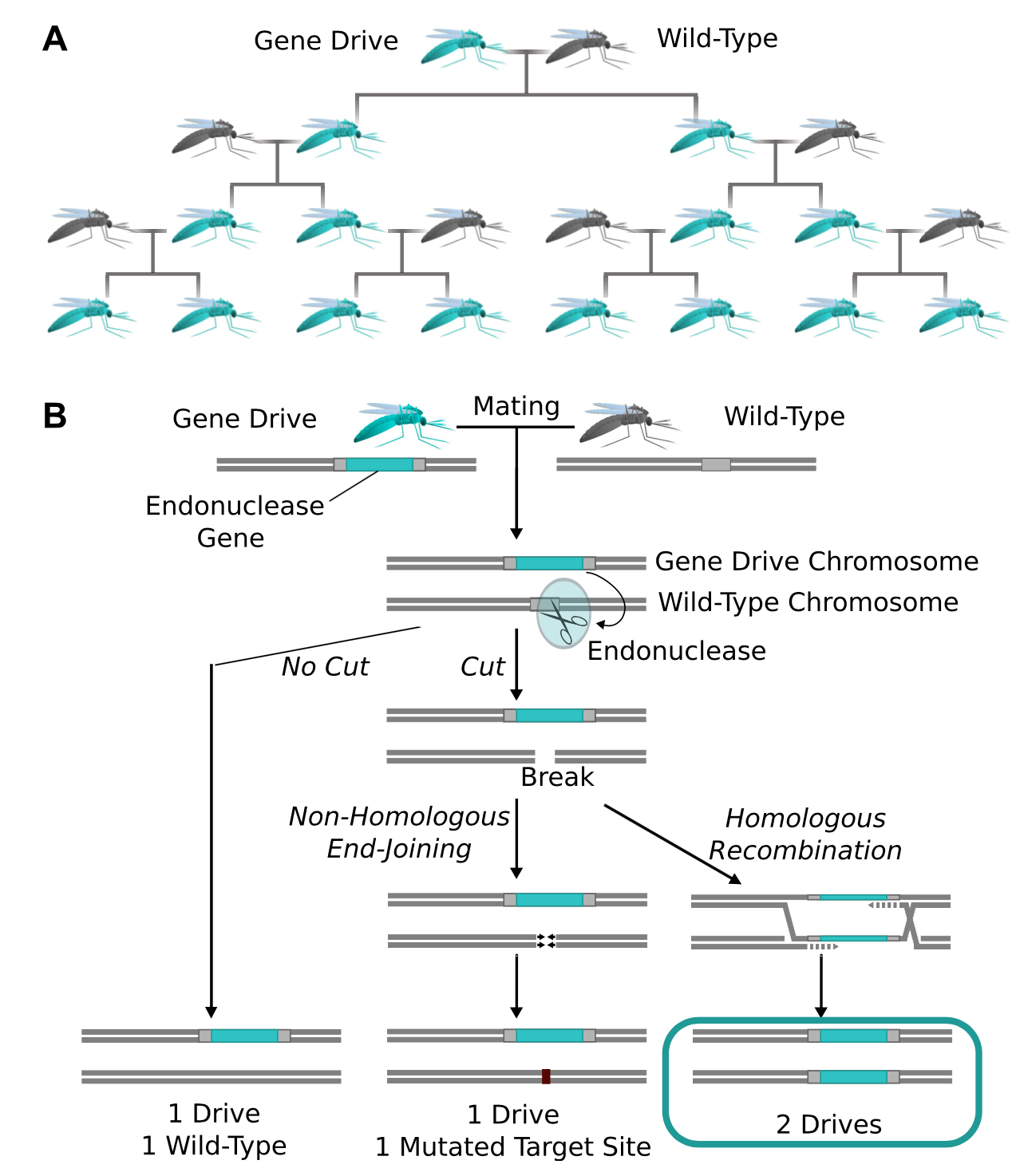
再发生双键断裂之后, 断裂的染色体DNA势必会以另一条同源染色体以模板进行同源重组的修复, 最终野性型的染色体DNA的断裂位置会编程和蓝色蚊子的染色体DNA 同位置的基因一样, 携带此染色体的蚊子无法携带疟原虫. 通过这样的基因驱动, 数代杂交过后, 所有蚊子理论上都不携带疟原虫基因了.
基因驱动的技术不止可以应用于蚊子, 而且可以应用于很多物种. 但是这种技术虽然可以快速根除疟疾, 但是可能会对生态圈造成不可逆转的改变. 因此在伦理上存在着诸多问题, 是否能够付诸于实践. 还需要继续讨论
(8) 总结
CRISPR/Cas技术的确是一个非常强大且使用简便的基因编辑工具, 在医疗和科研技术上有着不可低估的潜力. 但是同样CRISPR技术的发展面临着技术和伦理的限制, 就技术层面上来说, 很多实验表明CRISPR 系统体外的脱靶效率还是较高的, 如果想把CRISPR技术应用到医疗领域, 那么如何降低脱靶效率是一个迫切需要解决的问题. 就伦理层面, CRISPR技术的简便和强大是否会造成不恰当的使用? 这些都需要谨慎考虑.
本篇文章详细的介绍了CRISPR 技术的原理和应用, 但还是比较缩减和片面的. 有很多东西由于篇幅限制没有提到, 那么作者也会在今后的文章中进行更全面的介绍, 并介绍CRISPR 技术最新的应用, 敬请关注.
附录:
Appendix 1:
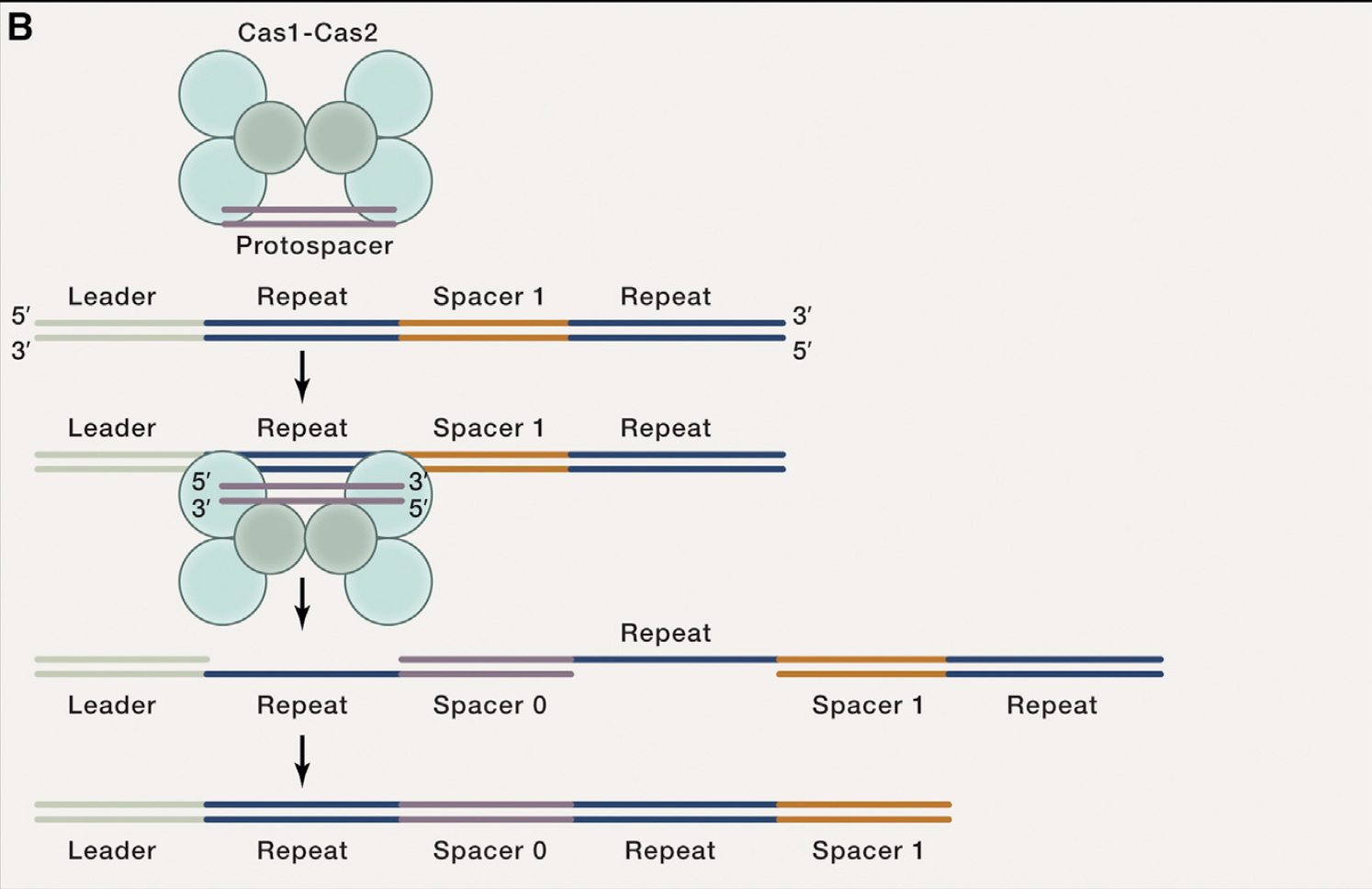
Cas的相关基因再选择protospacer后会携带这双联的DNA附着到CRISPR array上. Cas蛋白复合体会切断CRISPR array第一个repeat序列两条链的3末端, 并插入protospacer到断裂的第一个repeat中间. 这样就实现了repeat的复制和protospacer的插入. 同时也满足了下一个spacer插入的条件.
Appendix 2:
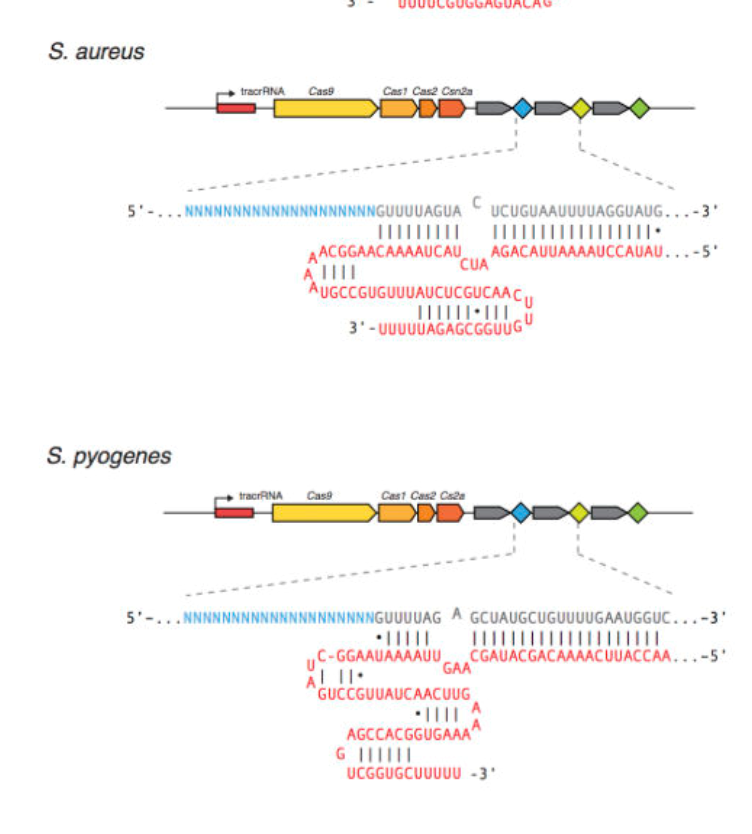
本图是SaCas9 和SpCas9的guide RNA 的示意图. 红色的序列代表tracrRNA, 灰色的代表repeat序列, 蓝色的代表spacer序列.
参考文献:
Chen, Baohui, et al. "Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system." Cell 156.1 (2014): 373.
Deveau, H., Garneau, J.E. and Moineau, S., 2010. CRISPR/Cas system and its role in
phage-bacteria interactions. Annual review of microbiology, 64, pp.475-493.
La Russa, M.F. and Qi, L.S., 2015. The new state of the art: Cas9 for gene activation and repression. Molecular and cellular biology, 35(22), pp.3800-3809.
Makarova, K.S., Wolf, Y.I., Alkhnbashi, O.S., Costa, F., Shah, S.A., Saunders, S.J.,
Barrangou, R., Brouns, S.J., Charpentier, E., Haft, D.H. and Horvath, P., 2015. An updated evolutionary classification of CRISPR–Cas systems. Nature Reviews. Microbiology, 13(11), p.722.
Ran, F.A., Cong, L., Yan, W.X., Scott, D.A., Gootenberg, J.S., Kriz, A.J., Zetsche, B., Shalem, O., Wu, X., Makarova, K.S. and Koonin, E.V., 2015. In vivo genome editing using Staphylococcus aureus Cas9. Nature, 520(7546), pp.186-191.
Shmakov, S., Abudayyeh, O.O., Makarova, K.S., Wolf, Y.I., Gootenberg, J.S., Semenova,
E., Minakhin, L., Joung, J., Konermann, S., Severinov, K. and Zhang, F., 2015. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular cell, 60(3), pp.385-397.
Wang, H., La Russa, M. and Qi, L.S., 2016. CRISPR/Cas9 in genome editing and beyond. Annual review of biochemistry, 85, pp.227-264.
Wright, A.V., Nuñez, J.K. and Doudna, J.A., 2016. Biology and applications of CRISPR systems: harnessing nature’s toolbox for genome engineering. Cell, 164(1), pp.29-44.
原文地址:https://zhuanlan.zhihu.com/p/29204199 |
|
 /3
/3 