金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
1.前言
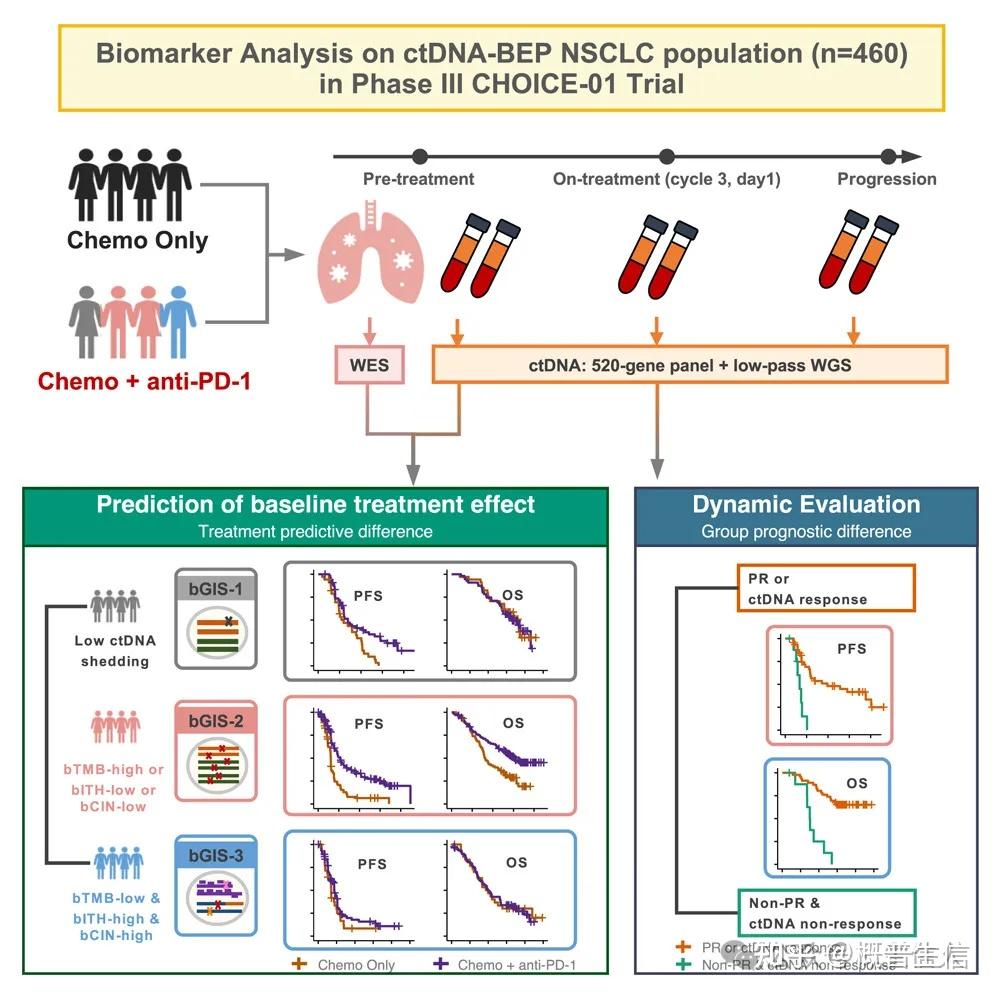
2024年9月9日,中国医学科学院肿瘤医院王洁教授团队在Cell Press细胞出版社旗下期刊Cancer Cell(IF:48.80)在线发表了题为“Circulating tumor DNA-based stratification strategy for chemotherapy plus PD-1 inhibitor in advanced non-small-cell lung cancer”的前瞻性转化研究成果。
该研究对 3 期 CHOICE-01 研究中 460 例患者在不同时间点前瞻性收集的循环肿瘤 DNA (ctDNA) 标本进行了高通量低深度NGS和低通全基因组测序。确定了化疗加 PD-1 抑制剂的预测标志物,包括 ctDNA 状态和基因组特征,例如基于血液的肿瘤突变负荷、肿瘤内异质性和染色体不稳定性;还建立了一种基于 ctDNA 的综合分层策略,即基于血液的基因组免疫亚型 (bGIS) 方案,以区分在一线化疗中加入 PD-1 抑制剂的患者;展示了 ctDNA 动态监测的潜在应用。总体而言,提出了一种基于基于 ctDNA 的分层策略的潜在治疗算法,阐明了晚期 NSCLC 患者免疫化疗的个体化管理。
在接受Cell Press细胞出版社专访时,王洁教授指出,这项转化研究是首个基于“免疫联合化疗”模式的晚期非小细胞肺癌(NSCLC)大规模基因组生物标志物探索。研究建立了循环肿瘤DNA(ctDNA)特征与肿瘤生物学特征之间的直观联系,识别出几种能预测免疫联合化疗疗效的关键基因组特征,并构建了基于ctDNA的精准免疫治疗分层管理策略。值得注意的是,该策略中所有指标均可通过外周血无创检测获得,进一步推动了无创分型向精细化免疫治疗的转化。
这是王洁教授近20年聚焦ctDNA分子分型系列研究的拓展和延伸。在此处,从无创检测的概念入手,我们可以理解其重要性。
临床活体取样是现代医学诊断的重要手段,通过从患者体内获取组织样本进行分析,为疾病的诊断和治疗方案提供关键信息。这一技术广泛应用于癌症、自身免疫性疾病等多种疾病的诊断。常见的取样方法包括穿刺活检、内镜活检和手术活检等。近年来,随着精准医疗的发展,活体取样技术不断进步,但仍面临许多挑战:某些深部或微小病变的精准定位和取样依然困难;出血和感染等并发症无法完全避免;此外,活体取样还可能增加患者的身心负担和经济压力。由于肿瘤等病变组织常常存在异质性,单次取样可能无法全面反映病变特征,理论上还可能导致肿瘤细胞的播散。
随着影像引导技术的进步,微创活检逐渐成为趋势,它能在局部麻醉下进行,显著减少了患者的痛苦和并发症风险。同时,液体活检等新兴技术也为传统活检提供了部分替代方案。液体活检从外周血及其基因组中分离肿瘤相关成分或进行蛋白质组学评估,催生了一种新的诊断工具。与传统组织活检相比,癌症液体活检具有多项优势:
- 取样便利性:液体活检采用无创或微创方法,血液、唾液或尿液等体液更易获取,显著降低了活检手术的风险和成本。
- 全身性特征:它能够提供人体所有肿瘤病变的全身性和均质性特征,克服了组织活检在肿瘤内或肿瘤间异质性带来的局限。
- 实时监测:根据需要进行采样,可以监测癌症在不同发展阶段的实时变化。
液体活检中的生物标志物,特别是循环肿瘤DNA(ctDNA)、循环肿瘤RNA(ctRNA)、循环肿瘤细胞(CTC)、细胞外囊泡(EV)等,为癌症的早期诊断、治疗选择、监测和疾病评估提供了希望。由于液体活检的非侵入性特点,以及其在多个时间点的重复采样能力,使得疾病的监测变得更加方便,因此,精准医学越来越关注液体活检这一领域。 接下来将简单介绍液体活检生物标志物及其相关的研究发现。
2.cfDNA/ctDNA
cfDNA是液体活检的重要标志物,已广泛用于无创产前检查和癌症液体活检研究。接下来将简单介绍cfDNA检测技术及其潜在的临床应用。
血浆中cfDNA片段的断裂模式以及基因组上的覆盖是不均匀的,这些都可以间接反映体内基因表达调控的状态,例如核小体定位和基因表达活性,具体的片段特征包括:Size、末端位置及末端序列Motif等。此外,外周血中的cfDNA是在细胞凋亡或坏死后释放的,主要来自健康人的造血细胞和病人的病变组织,cfDNA片段组学的最新研究成果也展示出追踪肿瘤游离DNA(ctDNA)来源的潜力。cfDNA 由双链DNA片段组成,这些片段非常短,长度基本小于200碱基对(bp), 通常浓度很低。cfDNA片段模式的主要峰约为166个碱基对,并被认为与核 小体结构有关。cfDNA从大约143bp开始,相距约10bp的一系列连续峰值。
一种称为DELFI(DNA evaluation of fragments for early interception)的方法,通过全基因组分析cfDNA片段模式,能够检测大量异常。该方法依赖于低覆盖率的全基因组测序,分析健康和癌症人群中cfDNA片段的覆盖率和大小分布。与健康样本相比,癌症患者显示出多种基因组差异,片段大小在不同区域呈现增加和减少。DELFI可同时分析微量cfDNA中的数十到数百种肿瘤特异性异常,有助于确定cfDNA的来源,并利用临床特征和甲基化变化进一步改进诊断。其所需的全基因组测序量少,显示出在癌症筛查和管理中的广泛应用潜力。
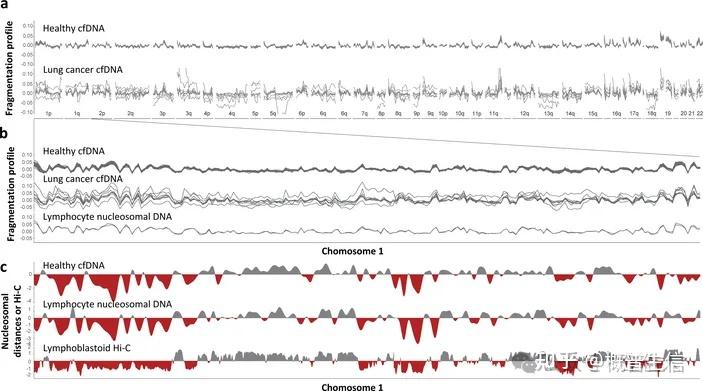
Peter Ulz开发并验证了一种基于cf DNA 测序及核小体足迹分析的微创方法,能够评估转录因子(TF)的活性。作者观察到肿瘤特异性的TF结合位点模式可用于癌症早期诊断和分子分型。基于血液样本在体内绘制肿瘤特异性转录因子结合的方法使非编码基因组的关键部分适合临床分析。由于 TF 在整个基因组中调节基因转录的潜力及其通常精致的谱系特异性,它们基于 cfDNA 的详细无创分析为改进临床诊断提供了独特的机会。
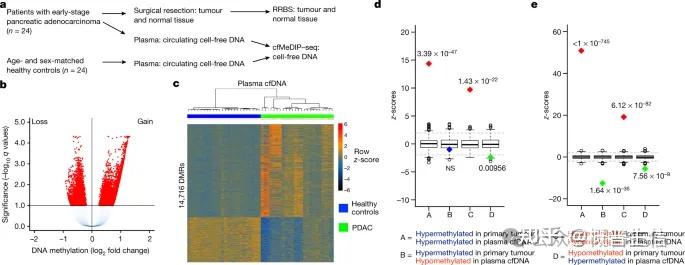
Shen SY 等人开发了一种灵敏的、基于免疫沉淀的方案来分析少量循环游离 DNA 的甲基化组,即开发了甲基化 cfDNA 免疫沉淀和高通量测序 (cfMeDIP-seq),用于全基因组无亚硫酸氢盐血浆 DNA 甲基化分析,并展示了检测富含肿瘤特异性模式的大规模 DNA 甲基化变化的能力。还在来自多种肿瘤类型的大量血浆样本中展示了癌症检测和分类方面的稳健表现。这项工作为建立基于浆细胞游离 DNA 甲基化模式的早期癌症微创检测、拦截和分类生物标志物奠定了基础。
除了上述应用,cfDNA分析还可通过新一代测序技术(NGS)评估体细胞的改变,包括基因突变、拷贝数变异和基因融合。cfDNA技术还包括T细胞受体测序及相关微生物组分析技术,展示了其在现代医学中的广泛应用前景。
3.CTC
CTC(Circulating Tumor Cells)是指从原发肿瘤脱落并进入血管系统的细胞,这些细胞具有完整的活核、阳性细胞角蛋白、上皮细胞粘附分子(EpCAM)及阴性CD45分子。在癌症诊断和个性化医疗中,CTC的数量和异质性发挥着关键作用。
几项研究表明,CTC 计数的差异可用于结直肠癌 (colorectal cancer ,CRC) 以区分良性疾病患者和结直肠癌患者。Yang 等人的一项研究报告称,与结直肠息肉患者相比,结直肠癌患者的 CTC 计数明显更高。另一方面,CTC 计数也根据解剖位置和组织分化而变化,低分化肿瘤显示计数较高。位置方面,在乙状结肠瘤中观察到的计数最高,其次是直肠肿瘤、升结肠、横结肠和降结肠,表明 CTC 计数在诊断/筛查中的作用 。同样,一项关于 I-IV 期 CRC、腺瘤和健康对照者的研究表明,IV 期 CRC 患者的平均 CTC 计数最高,而腺瘤和健康对照者的平均 CTC 计数较低。该研究证明,CTC 计数可以区分健康组和腺瘤组以及处于不同癌症阶段的患者,表明 CTC 计数在 CRC 中的诊断价值。
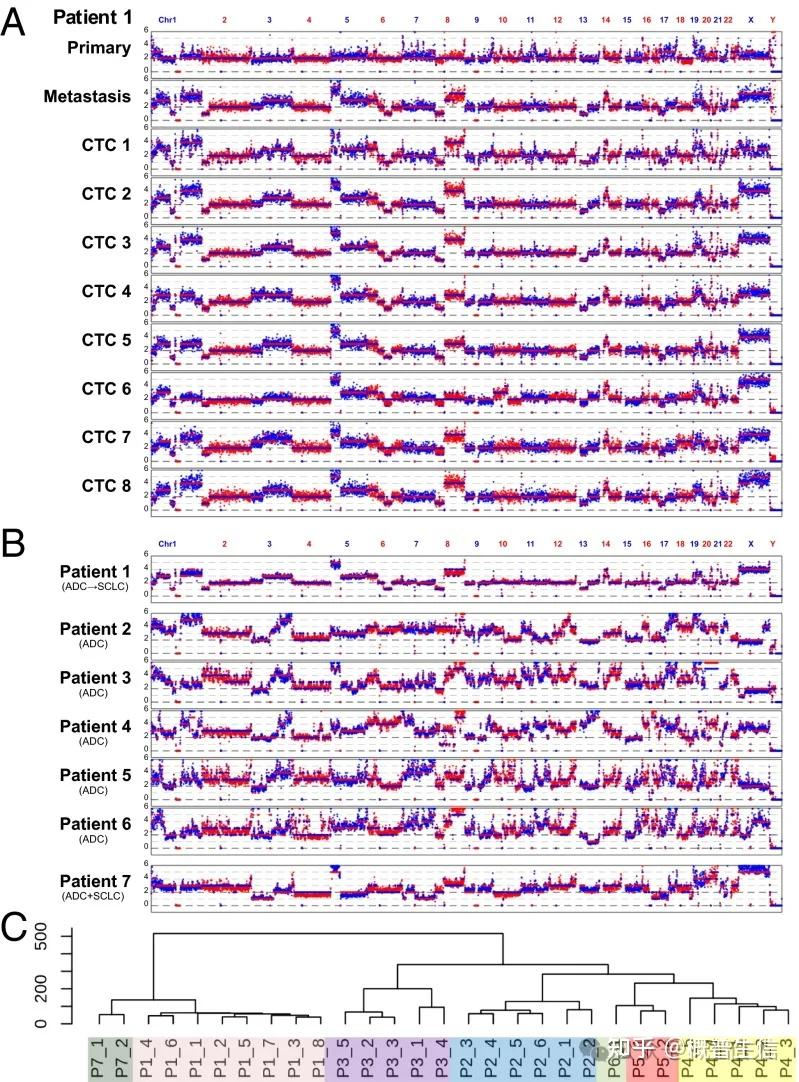
CTC的分子特征有助于深入理解转移过程中的关键环节,并为个性化医学的方法提供支持。在此处,我们简单例举了CTC基因组的相关研究。研究者报告了使用开发的基于多重退火和循环的扩增循环对肺癌患者的单个 CTC 进行全基因组扩增。在 CTC 的外显子组中观察到特征性的癌症相关单核苷酸变异和插入/缺失。这些突变提供了个体化治疗所需的信息,例如耐药性和表型转换,但细胞间存在异质性。相比之下,来自个体患者的每个 CTC,无论癌症亚型如何,都表现出可重复的拷贝数变异 (CNV) 模式,类似于同一患者的转移性肿瘤。有趣的是,具有相同肺癌腺癌 (ADC) 的不同患者在其 CTC 中具有相似的 CNV 模式。更有趣的是,小细胞肺癌患者的 CNV 模式与 ADC 患者的 CNV 模式明显不同。这些研究结果表明,某些基因组位点的 CNV 被选择用于癌症的转移。癌症特异性 CNV 的可重复性为基于 CTC 的癌症诊断提供了潜力。
4.EV
细胞外囊泡 (EV) 已成为细胞间通讯的关键参与者,促进了细胞之间关键货物的转移。液体活检,特别是通过分离 EV,揭示了健康和疾病潜在生物标志物的丰富来源,包括蛋白质和核酸。
Susana García-Silva等人首次证明渗出性血清肿( exudative seroma ,ES )衍生的 EV 可能代表黑色素瘤进展的有用替代标志物,并且适用于检测黑色素瘤特异性突变。与血浆相比,通过淋巴引流获得的 ES 是一种富含 EV 的生物流体。还发现 ES 衍生的外泌体和黑色素瘤细胞系分泌的外泌体中的蛋白质和通路之间存在显着重叠。此外,在通过淋巴引流获得的 ES 囊泡中检测 BRAF 突变可能是识别可能由于残留病灶而有复发风险的患者的新参数。这些患者随后可以在手术后立即从特定的辅助治疗中受益。
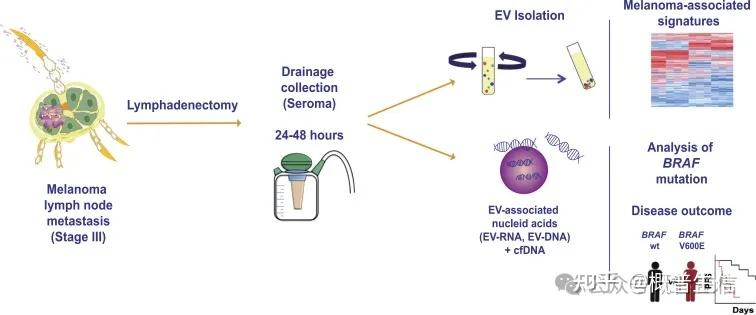
鉴于 EV 封装了反映细胞特异性病理过程的受保护携带物,它们作为循环生物标志物具有巨大潜力,为增强当前的癌症监测、预后和治疗策略提供了一条有前途的途径。这反映在许多采用液滴数字(dd)PCR 和各种测序方法来检测 EV 相关突变 DNA 拷贝的研究中。
总结
液体活检是一个不断发展的概念,随时间推移,其外延也在不断扩展。从广为人知的蛋白类肿瘤标志物,到如今的CTC、ctDNA,再到外泌体,液体活检始终是一个试验性、探索性的课题。相信在不久的将来,液体活检将能够更有效地促进癌症的早期诊断及后期治疗,为广大患者带来福音。
参考文献
1. Xu J, Wan R, Cai Y, et al. Circulating tumor DNA-based stratification strategy for chemotherapy plus PD-1 inhibitor in advanced non-small-cell lung cancer. Cancer Cell. 2024;42(9):1598-1613.e4. doi:10.1016/j.ccell.2024.08.013
2.Palmirotta R, Lovero D, Cafforio P, et al. Liquid biopsy of cancer: a multimodal diagnostic tool in clinical oncology. Ther Adv Med Oncol. 2018;10:1758835918794630. Published 2018 Aug 29. doi:10.1177/1758835918794630
3. Ding Z, Wang N, Ji N, Chen ZS. Proteomics technologies for cancer liquid biopsies. Mol Cancer. 2022;21(1):53. Published 2022 Feb 15. doi:10.1186/s12943-022-01526-8 SPEICHER M R, PANTEL K. Tumor signatures in the blood.
4. Fleischhacker M, Schmidt B. Circulating nucleic acids (CNAs) and cancer--a survey. Biochim Biophys Acta. 2007;1775(1):181-232. doi:10.1016/j.bbcan.2006.10.001Maternal plasma DNA sequencing reveals the genome-wide genetic and mutational profile of the fetus [J]. Science Translational Medicine, 2010, 2 (61): 61ra91-61ra91. [44] Dennis M Y L ,C S D H ,Peiyong J , et al.
5. Lo YMD, Han DSC, Jiang P, Chiu RWK. Epigenetics, fragmentomics, and topology of cell-free DNA in liquid biopsies. Science. 2021;372(6538):eaaw3616. doi:10.1126/science.aaw3616.
6. Cristiano S, Leal A, Phallen J, et al. Genome-wide cell-free DNA fragmentation in patients with cancer. Nature. 2019;570(7761):385-389. doi:10.1038/s41586-019-1272-6Inferring expressed genes by whole-genome sequencing of plasma DNA
7.Esfahani MS, Hamilton EG, Mehrmohamadi M, et al. Inferring gene expression from cell-free DNA fragmentation profiles. Nat Biotechnol. 2022;40(4):585-597. doi:10.1038/s41587-022-01222-4
8.Shen SY, Singhania R, Fehringer G, et al. Sensitive tumour detection and classification using plasma cell-free DNA methylomes. Nature. 2018;563(7732):579-583. doi:10.1038/s41586-018-0703-0
9.Yang C, Zhuang W, Hu Y, Zhu L. Clinical significance of peripheral circulating tumor cell counts in colorectal polyps and non-metastatic colorectal cancer. World J Surg Oncol. 2018;16(1):13.
10.sai WS, You JF, Hung HY, Hsieh PS, Hsieh B, Lenz HJ, et al. Novel Circulating Tumor Cell Assay for Detection of Colorectal Adenomas and Cancer. Clin Transl Gastroenterol. 2019;10(10):e00088.
11.Ni X, Zhuo M, Su Z, et al. Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients. Proc Natl Acad Sci U S A. 2013;110(52):21083-21088. doi:10.1073/pnas.1320659110
11. Tsering T, Nadeau A, Wu T, Dickinson K, Burnier JV. Extracellular vesicle-associated DNA: ten years since its discovery in human blood. Cell Death Dis. 2024;15(9):668. Published 2024 Sep 12. doi:10.1038/s41419-024-07003-y
12.García-Silva S, Benito-Martín A, Sánchez-Redondo S, et al. Use of extracellular vesicles from lymphatic drainage as surrogate markers of melanoma progression and BRAFV600E mutation [published correction appears in J Exp Med. 2019 May 6;216(5):1230. doi: 10.1084/jem.2018152204162019c]. J Exp Med. 2019;216(5):1061-1070. doi:10.1084/jem.20181522
原文地址:https://zhuanlan.zhihu.com/p/721967421 |
|
 /3
/3 