金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
太长不看版:
1. 病毒也可以有寄生体,被称为噬病毒体
2. 噬病毒体可以干扰宿主病毒的复制
3. 噬病毒体是否属于卫星病毒尚存异议
我们小学二年级就学到过,病毒是一种寄生体。它们没有细胞结构,必须通过寄生于细胞才能够复制。如果继续套娃的话,那么病毒的病毒(以下称为噬病毒体)应当是依赖宿主病毒才能复制的寄生体。噬病毒体的发现和另外一种神奇的病毒息息相关-巨大病毒。
病毒也可以很大
传统意义上病毒应当是非常微小的,光学显微镜下不可见的生物。然而生物界当中总是不缺少意外的,80年代早期科学家在调查军团菌(自然条件下有很多是需要寄生在阿米巴原虫当中的)时发现了一些看起来像是小的革兰氏阳性球菌(传说中的金黄色葡萄球菌就是革兰氏阳性球菌)。在电子显微镜下这个“细菌”才显现出它的真面孔,即使直径约600 纳米,它却具有一个多面体对称的常见于病毒的结构,被称为米米病毒(mimivirus)(Colson et al. 2017)。
米米病毒是一种双链DNA病毒,除了巨大以外还有许多独特的特征:它的1.2兆DNA基因组的大小可以媲美一些细菌;自带转录因子,tRNA合成酶和编码六种tRNA等等(Colson et al. 2017)。要知道传统上看病毒应当是一种很“懒”的生物,只会自带一些十分必要的基因(至少RNA病毒是这样)。最为离谱的是,它居然还具有一套很可能类似于CRISPR的免疫系统,名为MIMIVIRE(Levasseur et al. 2016)。作为寄生体的米米病毒,究竟要免疫谁呢?
图片来源(Colson et al. 2017)
病毒的寄生虫-噬病毒体(virophage)
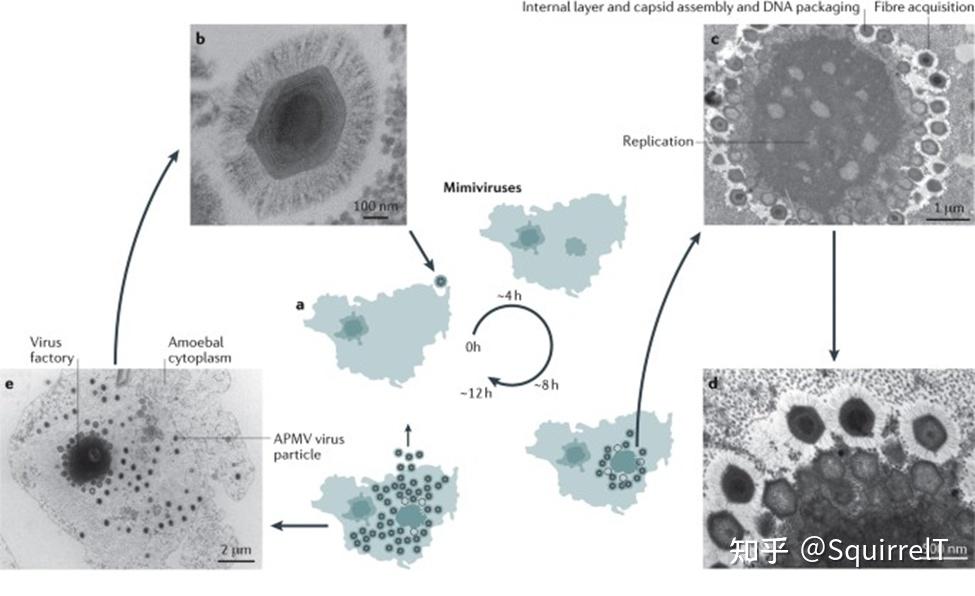
在另外一种巨大病毒玛玛病毒(mamavirus,不得不说这群人起名有点恶趣味)在阿米巴原虫当中复制时会形成一个病毒工厂来生产新的玛玛病毒,然而在这个工厂当中科学家却观察到了另一种更小的多面体(~50纳米),暗示着另外一种病毒的存在(这里要说并不是所有的病毒都有多面体结构)。进一步的研究显示,这种病毒仅仅在玛玛病毒感染阿米巴原虫时才能复制,并且这种未知病毒(sputnik)复制时会干扰玛玛病毒的正常形成和降低玛玛病毒的产量,少数情况下sputnik会被包裹进入新形成的玛玛病毒当中。这种新的病毒被归类为噬病毒体(La Scola et al. 2008)。
后续研究陆续发现了新的噬病毒体,其中一个有趣的存在是一种被称为mavirus的噬病毒体。众所周知,我们的基因组当中存在着许多内源性病毒,它们在历史上的某个阶段感染并整合进某个幸运的生殖细胞当中被保留下来,虽然被认为基本上失去活性,但有些产物依然起着十分重要的作用(这是一个十分有趣的话题,改日再聊)。在原生动物(阿米巴原虫是其中之一)当中也存在着这么一段被称为前病毒的序列,当这个原生动物被一种巨大病毒感染时会被激活并制造出噬病毒体,这些噬病毒体可能并不能保护这只原生动物,但是可以保护其他的原虫,形成了十分有趣的生态现象。你似乎也可以把mavirus视为这些原虫群体免疫系统的一部分(Fischer & Suttle 2011; Fischer & Hackl 2016)。

图片来源(La Scola et al. 2008)
噬病毒体与卫星病毒
至此,接受过高于二年级微生物学教育的同学们似乎有想到一个问题,这些噬病毒体与卫星病毒有什么区别?卫星病毒也是一种依赖于助手病毒(helper virus)才能够复制的病毒,比较经典的有丁肝病毒(HDV),它的“宿主”or“助手”病毒是乙肝病毒(Mentha et al. 2019)。丁肝病毒几乎所有的蛋白都来自于乙肝病毒,自身仅仅由一段~1680碱基大小的基因组表达一种蛋白(神奇但无关的是:这段两端共价相连,中间大部分碱基配对的RNA基因组似乎由宿主的DNA为模板的RNA聚合酶复制)(Mentha et al. 2019)。当两种病毒共同感染时,通常会导致两种病毒都被宿主消除;而当HDV感染已有慢性乙肝的患者时会加重肝炎的破坏。可见,丁肝病毒作为卫星病毒似乎和噬病毒体有诸多相似点(Negro 2014)。
事实上,噬病毒体究竟是否属于卫星病毒是一个值得辩论的问题(看科学家吵架!)(Krupovic & Cvirkaite-Krupovic 2011b; Krupovic & Cvirkaite-Krupovic 2011a; Fischer 2011)。从定义上看,噬病毒体符合卫星病毒依赖于助手病毒复制的定义。可以理解的是,作为噬病毒体的发现者认为由于噬病毒体具备一些独特的特性:较大的基因组和干扰助手病毒复制。然而这些特征也被指出并非噬病毒体独有。
个人认为,虽然噬病毒体作为独立于卫星病毒存在的单独分类似乎有些牵强,但噬病毒体依然具有许多有趣的特征,毫无疑问的扩展了我们对病毒世界的理解,且可能存在着生态学和进化方面的重要意义。一点扩展,虽然很可能人们已经想到的是,可否利用噬病毒体/卫星病毒用作治疗病毒感染。
参考文献
Colson P, La Scola B, Levasseur A, Caetano-Anollés G & Raoult D (2017) Mimivirus: leading the way in the discovery of giant viruses of amoebae. Nature Reviews Microbiology 2017 15:4 15, 243–254. Available at: https://www.nature.com/articles/nrmicro.2016.197 [Accessed December 21, 2023].
Fischer MG (2011) Sputnik and Mavirus: more than just satellite viruses. Nature Reviews Microbiology 2011 10:1 10, 78–78. Available at: https://www.nature.com/articles/nrmicro2676-c1 [Accessed January 1, 2024].
Fischer MG & Hackl T (2016) Host genome integration and giant virus-induced reactivation of the virophage mavirus. Nature 540, 288–291. Available at: https://www.nature.com/articles/nature20593 [Accessed December 21, 2023].
Fischer MG & Suttle CA (2011) A virophage at the origin of large DNA transposons. Science (1979) 332, 231–234. Available at: https://www.science.org/doi/10.1126/science.1199412 [Accessed December 21, 2023].
Krupovic M & Cvirkaite-Krupovic V (2011a) Sputnik and Mavirus: not more than satellite viruses. Nature Reviews Microbiology 2011 10:1 10, 78–78. Available at: https://www.nature.com/articles/nrmicro2676-c2 [Accessed December 21, 2023].
Krupovic M & Cvirkaite-Krupovic V (2011b) Virophages or satellite viruses? Nature Reviews Microbiology 2011 9:11 9, 762–763. Available at: https://www.nature.com/articles/nrmicro2676 [Accessed December 21, 2023].
Levasseur A, Bekliz M, Chabrière E, Pontarotti P, La Scola B & Raoult D (2016) MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. Nature 2016 531:7593 531, 249–252. Available at: https://www.nature.com/articles/nature17146 [Accessed January 1, 2024].
Mentha N, Clément S, Negro F & Alfaiate D (2019) A review on hepatitis D: From virology to new therapies. J Adv Res 17, 3–15.
Negro F (2014) Hepatitis D Virus Coinfection and Superinfection. Cold Spring Harb Perspect Med 4, a021550. Available at: http://perspectivesinmedicine.cshlp.org/content/4/11/a021550.full [Accessed December 21, 2023].
La Scola B, Desnues C, Pagnier I, Robert C, Barrassi L, Fournous G, Merchat M, Suzan-Monti M, Forterre P, Koonin E & Raoult D (2008) The virophage as a unique parasite of the giant mimivirus. Nature 455, 100–104. Available at: https://www.nature.com/articles/nature07218 [Accessed December 21, 2023]. |
|
 /3
/3 