金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
一.液体活检之ctDNA概述
循环肿瘤细胞、循环肿瘤DNA 和外泌体的检测并称为“液体活检”。“液体活检”是一种新兴的无创检测技术, MIT Technology Review 杂志将“液体活检”技术评为2015 年度十大突破技术之一。
ctDNA(circulating tumor DNA,循环肿瘤DNA)是指人体血液循环系统中不断流动的携带一定特征(包括突变, 缺少,插入,重排,拷贝数异常,甲基化等)来自肿瘤基因组的DNA 片段。ctDNA 的主要来源包括:1、来自坏死的肿瘤细胞;2、来自凋亡的肿瘤细胞;3、循环肿瘤细胞;4、来自肿瘤细胞分泌的外排体。
ctDNA 对于早期和局部肿瘤的检测还有一定的局限性, 目前主要用于晚期肿瘤的检测。临床试验研究表明: 1) ctDNA浓度大小与肿瘤负荷大小成正比, 但并不能确定肿瘤的分期、定位和大小; 2) ctDNA 检测尤其是ctDNA 突变的检测可监测肿瘤进展及预后, 而且有研究指出ctDNA 的水平可以作为评估某些肿瘤(如卵巢癌和子宫内膜癌)预后的一个独立指标; 3) 通过对ctDNA 水平的监测能够检测到肿瘤患者组织和血浆中存在的特有的突变以便准确地对肿瘤进行分型, 从而指导临床靶向治疗; 4) ctDNA 检测可反映肿瘤是否发生复发转移及是否伴有微小残留疾病的存在; 5) ctDNA 检测可反映抗癌治疗是否起效及是否出现耐药性信息, 以便及时调整治疗方案, 减少昂贵的无效治疗, 实现个体化用药和治疗。
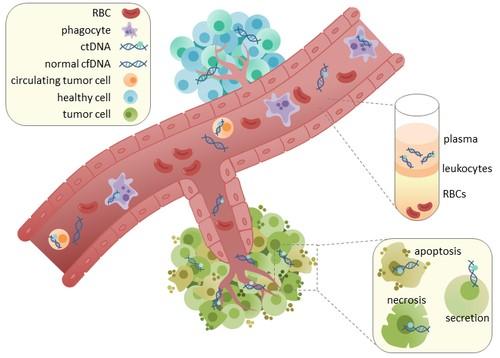
图一.血液中的ctDNA
二.ctDNA的提取
ctDNA在外周血中含量很少, 片段很小, 容易与血浆蛋白结合, 常规的提取效率不高, 同时
为排除凝血过程产生污染, 目前多采用血浆进行ctDNA 抽提。类似于PCR分子诊断的核酸前处理,常用于分离ctDNA 的方法也有两种,二者并没有本质区别,只是二氧化硅微粒的载体不同。
离心柱法:以硅胶滤膜作为固相载体的,基于二氧化硅选择性结合DNA 的独特属性。其原理是带负电的DNA骨架与带正电的硅胶滤膜之间的高亲和力。钠离子起到阳离子桥的作用,它可以吸引核酸磷酸骨架里带负电荷的氧。在高盐条件(pH≦7)下,钠离子可破坏水中的氢与硅中带负电荷的氧之间的氢键。通过大量漂洗去掉所有杂物后,用TE或Tris-Hcl 缓冲液或蒸馏水在低离子强度下(pH≧7)洗脱纯化的DNA。
磁珠法:带有磁荷的颗粒可通过磁场中的永磁将其移除。这些磁颗粒可用表面包被二氧化硅的氧化铁颗粒制成。因表面积大,结合核酸的能力较强,可作为较好的分离载体。如果赋予容器侧壁磁性,样品混合物中结合有核酸的磁珠则聚集到容器壁,直接倾倒容器可将其他杂质去除,避免了反复离心。磁珠表面包被有活性基团可特异性吸附核酸。
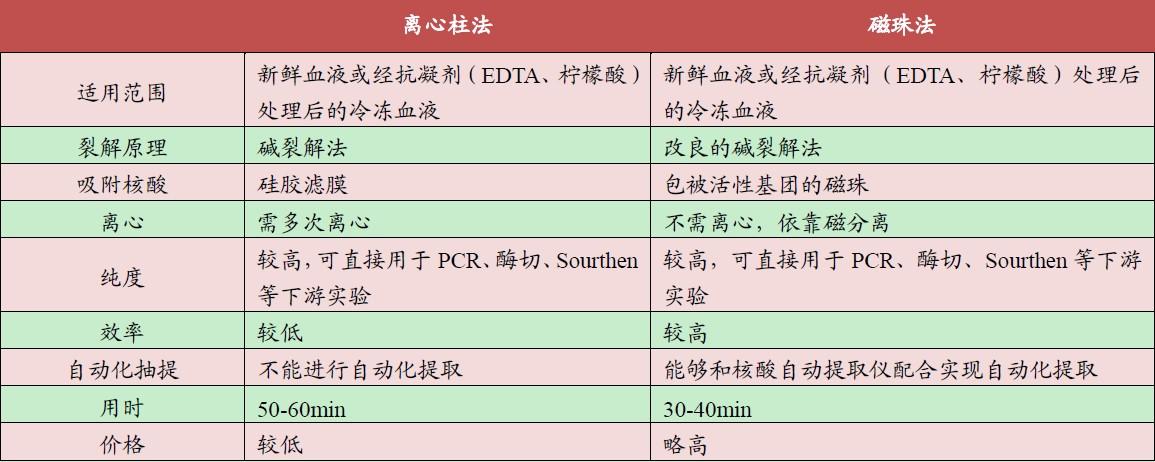
图二.ctDNA提取的两种方法的比较
三.已知突变的ctDNA的检测
由于肿瘤分离出的DNA 的质量和数量变化极大, 因此需要高特异性和高灵敏度的方法检测ctDNA。对于已知突变的ctDNA而言,检测手段有数字PCR (digital PCR, dPCR)、BEAMing (bead, emulsion, amplification and magnetic)、ARMS-PCR(amplification refractory mutation system, ARMS)。
3.1 digital PCR
20 世纪末,Vogelstein 等提出数字PCR( digitalPCR,dPCR) 的概念,通过将一个样本分成几十到几万份,分配到不同的反应单元,每个单元包含一个或多个拷贝的目标分子( DNA 模板) ,在每个反应单元中分别对目标分子进行PCR 扩增,不同于 qPCR对每个循环进行实时荧光测定的方法,dPCR 技术是在扩增结束后对每个反应单元的荧光信号进行采集,最后根据泊松分布原理及阳性微滴的个数与比例得出靶分子的起始拷贝数或浓度。
由于其成本低,灵敏度高,适合检测血液中微量的DNA,digital PCR 成为ctDNA 使用最广泛的检测技术之一。但是digital PCR也有其不可避免的缺陷:低通量,不能检测未知突变。
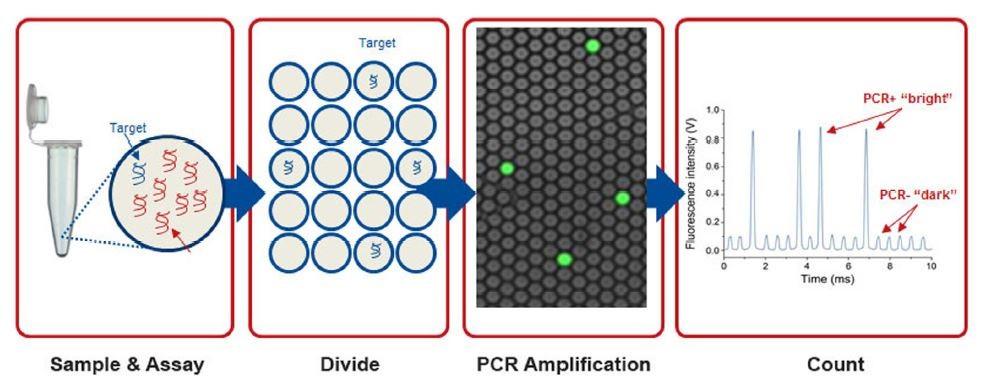
图三. digital PCR原理
3.2 BEAMing
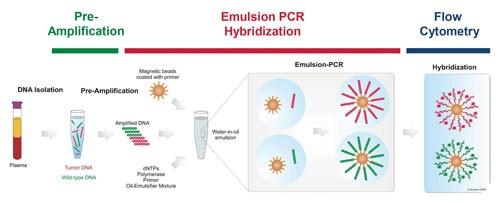
图四. BEAMing原理
BEAMing结合数字PCR与流式技术,通过磁珠克隆DNA。利用特异性PCR 引物扩增目标突变区后,与磁珠(磁珠上固定有特异的PCR 引物)混合进行油包水单分子扩增反应。反乳化作用后,利用不同颜色的荧光探针结合磁珠上的PCR 产物,发出红色或绿色荧光。使用流式细胞仪分析磁珠颜色来确定突变情况。作为DNA检测最灵敏的技术之一, BEAMing扩增法的应用使ctDNA检测的灵敏度大大提高。同样的,BEAMing技术在ctDNA检测方面也存在一定缺陷:低通量,不能检测未知突变。
3.3 ARMS-PCR
突变扩增系统(amplification refractory mutation system, ARMS)是Newton等首先建立用来检测已知突变的方法。其基本原理是,如果引物的3′端碱基与模板碱基不互补,则用一般耐热DNA聚合酶无法延伸。因此根据已知点突变设计3条引物,其3′端碱基分别与突变和正常的模板碱基互补,从而将有某种点突变的模板与正常模板区分开来。此法已用于多种疾病的点突变的检测。
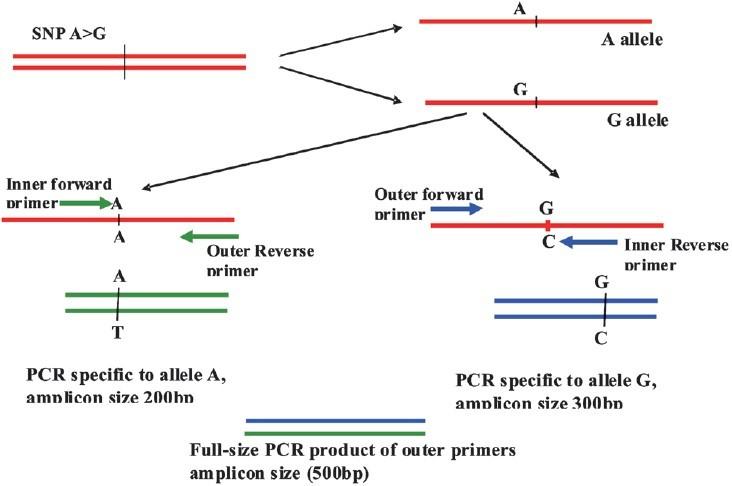
图五.ARMS-PCR原理
四.未知突变的ctDNA的检测
前面提到的几种技术虽然准确率高,成本低,但是通量低,不能检测未知突变,未来发展能力有限。以肺腺癌为例,最大类的两个突变KRAS 和EGFR 分别占了16%和8%,有65%的突变是未知的。目前,检测未知序列的方法主要由两种:标记扩增深度测序(tagged-amplicon deepsequencing, TAm-Seq)技术和癌症个体化深度测序分析方法(cancer personalized profiling by deep sequencing, CAPP-Seq)。
4.1 TAm-Seq
TAm-Seq是一种新一代的高通量测序技术,主要特点是测序通量高、测序时间和成本显著降
低, 一次能对几十万到几百万条DNA 分子进行序列测定。该方法的基本原理是设计特异性引物对目标区域进行循环预扩增,产生大小200bp以下末端重叠覆盖整个区域的扩增子(预扩增),接着通过单重PCR选择性扩增带突变的扩增子区(标签扩增),从而排除非特异性产物,最后在回收的产物上加接头和特异性条形码,进一步通过单端测序得到最终结果。
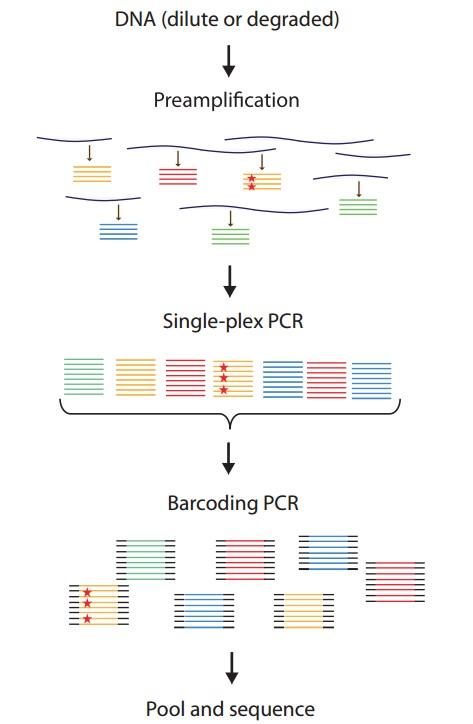
图六. TAm-Seq原理
4.2 CAPP-Seq
CAPP-Seq 法先在在肿瘤基因突变数据库(COSMIC,“筛选库”)来源寻找重复出现的突变相关的外显子,再从肿瘤基因图谱库患者全基因组测序结果筛选突变,设计探针,靶向富集含常见突变基因中的外显子和内含子,有效的把测序区段浓缩到整个基因组大小的0.004%,使得后续超高深度测序得以实现。其对肿瘤的ctDNA 检测灵敏度更高,特异性更强,与全外显子测序等相比经济可行。
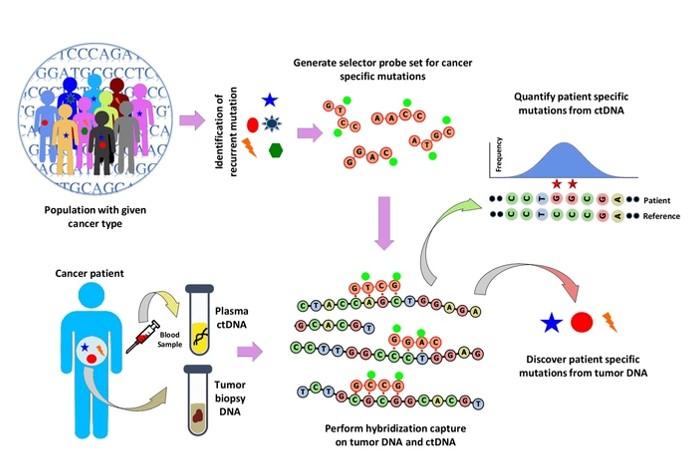
图七. CAPP-Seq原理
五.液体活检ctDNA技术展望
ctDNA 的精确检测同时也给精准医学带来了新的机遇: 1) 精准预防。尤其是对肿瘤高危人群的鉴别, 一旦鉴别出来就可以对其进行干预、跟踪、评估; 2) 精准康复。手术后患者是否具有复发的风险, 如何进行有效的预防和康复干预, 以及治疗效果的评估, 都需要以检测ctDNA 为基准来实现个体化预防和治疗, 提高癌症患者的生存率。

图八.液体活检的检测技术的灵敏度比较
原文地址:https://zhuanlan.zhihu.com/p/30942221 |
|
 /3
/3 